Прикладная биохимия и микробиология, 2020, T. 56, № 2, стр. 123-134
Биочип для одновременной идентификации генов бета-лактамаз и карбапенемаз, обуславливающих устойчивость бактерий к бета-лактамным антибиотикам
М. Ю. Рубцова 1, *, М. М. Уляшова 1, Ю. И. Поболелова 1, Г. В. Преснова 1, А. М. Егоров 1
1 Химический факультет, Московский государственный университет им. М.В. Ломоносова
119991 г. Москва, Россия
* E-mail: mrubtsova@gmail.com
Поступила в редакцию 05.09.2019
После доработки 15.10.2019
Принята к публикации 01.11.2019
Аннотация
Бактериальные бета-лактамазы и карбапенемазы обуславливают устойчивость возбудителей инфекционных заболеваний к классу бета-лактамных антибиотиков, включающих пенициллины, цефалоспорины, карбапенемы и монобактамы. Их широкое распространение среди бактерий-возбудителей инфекционных заболеваний человека и животных представляет глобальную угрозу. Разработан биочип с колориметрической детекцией на основе пероксидазы хрена для одновременной идентификации генов всех клинически значимых бета-лактамаз класса А и карбапенемаз классов А, В и D. На биочипе также определялись 25 замен в нуклеотидной последовательности бета-лактамаз класса А, кодирующие их ключевые замены аминокислотных остатков. Оптимизированы условия аллель-специфической гибридизации меченных биотином ДНК-мишеней с олигонуклеотидными зондами, иммобилизованными на поверхности биочипа. Для получения ДНК-мишеней разработан метод мультиплексной амплификации всех исследуемых генов в одной реакции с одновременным введением биотина. Апробация биочипа проведена с использованием смесей генов бета-лактамаз и карбапенемаз, а также 68 образцов ДНК, выделенных из клинических штаммов грамотрицательных бактерий. Общее время анализа образца ДНК составляло ~4 ч. Продемонстрирована высокая специфичность идентификации комбинаций генов исследуемых ферментов, что может быть использовано для исследования мультирезистентных бактерий.
С момента открытия и до настоящего времени бета-лактамные антибиотики являются наиболее широко используемыми антибактериальными препаратами для лечения инфекционных заболеваний бактериальной природы. Однако широкое распространение бактерий, устойчивых к данному классу антибиотиков, признано глобальной угрозой и существенно ограничивает возможности химиотерапии [1–3]. Основным механизмом устойчивости грамотрицательных бактерий к бета-лактамам является продукция ферментов бета-лактамаз (КФ 3.5.2.6), гидролизующих бета-лактамное кольцо антибиотиков [4]. С каждым годом стремительно растет количество известных бета-лактамаз, и к настоящему времени описано около 2800 различных представителей этого суперсемейства ферментов, которые подразделяют на четыре молекулярных класса A, B, C и D [4, 5]. Ферменты классов A, C и D содержат остаток серина в каталитическом активном центре, а ферменты класса В являются металло-бета-лактамазами. В настоящее время наибольшую опасность представляют бета-лактамазы расширенного спектра (БЛРС), способные расщеплять пенициллины и цефалоспорины I–IV поколений, а также карбапенемазы (КФ 3.5.2.6), гидролизующие наряду с пенициллинами и цефалоспоринами самые новые антибиотики класса бета-лактамов – карбапенемы.
Наиболее распространенные БЛРС относятся к трем типам (TEM, SHV и CTX-M), причем все они представлены сериновыми гидролазами класса А. Внутри каждого типа ферментов присутствует ограниченное число аминокислотных мутаций, называемых ключевыми и влияющих на каталитические свойства, субстратную специфичность и устойчивость ферментов к ингибиторам бета-лактамной природы (клавулановой кислоте, сульбактаму и тазобактаму) [6]. У бета-лактамаз ТЕМ типа к ключевым относят замены аминокислотных остатков в положениях 69, 130, 104, 164, 238, 240, 244, 275 и 276, у бета-лактамаз SHV типа – 35, 69, 130, 179 и 234, 238, 240, у СТХ-М типа – 167 и 240. Большинство этих замен кодируется единичными заменами в нуклеотидной последовательности генов, то есть однонуклеотидным полиморфизмом (ОНП).
В отличие от бета-лактамаз класса А карбапенемазы разнообразны по своему строению и относятся к трем молекулярным классам A, B и D [4, 7]. Среди сериновых карбапенемаз класса А наиболее распространенными являются ферменты КРС типа [8]. Эти ферменты гидролизуют практически все группы бета-лактамных антибиотиков: пенициллины, цефалоспорины всех поколений, карбапенемы и азтреонам, что обусловлено структурой активного центра и расположением каталитического остатка серина таким образом, что он доступен для различных субстратов [9].
Молекулярный класс В объединяет все металло-бета-лактамазы (МБЛ), в активном центре которых присутствуют один или два иона Zn2+ [10]. Данные ферменты характеризуются высокой каталитической активностью и широким спектром субстратной специфичности, включающим практически все бета-лактамные антибиотики за исключением монобактамов. МБЛ нечувствительны к ингибиторам бета-лактамной природы, а ингибируются только хелатирующими агентами, например, ЭДТА. Наиболее распространенными типами карбапенемаз являются VIM и IMP, которые на основании гомологии последовательностей разделяются на три и пять субкластеров соответственно.
В 2007 г. были впервые обнаружены новые МБЛ, относящиеся к NDM типу и гидролизующие все классы бета-лактамных антибиотиков, за исключением монобактамов [11], а уже к 2012 г. эти ферменты были найдены у бактерий, присутствующих на всех континентах земного шара. Бактерии, продуцирующие данные МБЛ, как правило, относятся к мульти- и пан-резистентным, поскольку на плазмидах, несущих ген blaNDM, всегда располагается комплекс интегрона класса 1, включающий комплекс других генов устойчивости к бета-лактамам (гены карбапенемаз KPC, ОХА, VIM типов, БЛРС и бета-лактамаз класса С), макролидам (гены эритромицинэстеразы), аминогликозидам (гены 16S метилазы), хинолонам (гены Qnr), тетрациклинам (гены системы эффлюкса), сульфонамидам (гены дигидроптероатсинтетазы) и рифампицину (гены рифампицин-модифицирующих ферментов) [12, 13].
Среди карбапенемаз ОХА типа класса D выделяют 5 субкластеров: OXA-23, OXA-40, OXA-51, ОХА-48 и OXA-58. Их субстратная специфичность сильно различается, но, в основном, они гидролизуют карбапенемы, пенициллины и цефалоспорины I поколения (цефалотин и цефалоридин) [14].
В настоящее время появление множественной устойчивости к нескольким и даже практически ко всем существующим антибактериальным препаратам привело к появлению мультирезистентных, экстремально резистентных и пан-резистентных штаммов [12, 15]. При этом наблюдается экспоненциальный рост устойчивости как среди возбудителей нозокомиальных (внутрибольничных), так и внебольничных инфекций. Обнаружены бактерии, продуцирующие одновременно несколько бета-лактамаз класса А совместно с карбапенемазами различных классов. Появление множественной устойчивости возбудителей одновременно к нескольким типам антибиотиков существенно ограничивает выбор адекватной лекарственной терапии. Для рационального применения антибактериальных препаратов необходимо понимание молекулярных механизмов индукции резистентности и наличие адекватных высокотехнологичных методов ее идентификации.
В связи с многообразием и опасностью широкого распространения бета-лактамаз и карбапенемаз становится актуальной задача поиска надежных методов их диагностики. В настоящее время для этих целей, в основном, используются микробиологические тесты [16]. Однако они длительны по времени (24–72 ч) и малоэффективны при определении карбапенемаз. При этом трактовка их результатов часто неоднозначна, особенно в случаях, когда наблюдается одновременная продукция нескольких типов бета-лактамаз и карбапенемаз. В последние годы активно развиваются молекулярно-генетические методы идентификации генов бета-лактамаз, включающие амплификацию нуклеиновых кислот методом полимеразной цепной реакции (ПЦР), ПЦР в реальном времени (ПЦР РВ) и методы молекулярной гибридизации [18–20]. К их недостаткам можно отнести необходимость проведения множества параллельных процессов амплификации для увеличения количества определяемых типов генов. Как правило, подразделения гетерогенных типов ферментов на субкластеры не проводятся.
Перспективными для одновременного определения множества генов в одном анализе являются методы анализа ДНК на биочипах [21–23]. Данная технология характеризуется высокой производительностью и позволяет проводить анализ в малом объеме реакционной смеси, что снижает расход реагентов и стоимость анализа в целом. В предыдущей работе [24] был разработан метод гибридизационного анализа ДНК на биочипах с колориметрической детекцией на основе пероксидазы хрена. При этом на поверхности мембранного носителя образуется нерастворимый окрашенный продукт, определение интенсивности окраски которого проводится с использованием оптического сканера высокого разрешения. Данный биочип был использован для определения генов трех типов БЛРС (ТЕМ, SHV и СТХ-М), относящихся к классу А.
Цель работы − разработка метода одновременной идентификации трех типов генов бета-лактамаз класса А (TEM, SHV, CTX-M) и восьми типов генов карбапенепаз (KPC, NDM, VIM, IMP, SPM, SIM, GIM и OXA) путем гибридизационного анализа на биочипах с колориметрической детекцией.
МЕТОДИКА
Набор праймеров для ПЦР и амино-модифицированные олигонуклеотидные зонды синтезированы в “Синтол” (Россия). Образцы ДНК, выделенных из культур контрольных штаммов микроорганизмов-продуцентов бета-лактамаз и карбапенемаз, а также образцы ДНК, выделенных из клинических образцов возбудителей внутрибольничных инфекций, были предоставлены сотрудниками НИИ антимикробной химиотерапии Смоленской государственной медицинской академии (Смоленск, Россия) и НИИ нейрохирургии имени Н.Н. Бурденко (Москва, Россия).
Иммобилизацию олигонуклеотидных зондов на поверхности биочипов из нитроцеллюлозы BioTrace NT (“Pall Corporation”, CША) проводили как описано в работе [24]. Структуры зондов для идентификации генов карбапенемаз и бета-лактамаз класса А и определения ОНП в них были описаны ранее [24, 25]. Амплификацию исследуемых генов с одновременным включением в качестве метки биотина проводили в процессе мультиплексной ПЦР: общий объем смеси составлял 25 мкл и содержал: 5 мкл 5 × KAPA2G буфер М, 2 мM хлорид магния, KAPA2G Fast HotStart ДНК-полимераза (“KAPA Biosystems”, США), по 100 мкМ dATP, dGTP и dCTP, 60 мкМ dTTP, 40 мкМ dUTP-12-биотин (“ДНК-синтез”, Россия), по 0.4 мкМ прямого и обратного праймеров (список и структуры праймеров приведены в табл. 1) и 1 мкл раствора матрицы ДНК. Амплификацию проводили в амплификаторе Mastercycler gradient (“Eppendorf”, Германия) по следующему протоколу: начальная денатурация при 94°С (2 мин), 30 циклов амплификации (10 с – денатурация при 94°С, 30 с – отжиг праймеров при 65°С, 10 с – элонгация при 72°С), 2 мин − завершающий этап элонгации при 72°С.
Таблица 1.
Последовательность и характеристики праймеров для амплификации генов бета-лактамаз и карбапенемаз в мультиплексной ПЦР
| Тип фермента | Последовательность праймера, 5' → 3' | Длина, нукл. | GC, % | Тпл., °С | Длина ампликона, п.н. | |
|---|---|---|---|---|---|---|
| TEM | Пр | ATGAGTATTCAACATTTCCGTGTC | 24 | 37.5 | 63.0 | 851 |
| Об | TTAATCAGTGAGGCACCTATCTC | 23 | 43.5 | 63.0 | ||
| SHV | Пр | TTATATTCGCCTGTGTATTATCTC | 24 | 33.3 | 60.0 | 856 |
| Об | GTTAGCGTTGCCAGTGCTCG | 20 | 60.0 | 67.0 | ||
| CTX-M-1 | Пр | ATGGTTAAAAAATCACTGCGCCAG | 24 | 41.7 | 66.0 | 869 |
| Об | CCGTCGGTGACGATTTTAGCCG | 22 | 59.1 | 68.2 | ||
| CTX-M-2 | Пр | ATGATGACTCAGAGCATTCGCC | 22 | 50.0 | 66.0 | 869 |
| Об | CCGTGGGTTACGATTTTCGCCG | 22 | 59.1 | 68.8 | ||
| CTX-M-8 | Пр | ATGATGAGACATCGCGTTAAGC | 22 | 45.5 | 64.4 | 869 |
| Об | CCGTCGGTGACGATTTTCGCG | 21 | 61.9 | 68.8 | ||
| CTX-M-9 | Пр | GGTGACAAAGAGAGTGCAACGG | 22 | 54.5 | 66.7 | 868 |
| Об | CCCTTCGGCGATGATTCTCGC | 21 | 61.9 | 68.3 | ||
| IMP | Пр | GAAGG(C/T)GTTTATGTTCATAC(T/A)TCG | 24 | 37.5–41.7 | 61.5–63.4 | 586 |
| Об | GTA(A/C)G(T/C)TTCAGAG(T/C)GA(T/C)GC(G/A)TC | 23 | 43.3–52.2 | 63.1–66.7 | ||
| VIM | Пр | GATGGTGTTTGGTCGCATATCGC | 23 | 52.2 | 67.1 | 505 |
| Об | CATTC(A/T)GCCAGATCGGCATCG | 21 | 57.1 | 66.6 | ||
| SPM | Пр | CGTTTTGTTTGTTGCTCGTTGCG | 23 | 47.8 | 67.2 | 643 |
| Об | CACATTGGCATCTCCCAGATAACC | 24 | 50.0 | 66.3 | ||
| SIM | Пр | GTTTGCGGAAGAAGCCCAGCCAG | 23 | 60.9 | 70.8 | 588 |
| Об | GTCTCCGATTTCACTGTGGCTTGGG | 25 | 56.0 | 69.7 | ||
| GIM | Пр | CTTGTAGCGTTGCCAGCTTTAGCTCG | 26 | 53.8 | 70.0 | 614 |
| Об | CTGAACTTCCAACTTTGCCATGCCCC | 26 | 53.8 | 70.7 | ||
| NDM | Пр | CTTCCTATCTCGACATGC | 18 | 50.0 | 58.3 | 503 |
| Об | CGTAGTGCTCAGTGTCG | 17 | 58.8 | 60.7 | ||
| KPC | Пр | TTCTGCTGTCTTGTCTCTCATGG | 23 | 47.8 | 65.5 | 801 |
| Об | CCTCGCTGTGCTTGTCATCC | 20 | 60.0 | 66.5 | ||
| OXA-23 | Пр | GAAACCCCGAGTCAGATTGTTCAAG | 25 | 48.0 | 66.5 | 689 |
| Об | GGCATTTCTGACCGCATTTCC | 21 | 52.4 | 65.7 | ||
| OXA-40 | Пр | GTTTCTCTCAGTGCATGTTCATC | 23 | 43.5 | 63.2 | 713 |
| Об | CATTTCTAAGTTGAGCGAAAAGGGG | 25 | 44.0 | 65.4 | ||
| OXA-51 | Пр | CGAAGCACACACTACGGGTG | 19 | 60.0 | 66.2 | 648 |
| Об | CTCTTTTCGAACAGAGCTAGGTATTC | 26 | 42.3 | 64.2 | ||
| OXA-58 | Пр | CTTGTGCTGAGCATAGTATGAGTC | 24 | 45.8 | 64.1 | 684 |
| Об | CCACTTGCCCATCTGCCTTTTC | 22 | 54.5 | 67.3 | ||
| OXA-48 | Пр | GCGTGTATTAGCCTTATCGGC | 21 | 52.4 | 64.3 | 672 |
| Об | CCACACATTATCATCAAGTTCAACC | 25 | 40.0 | 63.7 | ||
Для фрагментации меченые ПЦР-продукты растворяли до концентрации 30 нг/мкл в реакционном буфере (40 мМ Трис-HCl, pH 8.0, содержащий 10 мМ MgSO4 и 1 мМ CaCl2,) и добавляли ДНКазу I (0.5 мЕд./1 нг ДНК) (“Fermentas”, Германия). Инкубировали в течение 5 мин при комнатной температуре, затем добавляли 3 мМ ЭДТА и дополнительно инкубировали в течение 10 мин при 65°С. Размер полученных фрагментов составлял 50–150 п.н. 750 нг меченой фрагментированной ДНК растворяли в гибридизационном буфере 2хSSPE, рН 7.4, содержащем 0.02 М NaH2PO4, 0.002 М ЭДТА, 0.3 М NaCl и 1.6 пмоль/мл контрольного меченного биотином олигонуклеотида (положительный контроль гибридизации). Биочип помещали в гибридизационную смесь (300 мкл на 1 микрочип) и инкубировали в термомиксере Thermomixer Comfort (“Eppendorf”, Германия) в течение 1 ч при 45°С. После гибридизации мембраны отмывали два раза по 15 мин при комнатной температуре фосфатно-солевым буфером (ФСБ), содержащим 0.05% Твина 20 (ФСБТ). Для детекции результатов гибридизации микрочипы инкубировали в течение 30 мин при 37°С в растворе (1 мкг/мл) конъюгата стрептавидин-пероксидаза хрена (“Имтек”, Россия) в ФСБТ, содержащем 1% бычьего сывороточного альбумина (БСА). Микрочипы отмывали при перемешивании ФСБТ (10 мин) и ФСБ (10 мин) при комнатной температуре и помещали на 10 мин в субстратный раствор, содержащий 3,3',5,5-тетраметилбензидин (ТМБ), Н2О2 (“НВО Иммунотех”, Россия) и декстран сульфата натрия (“Sigma-Aldrich”, США) до конечной концентрации 0.5% (по массе). После высушивания биочипы сканировали на сканере Perfection V750 Pro (“Epson”, Германия) при разрешении 4800 dpi. Полученные изображения в формате tiff обрабатывали количественно с использованием программы Scan Array Express (“PerkinElmer”, version 3.0, Германия). Аналитическим сигналом являлась интенсивность окрашивания зоны биочипа. Сигнал принимался значимым, если средняя интенсивность превышала интенсивность фонового окрашивания более, чем на 5 величин стандартного отклонения. Принцип определения нуклеотида в позиции ОНП и кодируемой аминокислотной замены описан в работе [24].
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
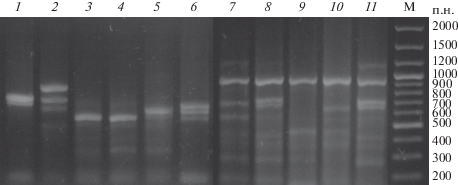
Совместная амплификация генов бета-лактамаз класса А и карбапенемаз методом мультиплексной ПЦР. Для получения ДНК-мишеней всех исследуемых генов был разработан метод одновременной амплификации генов трех типов бета-лактамаз класса А (TEM, SHV и CTX-M) и восьми типов карбапенемаз классов A, B, и D (KPC, NDM, VIM, IMP, SPM, SIM, GIM и OXA) в процессе одной мультиплексной ПЦР. Для реакции амплификации было использовано 18 пар праймеров (табл. 1). Поскольку гены, кодирующие бета-лактамазы СТХ-М типа и карбапенемазы ОХА типа, достаточно разнородны, они были разделены на основании гомологии нуклеотидных последовательностей на 4 и 5 кластеров соответственно. Для каждого генетического кластера была подобрана пара специфических праймеров. Результаты электрофореза продуктов амплификации исследуемых генов в 1%-ном агарозном геле показаны на рис. 1. Все целевые гены бета-лактамаз и карбапенемаз удалось амплифицировать с достаточным для дальнейшей гибридизации выходом (не менее 40 нг ДНК/мкл). При амплификации практически всех генов наблюдали образование неспецифических продуктов амплификации. Оптимизация температуры стадии отжига праймеров и времени проведения каждой стадии реакции амплификации позволила сократить общее время реакции до 40 мин.
Рис. 1.
Электрофореграмма продуктов амплификации генов бета-лактамаз класса А и карбапенемаз методом мультиплексной ПЦР: 1 – OXA-40, 2 – OXA-48, 3 – VIM-1, 4 – VIM-2, 5 – IMP-1, 6 – IMP-2, 7 – TEM-1, 8 – SHV‑5, 9 – SHV-18, 10 – CTX-M-15, 11 – CTX-M-3, M – ДНК-маркеры.
Влияние условий гибридизации на специфичность гибридизации идентификационных зондов. Для одновременной идентификации разнородных генов бета-лактамаз и карбапенемаз в одном анализе, включая одновременное определение единичных замен в генах бета-лактамаз класса А, была проведена оптимизация условий гибридизационного анализа ДНК-мишени с олигонуклеотидными зондами, иммобилизованными на поверхности биочипа. Целью оптимизации было повышение специфичности определения генов каждого типа, поскольку ДНК-мишени содержали побочные продукты амплификации. Было изучено влияние температуры гибридизации и солевого состава гибридизационного буфера, которые относятся к параметрам, наиболее существенно влияющим на специфичность определения ДНК в гибридизационном анализе. На биочипах с иммобилизованными олигонуклеотидными зондами проводили гибридизацию 300 нг биотинилированной ДНК, амплифицированной из контрольных штаммов-продуцентов бета-лактамаз, в течение 2 ч при интенсивном перемешивании. Далее определяли интенсивность сигналов гибридизации ДНК-мишени каждого типа бета-лактамаз со специфическими для них зондами, а уровень неспецифической гибридизации определяли по интенсивности сигналов гибридизации данной ДНК-мишени с зондами для других типов и субклассов бета-лактамаз.
Выбор оптимальной температуры гибридизации. При выборе температуры гибридизации основным критерием являлось то, что она должна быть ниже температуры плавления (Тm) образующихся гетеродуплексов между комплементарным зондом и ДНК-мишенью. Диапазон исследованных температур гибридизации составил от 40 до 50°С и не превышал Tm отдельных олигонуклеотидных зондов, а также ограничивался температурой, при которой наблюдали высокий уровень неспецифической гибридизации. Выборочные данные по гибридизации генов бета-лактамаз класса А (TEM, SHV и CTX-M типов) и карбапенемаз (VIM и OXA типов) с иммобилизованными на биочипах специфическими идентификационными зондами, полученные при разных температурах гибридизации, представлены на рис. 2.
Рис. 2.
Влияние температуры гибридизации (°С) на интенсивность окрашивания зоны биочипа (усл. ед.) при гибридизационном анализе генов бета-лактамаз (1 – TEM-1, 2 – SHV-1, 3 – CTX-M-15), карбапенемаз (4 – VIM-1, 5 – OXA-51) и неспецифической гибридизации (6).
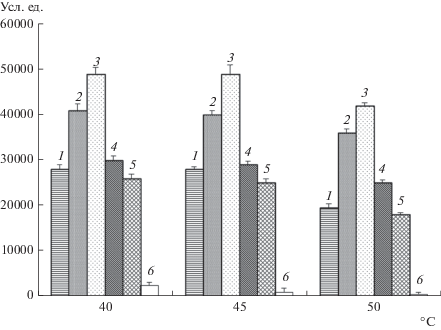
Анализ полученных результатов показал, что абсолютные интенсивности сигналов были выше при более низких температурах и уменьшались с увеличением температуры. При проведении гибридизации при 40°С абсолютная интенсивность сигналов оказалась высокой (для большинства зондов она превышала 25000 усл. ед.), однако при этом ряд наборов зондов, определяющих единичные замены в генах ТЕМ бета-лактамаз, показывали низкую дискриминирующую способность. Для генов подгрупп СТХ-М бета-лактамаз и IMP карбапенемаз при этой температуре наблюдалась сильная перекрестная гибридизация с наборами зондов других подгрупп (на диаграмме приведено значение для неспецифической гибридизации с наибольшей интенсивностью сигнала). При проведении гибридизации при 50°С интенсивность гибридизационных сигналов в целом была низкой и определение единичных замен, соответствующих мутациям аминокислотных остатков в позициях 69, 104 и 130 у ТЕМ бета-лактамаз и в позициях 69, 130 и 238/240 у SHV бета-лактамаз, оказалось невозможным. В качестве оптимальной температуры для проведения гибридизации было выбрано 45°С.
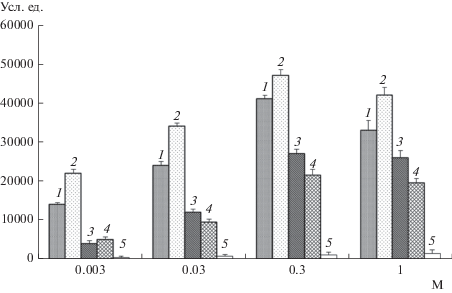
Выбор концентрации NaCl в гибридизационном буфере. Результат гибридизации олигонуклеотидных зондов с ДНК-мишенью зависел также от концентрации соли в гибридизационном буфере. Влияние концентрации NaCl на результат гибридизации изучали на двух вариантах генов карбапенемаз (IMP-1 и VIM-1) и двух различных генах бета-лактамаз класса А (SHV-1 и CTX-M-15). Гибридизацию проводили при 45°С. На рис. 3 показаны выборочно результаты гибридизации ДНК-мишени со специфическими зондами, полученные при использовании гибридизационного буфера с различным содержанием NaCl. Увеличение концентрации соли приводило до определенной ее концентрации к увеличению наблюдаемых интенсивностей гибридизационных сигналов вследствие уменьшения взаимного отталкивания заряженных фосфатных групп двух нуклеотидных цепей. Однако при использовании буфера с высокой концентрацией соли (1 М) специфичность реакции гибридизации падала. Для идентификации единичных замен в генах при проведении гибридизации при низкой концентрации соли в гибридизационном буфере (30 мМ) интенсивность гибридизационных сигналов была очень низкой, при этом не наблюдалось гибридизационных сигналов для более чем 50% наборов зондов. Это свидетельствовало о том, что в гибридизационной смеси оказывалось недостаточно содержания ионов натрия, чтобы полностью экранировать отрицательные заряды сахаро-фосфатных остовов олигонуклеотидных зондов и ДНК-мишеней, и между ними при гибридизации действовали силы электростатического отталкивания. Увеличение содержания ионов натрия в гибридизационной смеси приводило к увеличению интенсивности гибридизационных сигналов, которые достигали максимальных значений при 0.3–1.0 М NaCl, и при этом выявлялось максимальное количество точечных замен в генах. Однако следует отметить, что интенсивность сигналов неспецифической гибридизации также возрастала. При этом для большинства наборов зондов значения неспецифической гибридизации увеличивались в большей степени, чем увеличение специфических сигналов, что в результате снижало специфичность выявления нуклеотидов. Таким образом, концентрация NaCl, соответствующая 0.3 М, была оптимальной для наиболее удачного соотношения эффективности и специфичности гибридизации.
Рис. 3.
Влияние концентрации NaCl (М) на интенсивность окрашивания зоны биочипа в гибридизационном анализе генов бета-лактамаз (1 – SHV-1, 2 – CTX-M-15), карбапенемаз (3 – VIM-1, 4 – IMP-1) и неспецифической гибридизации (5).
Биочип для одновременной идентификации генов бета-лактамаз класса А и карбапенемаз. Для одновременной идентификации генов бета-лактамаз класса А (трех типов: ТЕМ, SHV и CTX-M), карбапенемаз (восьми типов: KPC, NDM, VIM, IMP, SPM, SIM, GIM и OXA) был проведен дизайн биочипа, который заключался в выборе количества специфических зондов для идентификации выбранных типов генов с учетом определения субкластеров и необходимого количества контрольных зондов, а также размещения зондов в необходимом количестве повторов.
Подобраны структуры зондов для идентификации отдельных субкластеров генов бета-лактамаз СТХ-М типа (СТХ-М-1, СТХ-М-2, CTX-M-8 и СТХ-М-9), карбапенемаз VIM типа (VIM-1, VIM-2 и VIM-7), IMP типа (IMP-1, IMP-2, IMP-5, IMP-11 и IMP-14), ОХА типа (ОХА-23, ОХА-40, ОХА-48, ОХА-51 и ОХА-58). Биочип включал олигонуклеотидные зонды для определения 15 позиций ОНП, кодирующих ключевые мутации в бета-лактамазах ТЕМ и SHV типов, обуславливающих изменение профиля субстратной специфичности и устойчивости к ингибиторам (8 позиций в бета-лактамазах ТЕМ типа, 7 − в бета-лактамазах SHV типа). Также биочип включал зонды для определения 10 позиций ОНП для идентификации бета-лактамаз внутри отдельных субкластеров (2 – в бета-лактамазах субкластера СТХ-М-1, 5 – в бета-лактамазах субкластера СТХ-М-2 и 3 − в бета-лактамазах субкластера СТХ-М-9), поскольку они наиболее распространены в РФ и других странах [26]. Схема расположения олигонуклеотидных зондов на поверхности биочипа представлена на рис. 4.
Рис. 4.
Расположение олигонуклеотидных зондов на биочипе для идентификации генов 3 типов бета-лактамаз класса А, 8 – карбапенемаз и 24 позиций однонуклеотидного полиморфизма в генах
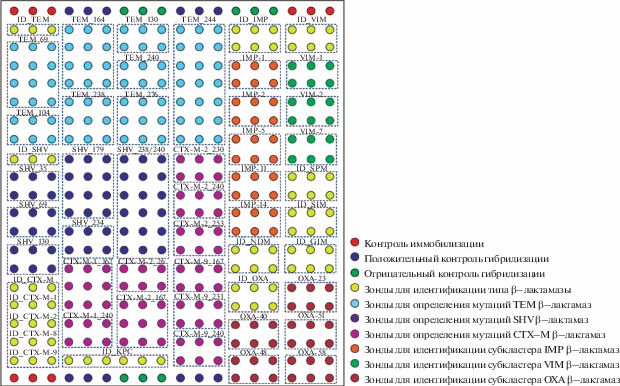
Помимо специфических зондов каждый биочип содержал 3 типа контрольных олигонуклеотидных зонда. Каждый зонд наносился в 3 повторах.
Биочип имел следующие размеры: 14 × 12 мм, зоны биочипа расположены в виде матрицы 21 × 18, общее количество зон 378, диаметр одной зоны с иммобилизованным олигонуклеотидным зондом составлял ~ 350 мкм, расстояние между центрами соседних зон ~ 500 мкм.
Тестирование смесей генов бета-лактамаз класса А и карбапенемаз на биочипе. Возможность одновременной детекции генов различных бета-лактамаз и карбапенемаз была изучена на искусственно приготовленных смесях ДНК различных бета-лактамаз. Предварительно было проведено сравнение результатов гибридизации продуктов амплификации изучаемых генов (по одному варианту для каждого типа генов) со специфическими праймерами и в процессе мультиплексной ПЦР. Для этого ДНК-мишени исследуемых ферментов, меченные биотином, были получены методом специфической ПЦР с одной парой специфических праймеров и методом мультиплексной ПЦР со смесью 18 пар праймеров. Несмотря на то, что мультиплексная ПЦР сопровождалась синтезом побочных продуктов реакции (рис. 1), было показано, что специфичность идентификации генов бета-лактамаз и карбапенемаз в гибридизационном анализе на биочипах оказалась одинаково высокой для всех исследованных ДНК-мишеней, полученных в ПЦР с одной парой специфических праймеров и в процессе мультиплексной ПЦР.
Далее ДНК-мишени всех исследуемых генов получали разработанным методом мультиплексной ПЦР. Были протестированы различные варианты генов бета-лактамаз (ТЕМ-1 и SHV-1), БЛРС класса А (ТЕМ-3, ТЕМ-6, SHV-2, SHV-5, СТХ-М-3, СТХ-М-9 и СТХ-М-15) и карбапенемаз VIM-1, VIM-2, IMP-1, IMP-2, KPC-3, VIM-1, VIM-2, IMP-1, IMP-2, SPM-1, NDM-1, ОХА-48, ОХА-23, ОХА-40, ОХА-51 и OXA-58 в различных сочетаниях. Все ДНК-мишени гибридизовались как индивидуально, так и в комбинациях только со специфическими зондами, при этом уровень неспецифической гибридизации не превышал заданный интервал (5-кратное превышение стандартного отклонения усредненного значения аналитического сигнала). Во всех экспериментах наблюдали хороший уровень воспроизводимости интесивности окрашивания всех трех повторов каждого зонда. Коэффициенты вариации интенсивностей окрашивания зон всех специфических зондов с комплементарной ДНК-мишенью, каждый из которых был нанесен в трех повторах, варьировали от 5 до 10%. Для зондов, уровень гибридизации ДНК-мишени с которыми был незначительным и соответствовал неспецифической гибридизации, наблюдали более высокие значения коэффициентов вариации, но они не превышали 20%.
Результаты гибридизационного анализа представлены выборочно для двух смесей ДНК. Смесь № 1 была получена из образцов ДНК карбапенемазы ОХА-48 и бета-лактамазы SHV-5, смесь № 2 состояла из ДНК металло-бета-лактамазы NDM-1 и бета-лактамазы СТХ-М-9. Продукция таких сочетаний бета-лактамаз класса А и карбапенемаз описана у мультирезистентных клинических штаммов возбудителей внутрибольничных инфекций [27]. На рис. 5 представлены изображения биочипов после проведения гибридизационного анализа двух смесей генов бета-лактамаз класса А и карбапенемаз, на рис. 6 – результаты количественной обработки интенсивностей окрашивания зон биочипа.
Рис. 5.
Изображение биочипов после проведения гибридизационного анализа смесей ДНК, выделенных из контрольных штаммов грамотрицательных бактерий: а − смеси генов бета-лактамазы SHV-5 (окрашивание зон биочипа получено для зондов 1 (тип SHV) и зондов, определяющих аминокислотные мутации в SHV бета-лактамазах: 2 – Leu35, 3 – Met69, 4 – Ser130, 5 – Asp179, 6 – Lys234, 7 – замены Gly238Ser/Glu240Lys) и карбапенемазы OXA-48 (окрашивание зон биочипа получено для зондов 8 (тип OXA) и 9 (субкластер OXA-48)); б − смеси генов бета-лактамазы СТХ-М-9, окрашивание зон биочипа получено для зондов 10 (тип CTX-M), 11 (субкластер CTX-M-9) и зондов, определяющих аминокислотные мутации в субкластере CTX-M-9: 12 – Pro167, 13 – Ala231, 14 – Asp240) и карбапенемазы NDM-1, окрашивание зон биочипа получено для зондов 15 (тип NDM).
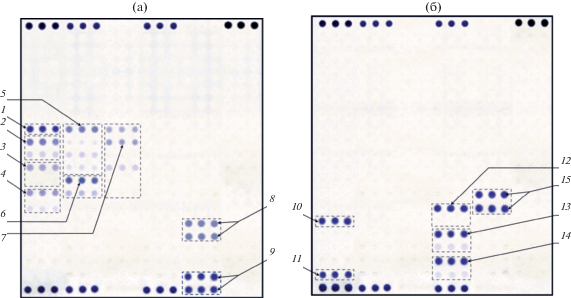
Рис. 6.
Результаты гибридизационного анализа смесей ДНК, выделенных из контрольных штаммов грамотрицательных бактерий, на которых идентифицированы: а, б – смесь генов бета-лактамазы SHV-5 и карбапенемазы OXA-48: а − значимые сигналы гибридизации получены для зондов “тип SHV”, “тип OXA”, “субкластер OXA-48”; б − значимые сигналы гибридизации получены для зондов, определяющих следующие аминокислоты в ключевых позициях SHV бета-лактамаз: Leu35, Met69, Ser130, Asp179, Lys234, замены Gly238Ser/Glu240Lys (соответствует SHV БЛРС); в, г – смесь генов бета-лактамазы СТХ-М-9 и карбапенемазы NDM-1: в − значимые сигналы гибридизации получены для зондов “тип NDM”, “тип CTX-M”, “субкластер CTX-M-9”, г − значимые сигналы гибридизации получены для зондов, определяющих следующие аминокислоты в ключевых позициях CTX-M бета-лактамаз: Pro167, Ala231, Asp240.
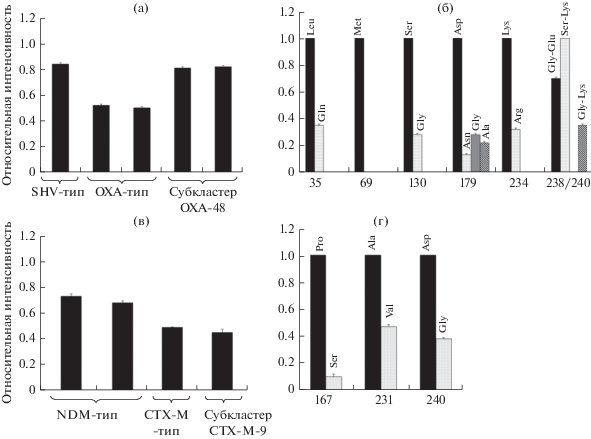
Поскольку все исследованные ДНК бета-лактамаз и карбапенемаз правильно и достоверно идентифицировались на биочипе, можно было заключить, что разработанный биочип позволял проводить высокоспецифическую идентификацию генов бета-лактамаз класса A и карбапенемаз с разделением отдельных типов ферментов на субкластеры даже при одновременной продукции нескольких ферментов в одном образце.
Апробация биочипа для идентификации генов карбапенемаз и бета-лактамаз молекулярного класса А. Разработанный олигонуклеотидный микрочип для определения генов карбапенемаз и бета-лактамаз молекулярного класса А был апробирован на 68 образцах ДНК, выделенных из клинических штаммов Enterobacteriaceae, Pseudomonas aeruginosa, Acinetobacter baumannii, полученных в различных лечебных учреждениях России. Все штаммы были предварительно охарактеризованы фенотипически с использованием диско-диффузионного метода. Так, 19 образцов были чувствительны к действию цефалоспоринов 3–4 поколений и карбапенемов (отсутствие продукции БЛРС и карбапенемаз), 27 образцов оказались устойчивы к действию цефалоспоринов 3-4 поколений и характеризовались фенотипом БЛРС (+), а 22 образца – устойчивы к карбапенемам (карбапенемазы +).
Результаты гибридизационного анализа этих образцов на биочипах представлены в табл. 2. При тестировании 19 образцов ДНК, выделенных из штаммов микроорганизмов, чувствительных к действию карбапенемов и цефалоспоринов 3–4 поколений, гены бета-лактамаз и карбапенемаз исследуемых типов не были обнаружены в 14, а среди 5 представителей Enterobacteriaceae spp. были идентифицированы гены пенициллиназ TEM-1 и SHV-1, которые обуславливали устойчивость к пенициллинам и цефалоспоринам I поколения. Таким образом, не было выявлено ложноположительных и ложноотрицательных результатов наличия генов бета-лактамаз и карбапенемаз при тестировании на биочипах ДНК клинических образцов.
Таблица 2.
Результаты гибридизационного анализа на биочипах образцов ДНК, выделенных из клинических штаммов грамотрицательных бактерий, на наличие генов бета-лактамаз и карбапенемаз
| Типы/Субтипы найденных бета-лактамаз и карбапенемаз | Число штаммов |
|---|---|
| Штаммы, чувствительные к цефалоспоринам 3–4 поколений и карбапенемам (n = 19) | |
| Не обнаружено генов бета-лактамаз | 14 |
| ТЕМ-1 | 2 |
| SHV-1 | 3 |
| Штаммы, устойчивые к цефалоспоринам 3–4 поколений (n = 27) | |
| Продуцирующие одну БЛРС, в том числе на фоне пенициллиназ ТЕМ-1 и/или SHV-1 (n = 19) | |
| СТХ-M-3 | 1 |
| СТХ-M-15 | 1 |
| СТХ-M-14 | 1 |
| TEM-1 + SHV-5 | 1 |
| TEM-1 + CTX-M-3 | 2 |
| TEM-1 + CTX-M-15 | 6 |
| TEM-1 + CTX-M-14 | 1 |
| SHV-1 + CTX-M-15 | 3 |
| TEM-1 + SHV-1 + CTX-M-15 | 3 |
| Продуцирующие две БЛРС, в том числе на фоне пенициллиназ ТЕМ-1 и/или SHV-1 (n = 8) | |
| SHV-5 + CTX-M-9 | 1 |
| TEM-1 + SHV-5 + CTX-M-3 | 1 |
| TEM-1 + SHV-5 + CTX-M-15 | 1 |
| TEM-1 + SHV-5 + CTX-M-14 | 1 |
| SHV-1 + SHV-5 + CTX-M-14 | 1 |
| TEM-1 + SHV-1 + SHV-5 + CTX-M-15 | 2 |
| TEM-1 + SHV-1 + SHV-5 + CTX-M-14 | 1 |
| Штаммы, устойчивые к цефалоспоринам 3–4 поколений и карбапенемам (n = 22) | |
| Продуцирующие одну карбапенемазу (n = 12) | |
| VIM-2 | 11 |
| NDM | 1 |
| Продуцирующие две карбапенемазы (n = 10) | |
| OXA-23 + OXA-51 | 1 |
| OXA-40 + OXA-51 | 5 |
| OXA-58 + OXA-51 | 4 |
При исследовании 27 образцов ДНК бактерий, устойчивых к цефалоспоринам 3–4 поколений, в 19 были идентифицированы гены одной БЛРС (СТХ-М типа (n = 18) или SHV типа(n = 1)), в 8 − гены одновременно двух БЛРС (сочетания генов ферментов СТХ-М и SHV типов). Ложноотрицательных результатов при тестировании клинических образцов не было выявлено.
При анализе 22 образцов ДНК бактерий, устойчивых к действию карбапенемов, в 12 образцах были идентифицированы гены одной карбапенемазы (типа VIM (n = 11) или NDM (n = 1)), а в 10 – гены одновременно двух карбапенемаз ОХА типа.
Идентификация генов бета-лактамаз на микрочипах показала 100%-ное совпадение с данными микробиологического фенотипирования, секвенирования и метода ПЦР РВ. Разработанный биочип позволял правильно определить тип фермента, принадлежность к определенному субкластеру и наличие ключевых мутаций в ферментах класса А, определяющих профиль субстратной специфичности и устойчивости к ингибиторам.
Таким образом, оптимизация гибридизационного анализа на биочипах и пробоподготовки ДНК-мишени, проведенная методом мультиплексной ПЦР, позволила создать биочип, с помощью которого в одном анализе можно специфично идентифицировать гены всех клинически значимых на сегодняшний день бета-лактамаз. К его преимуществам можно отнести впервые выполненную на одном биочипе идентификацию генов нескольких типов ферментов на уровне субкластеров (четырех для бета-лактамаз СТХ-М типа, пяти для карбапенемаз ОХА типа, трех для карбапенемаз VIM типа, пяти для карбапенемаз IMP типа). Одновременно биочип позволял высокоспецифично определять ключевые замены в генах бета-лактамаз класса А, что важно для идентификации БЛРС. Общее время анализа образца ДНК составило около 4 ч. К одному из основных преимуществ разработанного метода относится высокая специфичность идентификации нескольких генов в одном образце, что может быть использовано при исследовании мультирезистентных штаммов бактерий.
Работа выполнена при финансовой поддержке Российского научного фонда (проект № 15-14-00014-П).
Список литературы
Cassini A., Högberg L.D., Plachouras D., Quattrocchi A., Hoxha A., Simonsen G.S. // Lancet Infect Dis. 2019. V. 19. № 1. P. 56–66.
Roca I., Akova M., Baquero F., Carlet J., Cavaleri M., Coenen S., Cohen J., Findlay D., Gyssens I., Heuer O.E., Kahlmeter G., Kruse H., Laxminarayan R., Liébana E., López-Cerero L., MacGowan A., Martins M., Rodríguez-Baño J., Rolain J.M., Segovia C., Sigauque B., Tacconelli E., Wellington E., Vila J. // New Infections. 2015. V. 16. № C. P. 22–29.
Holmes A.H., Moore L.S.P., Sundsfjord A., Steinbakk M., Regmi S., Karkey A., Guerin P.J., Piddock L.J. // Lancet. 2016. V. 387. № 10014. P. 176–187.
Bush K. // Antimicrob. Agents Chemother. 2018. V. 62. № 10. P. 1–20.
Bonomo RA. // Cold Spring Harb. Perspect. Med. 2017. V. 7. № 1. P. 1–16.
Bush K., Bradford P.A. // Cold Spring Harb. Perspect. Med. 2016. V. 6. № 8. P. 1–22.
van Duin D, Doi Y. // Virulence. 2017. V. 8. № 4. P. 460–469.
Naas T., Dortet L., Iorga B.I. // Curr. Drug. Targets. 2016. V. 17. № 9. P. 1006–1028.
Jean S.-S., Lee W.-S., Lam C., Hsu C.-W., Chen R.-J., Hsueh P.-R. // Future Microbiol. 2015. V. 10. № 3. P. 407–425.
Somboro A.M., Osei S.J., Amoako D.G, Essack S.Y., Bester L.A. // Appl. Environ. Microbiol. 2018. V. 84. V. 7. № 18. pii: e00698-18.
Wu W., Feng Y., Tang G., Qiao F., McNally A., Zong Z. // Clin. Microbiol. Rev. 2019. V. 32. № 2. pii: e00115-18.
Eichenberger E.M., Thaden J.T. //Antibiotics (Basel). 2019. V. 8. № 2. P. pii: E37.
Groundwater P.W., Xu S., Lai F., Varadi L., Tan J. Perry J.D., Hibbs D.E. // Future Med. Chem. 2016. V. 8. № 9. P. 993−1012.
Evans B.A., Amyes S.G. // Clin. Microbiol. Rev. 2014. V. 27. № 2. P. 241−263.
Geisinger E., Isberg R.R. // J. Infect. Dis. 2017. V. 215. № S1. P. S9–S17.
Hammoudi D., Moubareck C.A., Moubareck D. // J. Microbiol. Methods. 2014. V. 107. P. 106–118.
Fleece M.E., Pholwat S., Mathers A.J., Houpt E.R. // Expert Rev. Mol. Diagn. 2018. V. 18. № 3. P. 207–217.
Sekyere J.O., Govinden U., Essack S. Y. // J. Appl. Microbiol. 2015. V. 119. № 5. P. 1219−1233.
Murugan N., Malathi J., Therese K.L., Narahari H., Madhavan R. // Kaohsiung J. Med. Sci. 2018. V. 34. № 2. P. 79e88.
Lowman W., Marais M., Ahmed K., Marcus L. // J. Hosp. Infect. 2014. V. 88. № 2. P. 66−71.
Ledeboer N.A., Hodinka R.L. // J. Clin. Microbiol. 2011. V. 9. Suppl. P. S20−S24.
Moore N.M., Canton R., Carretto E., Peterson L.R., Sautter R.L., Traczewski M.M., Carba-R Study Team // J. Clin. Microbiol. 2017. V. 55. № 7. P. 2268−2275.
Ledeboer N.A., Lopansri B.K., Dhiman N., Cavagnolo R., Carroll K.C., Granato P., Thomson R.Jr., Butler-Wu S.M., Berger H., Samuel L., Pancholi P., Swyers L., Hansen G.T., Tran N.K., Polage C.R., Thomson K.S., Hanson N.D., Winegar R., Buchan B.W. // J. Clin. Microbiol. 2015. V. 53. № 8. P. 2460−2472.
Rubtsova M.Yu., Ulyashova M.M., Edelstein M.V., Egorov A.M. // Biosens. Bioelectron. 2010. V. 26. № 4. P. 1252−1260.
Ulyashova M.M., Khalilova Yu.I., Rubtsova M.Yu., Edelstein M.V., Alexandrova I.M., Egorov A.M. //Acta Naturae. 2010. V. 2. № 3. P. 101−109.
Сухорукова М.В., Эйдельштейн М.В., Склеенова Е.Ю. Иванчик Н.В., Тимохова А.В., Дехнич А.В., Коз-лов Р.С. // Клиническая микробиология и антимикробная химиотерапия. 2014. Т. 16. № 4. С. 254−265.
Bush K. // J. Glob. Antimicrob. Resist. 2013. V. 1. № 1. P. 7−16.
Дополнительные материалы отсутствуют.
Инструменты
Прикладная биохимия и микробиология


