Прикладная биохимия и микробиология, 2020, T. 56, № 2, стр. 107-122
Системы секреции бактериальных фитопатогенов и мутуалистов (обзор)
Л. А. Ломоватская 1, А. С. Романенко 1, *
1 Сибирский институт физиологии и биохимии растений Сибирского отделения РАН
664033 Иркутск, Россия
* E-mail: rom@sifibr.irk.ru
Поступила в редакцию 01.04.2019
После доработки 09.09.2019
Принята к публикации 01.10.2019
Аннотация
В настоящее время у бактерий выделяют девять систем секреции белков через клеточные мембраны, существенно различающиеся по сложности функционирования. Большинство данных о таких системах получено на патогенах животных, тогда как фитопатогенам, и, тем более, мутуалистическим видам бактерий посвящены лишь отдельные работы, не дающие общего представления об их роли в реализации вирулентности, а также их сходстве и различиях с аналогичными системами у бактериальных патогенов животных. В обзоре представлено современное состояние исследований о структурных и функциональных особенностях систем секреции фитопатогенов и мутуалистов, а также их значение в подавлении фитоиммунитета.
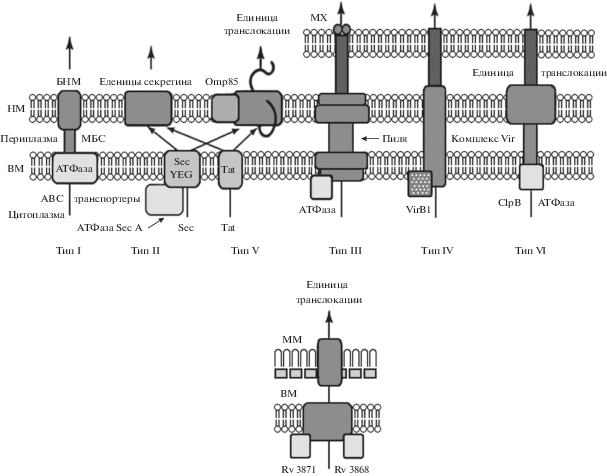
Все прокариоты независимо от их структурных особенностей и специализации секретируют белки через клеточные мембраны [1, 2]. Для бактерий этот процесс важен, так как значительное количество их белков, в том числе большинство факторов вирулентности локализованы вне цитоплазмы [3]. В настоящее время у бактерий выделяют девять систем секреции, существенно различающиеся по структуре и сложности функционирования [2, 4]. На рис. 1 изображены основные компоненты семи сигнальных систем, поскольку только они пока обнаружены у фитопатогенов и мутуалистов.
Рис. 1.
Упрощенные схемы 7 систем секреции бактерий: МХ – мембрана клетки хозяина, НМ – наружная мембрана, ВМ – внутренняя мембрана, ММ – микромембрана, БНМ – белок наружной мембраны, МБС – мембранный белок слияния, АТФаза SecA – АТФаза SecA-секреторного пути, АТФаза ClpB – АТФаза T6SS, Rv3871 и Rv3868 – шапероны и Omp85 – белок внешней мембраны бактерий.
Основные исследования бактериальных систем секреции выполнены на патогенах животных [3], и это вполне объяснимо, поскольку познание механизмов патогенности позволяет находить новые средства борьбы с ними. В тоже время соответствующие системы транспорта факторов вирулентности фитопатогенов и мутуалистических бактерий изучены значительно хуже, и им посвящены лишь отдельные работы [5, 6], не дающие представления о роли систем секреции в реализации вирулентности патогенов растений, их сходстве и различиях с аналогичными системами животных. Хотя по некоторым данным, несмотря на принципиальные различия в специализации, между системами секреции патогенов животных и растений имеются черты сходства. Так, показано, что белки lipB, lipC и lipD, представляющие компоненты системы секреции липазы патогена человека Serratia marcescens и компоненты PrtD, PrtE и PrtF транспортера металлопротеазы фитопатогенной бактерии Ewinia chrysanthemi характеризуются высокой степенью гомологии (45−55%) [7].
Бактериальные системы секреции различаются как по степени сложности, так и по видам секретируемых белков (субстратов). Еще в начале 2000 гг. признавалось существование только пяти типов секреторных систем [8–10], а в настоящее время получили экспериментальное подтверждение системы секреции VI−IX типов [11, 12].
Три из девяти систем относятся к двухэтапным (или двухшаговым). Первый этап такой секреции включает перенос предназначенного для секреции белка из цитоплазмы бактериальной клетки в периплазматическое пространство. Для его осуществления у бактерий существует два механизма. Один из них назван двуаргининовым путем (ТАТ, от pathway two-arginine translocation), второй – Sec-путь (от secretory – секреция) [13–15].
Оба эти пути перемещения белков через мембраны функционируют в клетках микроорганизмов всех уровней клеточной организации, поскольку соответствующие им белки обнаружены у архей, эубактерий и эукариот. Sec-путь предназначен для транспорта белков, не прошедших посттрансляционный фолдинг, в то время как ТАТ-путь обеспечивает проведение через мембрану только белков, полностью принявших конформацию, характерную для их активной формы [16]. У архей и грамположительных бактерий Sec- и TAT-пути обеспечивают выведение некоторых белков непосредственно в окружающую среду. У грамотрицательных бактерий они функционируют как начальные этапы двухступенчатой системы секреции, к которым относятся системы 2 (T2SS), 5 (T5SS) и 9 (T9SS) типов [8–10].
Sec-путь обеспечивается цитоплазматическим белком SecА и интегрированными в цитоплазматическую мембрану белками SecY, SecE, SecG и SecDF [15, 17, 18]. Белки, предназначенные для транспорта таким путем, имеют в качестве N-концевой сигнальную последовательность, с которой еще во время трансляции связывается белок SecА. Далее SecА-белок самостоятельно или с помощью белка-шаперона SecВ (этот шаперон используется некоторыми белками для предотвращения сворачивания готовой белковой цепи в нативную структуру) доставляет направляющийся на секрецию белок к тому месту внутренней мембраны, где находятся белки SecY, SecE и SecG. Он прикрепляет секретируемый белок к белку SecY и одновременно энергетически обеспечивает процесс транслокации, поскольку обладает АТФ-азной активностью. Транслоцируемый белок через образованную белками SecY, SecE и SecG пору перемещается в периплазматическое пространство, и специальная пептидаза отщепляет от него сигнальную последовательность, после чего освободившийся от связи с мембраной белок может принять нативную конформацию [19].
У грамположительных бактерий помимо основного Sec-пути была обнаружена его разновидность, так называемый ExPortal, который представляет собой уникальный одиночный микродомен клеточной мембраны [20]. На примере Streptococcus pyogenes было показано, что ExPortal функционирует как самостоятельная органелла, координирующая взаимодействия между возникающими вновь и не прошедшими фолдинг секретируемыми белками и связанными с мембраной шаперонами. В связи с этим белок HtrA (DegP), являющийся поверхностным заякоренным аксессорным фактором, необходимым для созревания цистеиновой протеазы SpeB, локализуется исключительно в ExPortal. В Lactococcus lactis для обработки секретируемых пробелков и для созревания их нативных форм используется единственная внеклеточная протеаза HtrA, ответственная за деградацию аномальных белков [21]. Мутации, которые нарушали закрепление HtrA, но не ее протеазную активность, изменяли кинетику созревания цистеиновой проеазы SpeB, демонстрируя, что для ее функционирования важна локализация на ExPortal. Эти данные показывают, что ExPortal обеспечивает механизм, с помощью которого грамположительные бактерии могут координировать секрецию белков и их последующий биогенез в отсутствие специализированного компартмента для фолдинга белков [20].
ТАТ-путь. ТАТ-путь реализуется для белков, у которых в сигнальной последовательности, также расположенной в N-концевой части, имеется характерное сочетание аминокислотных остатков с двумя остатками аргинина: S/TRRXFLK (слева направо: остатки серина или треонина, два остатка аргинина, остаток любой аминокислоты, фенилаланина, лейцина и лизина). Как уже упоминалось, через ТАТ-систему проводятся белки в своей окончательной конформации, и чаще всего это белки, включающие небелковый компонент, например, содержащий металлы-кофакторы. Поэтому канал, который формирует ТАТ-система в мембране, имеет диаметр, позволяющий пройти белкам с поперечными размерами около 70 Å, тогда как пора в Sec-пути имеет диаметр, соответствующий толщине α-спирали белковой молекулы около 12 Å [22]. Для формирования широкого канала объединяются несколько десятков молекул белка TatА, которые первоначально встроены в мембрану как отдельные субъединицы. В тоже время при выведении в периплазму по Sec-пути белка, не принявшего окончательную конформацию, достаточно присутствия в мембране тримера из белков SecY, SecE и SecG [2]. О существенной роли TAT и Sec-систем в жизнедеятельности фитопатогенов свидетельствует тот факт, что мутации в этих системах экспорта белков у P. syringae, Ralstonia solanacearum, Xanthomonas oryzae и Agrobacterium tumefaciens приводили к потери их вирулентности [2].
Система секреции первого типа (T1SS). Сравнительный анализ показывает, что система секреции 1, так же, как система 3 типа (T3SS), секретирует белки через цитоплазматическую и внешнюю мембраны бактерий за одну стадию [2, 3]. Энергозависимые стадии процесса транспорта обеспечивают АТФазы, включающие специфические АТФ-связывающие белки-транслокаторы, названные ABC-транспортерами. Интересно, что ABC-белки являются консервативными и осуществляют трансмембранный перенос большого количества субстратов как в прокариотических, так и в эукариотических клетках. Они, как правило, состоят из двух закрепленных в мембране гидрофобных и двух консервативных гидрофильных АТФ-связывающих доменов, которые могут быть частями как одного, так и нескольких отдельных полипептидов. Большинство бактериальных ABC-транспортных систем включает в себя различные дополнительные белки. Один из них – MFP (membrane fusion protein), обеспечивает слияние цитоплазматической и наружной мембран, и фактически образует канал, через который транспортируется секретируемый белок, другой – OMP (outer membrane protein), так называемый белок-щвейцар (gatekeeper), локализованный во внешней мембране. Его функцией является создание секреторного мембранного канала и его закрытие в отсутствие субстрата [23, 24].
Эксперименты по секреции α-гемолизина на модельной E. coli показывают, что ABC-транспортер и MFP ассоциируются еще до связывания с субстратом, а прикрепление субстрата к этому комплексу индуцирует контакт MFP с OMP. Это соединение является обратимым и разрушается сразу после экспорта субстрата. Энергия гидролиза АТФ посредством ABC-транспортера расходуется только на транслокацию субстрата и не требуется для связывания субстрата или для сборки комплекса [25]. В рамках первой системы секреции функционирует несколько их разновидностей. Так показано, что у азотфиксирующего микросимбионта бобовых R. leguminosarum bv. viciae, функционируют четыре различных вида системы секреции I типа [6]. Один из них состоит из трех компонентов TolC, HlyD и HlyB. Белок TolC является аналогом OMP для экспорта α-гемолизина и представляет собой тримерный комплекс, расположенный во внешней мембране. Предполагается, что он имеет α-складчатый мембранный домен с гидрофильной С-концевой областью, расположенный в периплазматическом пространстве [25]. Периплазматический MFP, аналогом которого является HlyD, также представляет собой тример и взаимодействует как с OMP, так и с ABC-транспортером, роль которого выполняет HlyB. HlyD содержит короткий гидрофильный N-концевой домен, заякоренный во внутренней мембране, включающий около 150 аминокислотных остатков, крупный гидрофобный домен, расположенный в периплазме, включающий 275 аминокислотных остатков, и С-концевой домен, также содержащий 275 аминокислотных остатков [26]. Точная структура HlyB пока не установлена, но предполагается, что он состоит из восьми доменов. Два из них расположены в N-концевой области, и шесть – в центральной гидрофобной области. Также в R. leguminosarum bv. viciae штамм 3841 был обнаружен белок, необходимый для процесса нодуляции, NodO, который секретируется с помощью T1SS и кодируется геном prsDE [6]. Мутация в tatC, которая кодирует Sec-независимую транслоказу белка в R. leguminosarum, продуцирует только белые, не фиксирующие азот клубеньки при инокуляции Pisum sativum [6].
Изучение секреции гемопротеина патогена человека Serratia marcescens и металлопротеазы бактериального возбудителя мягкой гнили растений Erwinia chrysanthemi указывает на существование еще одной модели 1 системы секреции. Согласно этой системе ABC-транспортер и MFP не связываются перед закреплением субстрата. Субстрат в первую очередь связывается с ABC-транспортером, а затем образовавшийся комплекс ассоциируется с MFP, и только потом происходит связывание с OMP, после чего протекает секреция субстрата [25].
В то же время показано, что не все комбинации между компонентами гибридных секреторных систем являются активными. Так, HasE формирует активные экспортеры и с PrtF, и с TolC, тогда как PrtE может формировать активный экспортер только с PrtF, но не с TolC. Исследования этих мультибелковых комплексов in vitro подтвердили существование некоторых функциональных различий между HasE и PrtE. У симбиотического азотфиксатора растений R. leguminosarum в среду роста секретируются 14 субстратов, характерных для I типа. Они не имеют сигнальных N-концевых последовательностей. Вместо них имеются C-концевые секреторные сигналы, расположенные в пределах последних 60 аминокислотных остатков, впервые обнаруженных в молекуле α‑гемолизина [27, 28]. Было установлено, что наименьшая С-концевая последовательность протеазы PrtG возбудителя мягкой гнили растений E. chrysanthemi, позволяющая начать эффективную секрецию, содержит ее последние 29 аминокислотных остатков. Однако низкая, но, все же существенная секреция может быть индуцирована и последними 15 аминокислотными остатками PrtG [29]. Было показано, что С-концевая сигнальная последовательность, состоящая из отрицательно заряженных аминокислотных остатков, является консервативной для гомологичных протеаз [30]. Сравнение последовательностей показало, что липазы тоже имеют сходные с протеазами С-концевые последовательности. Однако секреторные сигналы протеаз и различного рода токсинов различаются и являются специфическими, при этом комплементация между компонентами систем секреции этих двух семейств белков является очень незначительной. Тем не менее, наличие специфического транспортера определяет секрецию любого белка, даже чужеродного [31].
При изучении процессов секреции белков была показана важная роль особой области, расположенной на большинстве экспортируемых субстратов выше С-концевой сигнальной последовательности. Белки, содержащие такую последовательность, принадлежат к семейству RTX (Repeats-in-Toxins), классическими представителями которых являются цитолизин и гемолизин патогенов животных и человека [32]. Аденилатциклазы, токсины, протеазы и липазы, секретируемые системой секреции первого типа, имеют такую же область, состоящую из богатой глицином последовательности (GGXGXD), которая в зависимости от белка повторяется 4–36 раз. Возможно, что богатые глицином повторы действуют как внутренние шапероны, способствуя лучшему разделению секреторного сигнала и белка [31].
У ризобий с помощью T1SS секретируются белки PrsDE, участвующие в регуляции роста бактерий и переносе nod-факторов, в частности, NodO [33]. Этой системой секретируются глюканазы, включенные в процессинг бактериальных экзополисахаридов, и белки адгезии. Ферменты, гидролизующие клеточную стенку растений, целлюлазы и пектиназы, у R. leguminosarum bv. trifolii и viciae попадают на поверхность растительных клеток через систему секреции 1 типа [34]. У Sinorhizobium meliloti этим путем происходит индукция продукции полигалактуроназы [35]. В патогенезе мягкой гнили D. dadantii большая роль принадлежит RTX-протеазам [2]. Две из них секретируются в виде зимогенов, которые активируются после секреции [32].
Система секреции второго типа (T2SS). Эта система секреции устроена уже более сложно и является Sec-зависимой, поэтому секреция с помощью этого аппарата осуществляется в два этапа. Поскольку с помощью T2SS секретируется большое число разнообразных субстратов, его часто называют “общим секреторным путем” (GSP, General Secretory Pathway).
На первом этапе белки экспортируются через цитоплазматическую мембрану, далее они либо остаются в периплазме, либо секретируются через внешнюю мембрану посредством терминальных компонентов системы секреции T2SS [36]. В соответствии с этим различают белковые комплексы, встроенные во внутреннюю (IM) и внешнюю (OM) мембраны. Однако ещe никогда T2SS не была визуализирована в очищенной форме, поскольку ее организация носит динамический характер. В T2SS были выделены четыре мультибелковых комплекса (T2SS subassemblies), включающие псевдопили, комплекс внешний мембраны, платформу цитоплазматической мембраны и секреторную АТФазу, которая находится в цитоплазме и является Zn-зависимым мультисубъединичным ферментным комплексом [9, 36]. Псевдопили представляют собой волокнистые структуры в периплазматическом пространстве, они образованы пятью различными белками-псевдопилинами и включают множество копий основного псевдопилина. Комплекс внешней мембраны образован в основном мультимерным белком-секретином [36]. Платформа цитоплазматической мембраны является тем компонентом T2SS, который обеспечивает структурную целостность транслокационного комплекса и состоит по меньшей мере из четырех мембранных белков. Она взаимодействует с комплексом внешней мембраны, псевдопилями и секреторной АТФазой.
Располагающийся в цитоплазматической мембране канал, состоит из АТФазы – SecA, сигнальной пептидазы и нескольких белков, включающих SecD, SecE, SecF, SecG и SecY. Цитоплазматический шаперон SecВ транспортирует секретируемый белок к этому каналу. Сигнальная пептидаза отщепляет сигнальную последовательность в секретируемом белке, а SecA расщепляет АТФ, благодаря чему белок поступает в периплазматическое пространство, в котором под действием некоторых шаперонов претерпевает конформационные перестройки, необходимые для транспорта через наружную мембрану. Наряду с классическими шаперонами фолдингу способствуют также белки DsbA и Dsb-C, являющиеся дисульфидизомеразами [37]. Функция этих белков заключается в образовании дисульфидных мостиков между остатками цистеина в молекулах белков, которые стабилизируют третичную структуру и необходимы для его функционирования. Дисульфидные мостики образуются в периплазме с участием ферментов DsbA и DsbC. Если зрелый белок должен содержать дисульфидные связи, то без их образования под действием DsbAC секреция за пределы периплазмы, как правило, невозможна. Например, у фитопатогена E. chrysanthemi фермент DsbA необходим для получения почти всех секретируемых факторов вирулентности, стабильности и секреции целлюлаз и нескольких пектиназ, а также для регуляции процесса секреции на различных уровнях метаболизма от транскрипции до секреции белка [37]. Например, у мутанта dsbA экспрессия 25 генов, участвующих в моторике и хемотаксисе, была значительно уменьшена, а флагеллярные белки отсутствовали вовсе.
Как отмечалось ранее, секрецию с помощью второй системы часто называют общим секреторным путем, который имеет несколько разновидностей, называемых “терминальными ветвями общего секреторного пути”. Такой способ секреции хорошо исследован у патогенов животных, а у растительных патогенов, например у эрвиний, показано, что все гены, кодирующие компоненты терминальной ветви, собраны в один кластер размером 13–15 т. п. н., в котором локализовано 12–15 генов, имеющих общее название out или gsp [38].
В настоящее время нет устоявшегося мнения о том, как работает аппарат GSP. Вероятнее всего, многочисленные его компоненты взаимодействуют друг с другом, образуя трансмембранную структуру, простирающуюся от внутренней стороны цитоплазматической мембраны через периплазму вплоть до внешней мембраны [3].
Известно, что первым барьером при встрече патогена с растением является растительная клеточная стенка, состоящая из матрицы полисахаридов, белков и фенольных соединений. При инфицировании возбудителями мягких гнилей растений, к которым относятся различные виды Erwinia, Dickeya, Pectobacterium, Xanthomonas и Ralstonia, система T2SS используется для секреции большого количества широкого спектра гидролаз. Например, D. dadantii выделяет по меньшей мере девять различных пектатлиаз, расщепляющих полигалактуронат [39]. Эта избыточность в секретируемых ферментах, вероятно, обеспечивает эффективность механизмов патогенности. Помимо ферментов, разрушающих клеточные стенки растений, через T2SS секретируются и другие белковые факторы вирулентности фитопатогенов. Так, E. carotovora subsp. atroseptica [40] секретирует белок Svx, гомологичный белку AvrXca у X. campestris, без которого невозможно развитие гнили картофеля. Следует отметить, что система секреции 2 типа высоко специфична. Несмотря на то, что белки–компоненты секреторного аппарата очень сходны друг с другом, их субстраты не будут секретироваться даже близкородственными бактериями. Так, например, пектатлиазы фитопатогена E. carotovora не будут секретироваться из близкородственного вида E. chrysanthemi, несмотря на то, что сходство белков секреторного аппарата превышает 90%. Это свидетельствует о том, что аппарат секреции содержит какой-то видоспецифический компонент. Предполагают, что у D. dadantii и P. carotovorum на эту роль могут претендовать специфические “привратники” белки GSP, OutCGspC и OutDGspD [37].
Система секреции 3 типа (SS3T). Одним из главных инструментов патогенных грамотрицательных бактерий являются белки-эффекторы, секретируемые при помощи транспортной системы третьего типа в клетки растения-хозяина. Например, у P. syringae, подвиды которого паразитируют на широком круге видов растений, было выявлено более 30 эффекторных молекул [41].
Изучение Т3SS начиналось с патогена человека Yersinia enterocolitica. Девять генов, кодирующих белки этой системы (обозначенные как Ysc), высоко консервативны и их гомологи обнаружены у всех бактерий, для которых описана система третьего типа [42, 43]. Восемь из них имеют также гомологию с генами, участвующими в процессах образования жгутиков [44]. Считают, что кодируемые этими генами белки нужны для формирования специфического и находящегося внутри жгутика канала через внутреннюю мембрану, который обеспечивает экспорт компонентов, необходимых для построения собственно жгутика. Один из этих белков содержит типичную АТФ-связывающую последовательность и, очевидно, вовлечен в передачу энергии, необходимой как для сборки “секреторной машины”, так и для собственно процесса секреции. Белок YscC, не имеющий гомологии с белками биогенеза жгутиков, представляет собой типичный секретин [45]. Секреция белков–эффекторов происходит через шприцеподобную структуру, состоящую из 20 различных по структуре белков. При контакте с клеткой-мишенью белки полимеризуются и образуют канал, пронизывающий внутреннюю и наружную мембраны, а на поверхности формируют длинную пилеподобную структуру. Шприцеподобный канал в области наружной мембраны стабилизируется кольцеподобной структурой с большой центральной порой [45]. Отличительной чертой T3SS является доставка секретируемых белков прямо в клетку-мишень, которую называют также инжектосомой (injectosome), а 3ТSS относят к контактной секреции [46].
Когда инжектосома сформирована, секретируемый белок с сигнальным пептидом (20 аминокислотных остатков) в N-концевом участке связывается с цитоплазматическим шапероном, транспортируется по каналу SS3T и впрыскивается в клетку-мишень. Проникновение в клетку хозяина через SS3T сопряжено с полным или частичным разворачиванием белковой молекулы, что предполагает ее взаимодействие с шаперонами хозяина для восстановления структурной конформации и активации глобулы [46]. У растений в качестве таких шаперонов могут выступать пептидилпролил цис/транс изомеразы [47].
У бактерий, взаимодействующих с растениями, белки–эффекторы T3SS изучены в различной степени как у фитопатогенных представителей Pseudomonas, Xanthomonas, Ralstonia и Erwinia [41, 48–50], так и у симбиотических бактерий, в частности у различных видов R. leguminosarum [51, 52]. Патогенные и фитопатогенные бактерии различаются по количеству и организации генов T3SS, что и было взято за основу при ее систематизации. Всего было сформировано семь типов, три из которых представлены в мутуалистических и фитопатогенных бактериях: а − Rhizo у видов Rhizobia, б − Hrp1 (P. syringae и E. amylovora) и в − Hrp2 (Xanthomonas spp., R. solanacearum и Burkholderia spp.) [53].
Несмотря на то, что каждый вид фитопатогена имеет специфический набор эффекторов, которые принципиально можно разделить на две большие группы: Аvr- и Dsp-белки (avirulence и disease specific proteins). Avr-белки узнаются соответствующими белками резистентных растений (R-белки, resistans), что приводит к индукции реакции сверхчувствительности (СВЧ) [54, 55]. Той же системой секретируются хелперы–белки, как правило, не попадающие в цитоплазму растительной клетки, но способствующие транслокации эффекторов. К числу последних относятся богатые глицином харпины, которые не содержат остатки цистеина и являются термостабильными гидрофобными белками, транспортирующимися в межклеточное пространство организма хозяина и, вероятнее всего, изменяющими проницаемость мембраны растительной клетки для облегчения транслокации эффекторов [56].
У E. carotovora ssp. atroseptica с помощью SS3T секретируются харпины HrpN, HrpW и эффекторный белок DspE. При этом харпин HrpW и белок DspE Еса секретируются за пределы клетки и легко обнаруживаются в культуральной среде [57], тогда как харпин HrpJ, хотя и секретируется за пределы клетки, но остается ассоциированным с ее поверхностными структурами и в культуральной среде не обнаруживается. Сочетание хелперного белка HrpN и эффектора DspE играет важную роль в блокировании защитного системного ответа. При этом белок DspE является основным действующим фактором, а хелперный белок HrpN участвует в транслокации DspE в клетки растений [58].
На основании того, что в клетках яблони белок DspE из E. amylovora способен связываться с несколькими мембранными рецепторными киназами, было сделано предположение о том, что аналогичное взаимодействие в клетках картофеля может нарушить сигнальный каскад, приводящий к синтезу медиаторов системного ответа, что в свою очередь способствует успешной пролиферации патогена в организме растения [48].
Для доказательства транслокации отдельных эффекторов в клетки растений была создана остроумная конструкция, содержащая аденилатциклазный домен (Суа) циклолизина Bordetella pertussis. Он выполнял роль биохимического репортера транслокации бактериальных белков в клетки эукариот и успешно применялся для доказательства транслокации Avr-белков в растения из клеток X. campestris [59] и P. syringae [60]. Так, AvrPto – ингибитор протеинкиназы, и AvrPtoB – убиквитин лигаза E3, подавляют рецепторные киназы МАМР (microbe-associated molecular patterns) индуцированного иммунитета растений, ингибируя активность или разрушая через их убиквитинирование такие рецепторные киназы, как рецепторная киназа FLS2 флагеллина, корецепторная киназа BAK1 и рецепторная киназа хитина CERK1 [61]. Установлено, что фосфотреонин лиаза HopAI1 инактивирует MAМP киназу, тирозин фосфатаза HopPtoD2 подавляет реакцию СВЧ, цистеиновая протеаза AvrRpt2 разрушает RIN4, заякоренный в плазматической мембране арабидопсиса белок, действующий как ключевой регулятор иммунитета растений, а ацетилтрансфераза HopZ1a разрушает сеть растительных микротрубочек и везикул [62]. У X. campestris pv. vesicatoria обнаружено 19 генов белков–эффекторов T3SS [47], среди которых хорошо известен эффекторный белок AvrBsT, член семейства белков YopJ/AvrRxv, найденного как у патогенов животных, так и у фитопатогенов. Этот белок, попадая в цитоплазму растения-хозяина, взаимодействует с широким спектром белков, включающих альдегиддегидрогеназу 1 перца (CaALDH1), heatshock белок перца 70a (CaHSP70a), супрессор G2 аллели ofskp1перца (CaSGT1), киназу перца SNF1(SnRK1) и ацетилированный белок из арабидопсиса (ACIP1). Механизм такого взаимодействия показан на примере белка CaSGT1, который связывает одновременно AvrBsT и рецепторную киназу (CaPIK1), локализованную в цитоплазме клеток перца. В результате происходит фосфорилирование AvrBsT и формирование активного комплекса AvrBsT–CaSGT1–CaPIK1. У устойчивых сортов растений это приводит к развитию реакции СВЧ и других защитных ответов [63, 64]. У X. campestris pv. vesicatoria также выявлены гены трех белков Xops (Xanthomonas outer proteins), которые участвуют в адгезии патогена на поверхности клеток растений и индуцируют реакцию СВЧ. Гомологи таких генов найдены и у других фитопатогенов [65]. Следует отметить, что нехозяйские виды растений реагируют на многие эффекторы P. syringae чаще и более интенсивно, чем хозяйские виды, и гомологичные/не гомологичные по последовательностям эффекторы вызывают некрозы в одних и тех же видах растений [50]. На основании этих данных было сделано предположение о том, что такие эффекторы могут взаимодействовать с одинаковыми молекулярными мишенями или сигнальными путями растений [66]. Так, среди изученных эффекторов HopAM1-1 из мутанта P. syringae PtoDC3000 вызывал некроз во всех генотипах испытанных томатов, включая чувствительный сорт для PtoDC3000 [66].
Довольно долго считалось, что система SS3T присуща только грамотрицательным бактериям, в то время как аналогичный механизм транслокации факторов вирулентности грамположительных бактерий не был известен. Лишь недавно на модели “культура кератиноцитов–стрептококк” была предложена схема транслокации факторов вирулентности в эукариотические клетки. По механизму действия она была названа “цитолизин опосредованной транслокацией” [67]. Показано, что в цитоплазматической мембране стрептококков помимо общего секреторного пути (Sec) с помощью стрептолизина О формируются поры диаметром до 30 нм, через которые осуществляется транспорт эффекторных молекул непосредственно в эукариотическую клетку. Эта конструкция вводит, по меньшей мере, один фактор вирулентности, НАД-гликогидролазу (SPN), в цитоплазму кератиноцитов [68]. Чтобы создать поры, необходимые для транслокации SPN, другой белок, SLO, сначала секретируется по пути Sec. SLO является членом класса токсинов, называемых холестерин-зависимыми цитолизинами (CDC), которые связывают холестерин на поверхности эукариотических клеток и внедряются в их мембраны, создавая поры. После образования пор с помощью SLO белок SPN транслоцируется через плазматическую мембрану в эукариотическую клетку также с помощью Sec. Как только он достигает цитозоля клетки хозяина, то расщепляет гликозидную связь β-NAD+ с образованием никотинамида и АДФ-рибозы, мощного вторичного мессенджера, тем самым нарушая нормальные клеточные функции. По этой причине SPN служит основным фактором вирулентности для S. pyogenes. Для патогенов животных этот феномен имеет большое значение, поскольку может быть использован в качестве мишени для действия новых противоинфекционных препаратов. Для фитопатогенов такие исследования пока малочисленны, однако существуют данные о том, что культуральная жидкость вирулентного штамма Clavibacter michiganensis ssp. sepedonicus, возбудителя кольцевой гнили картофеля, инфильтрованная в листья табака, вызывала реакцию СВЧ. На основании этого было высказано предположение, что данный грамположительный фитопатоген секретирует специфические термостабильные белки, близкие по функциям к харпинам [69, 70].
Несмотря на то, что инфицирование симбиотическими азотфиксаторами направлено на установление взаимовыгодных отношений с растениями, на ранних этапах эти бактерии подавляют также фитоиммунитет [71]. В этом процессе SS3T микросимбионтов, в частности Rhizobium [52, 72] и Bradyrhizobium [73], играют существенную роль. Известно, что удаление T3SS предотвращает транспорт эффекторных белков в растение и блокирует развитие симбиоза. Гены, кодирующие T3SSs у Rhizobium, называются rhc и подразделяются на 4 семейства Rhc-1- Rhc-4 [74]. Однако в симбиозе участвует только Rhc-1 [75], а функции белков других семейств пока не установлены. Экспрессия T3SS индуцируется узнаванием растительных флавоноидов путем активации фактора транскрипции TtsI Rhizobium [76]. TtsI инициирует транскрипцию генов T3SS и эффекторных белков путем связывания со специфичными cis-элементами, известными как tts - боксы [77]. Количество и локализация таких боксов варьирует у разных видов. Например, у B. japonicum известно 52 различных tts-боксов. Белки, секретируемые через систему T3SS у симбиотических азотфиксаторов, подразделяют на две категории, образующие пили и эффекторы. К белкам, участвующим в формировании пилей, относят NopA, NopB и NopX, которые проникают через клеточную стенку и плазматическую мембрану растения [51, 71]. Следует отметить, что эффекторы T3SS Rhizobium могут влиять как позитивно, усиливая нодуляцию, так и негативно, ингибируя этот процесс. У Bradyrhizobium sp. методами in silico было выявлено 14 эффекторов T3SS и показано, что у четырех видов растений (Arachis hypogea, Vigna radiata, Crotalaria juncea и Macroptilium atropurpureum) эти эффекторы позитивно влияли на нодуляцию, способствуя увеличению количества клубеньков, а у одного вида (Stylosanthes hamata) воздействовали негативно, уменьшая количество клубеньков и уровень азотфиксации, однако на четыре вида (Aeschynomene afraspera, Indigofera tinctoria, Desmodium tortuosum и A. americana) эти эффекторы не оказывали воздействия [73]. Аналогичный эффект был показан при инокуляции Sinorhizobium fredii вигны (Vigna unguiculata). Таким образом, последствия от действия эффекторов T3SS во многом зависят от того, какой вид бобового растения участвует в симбиозе [78, 79].
Роль эффекторных белков ризобий во многом схожа с их ролью у фитопатогенов. Так, на примере “не азотфиксирующего” растения Nicotiana tabacum показано, что эффекторный белок NopL может фосфорилироваться растительными киназами [80] и влиять на передачу сигналов от митоген-активируемой протеинкиназы (MAPK). При этом NopL способен подавлять развитие клеточной смерти, индуцированной сверхэкспрессией MAP-сигналинга [81]. Известно, что MAP-киназы участвуют в узнавании патогенов на ранних стадиях патогенеза и связаны с активацией R-генов [82]. Эффекторный белок NopT обладает активностью цистеиновой протеазы. Предполагается, что он может индуцировать целевой автопротеолиз клеточных мембран [83]. Некоторые эффекторные белки данной системы осуществляют специфическую регуляцию симбиотической азотфиксации. Так, Nop M обладает Е3 убиквитинлигазной активностью, которая необходима при формировании клубеньков, потому что потеря этой активности приводила к неспособности растений формировать их [84]. У B. elkanii эффекторные белки SS3T взаимодействуют с генами сои ENOD40 и NIN, отвечающими за узнавание Nod-факторов (липополисахаридов ризобий). При этом мутант Bradyrhizobium, дефицитный по SS3T, не был способен индуцировать развитие клубеньков. Таким образом, как у фитопатогенов, так и у мутуалистов большинство эффекторов SS3T направлено на подавление MAMP и ETI (эффектор-запускаемого) типов иммунитета растений [85, 86].
Система секреции 4 типа (SS4T). Система SS4T на основании первичных функций может быть разделена на три подтипа: а − ДНК коньюгация, б − ДНК поглощение/освобождение и в − транслокация белков. T4SS имеет выраженное сходство с аппаратом конъюгации ряда плазмид и, вероятно, произошла от него [2]. По некоторым данным отдельные компоненты этой системы, например, пили и флагеллы, играют существенную роль в подвижности, адгезии бактерий к твердым поверхностям и формировании биопленок [87, 88]. Эта экспортная система характеризуется чрезвычайно широкой специфичностью как по отношению к субстратам (крупные нуклеопротеидные комплексы, мультикомпонентные белковые токсины и мономерные белки), так и по отношению к мишеням секреции, в качестве которых могут служить бактерии, грибы, растения и животные, иными словами – клетки представителей всех царств за исключением архей.
Известными примерами бактериальных патогенов животных, использующих T4SS для проявления вирулентности, являются Neisseria gonorrhoeae L. pneumophila, Brucella suis и Helicobacter pylori, которые используют их для транслокации эффекторных белков в клетки-хозяева во время инфекции [87, 89]. В настоящее время A. tumefaciens является единственным известным фитопатогеном, обладающим T4SS для транслокации T-DNA нуклеопротеинового комплекса в клетки растений [2].
VirB/DT4SS, функционируюшая у A. tumefaciens, содержит 12 белков, обозначенных как VirB1-VirB12 и VirD4 [90]. Самые большие из них являются мембранно-связанными и представлены несколькими копиями. Белки VirB6-10 найдены в периплазме, внутренней и внешней мембранах, и формируют секреторный канал так же, как их акссесорные белки. VirB4, VirB11 и VirD4 локализованы во внутренней мембране и функционируют как АТФазы, обеспечивая систему энергией. Белок VirD4 необходим для связывания других белков перед секрецией через канал. Считается, что субстратная ДНК или белок сначала вступают в контакт с VirD4, функционирующий как молекулярный “затвор” у основания аппарата секреции [91]. Затем VirD4 переносит субстрат к VirB11, который доставляет его к комплексу внутренних мембранных каналов. Наконец, субстрат переносится через периплазму к белковому комплексу наружной мембраны. T4SSs содержит внеклеточные пили, состоящие из самой большой (VirB2) и самой маленькой (VirB5) субъединиц. Точно неизвестно, какую роль пили T4SS играют в процессе секреции. Существует мнение, что, с одной стороны пиль может служить лишь прикрепительным устройством, позволяющим бактериям плотно контактировать с клетками-мишенями [90], а с другой – пиль может выполнять роль канала для транслокации субстрата, особенно в клетки-мишени [92].
Таким образом, конъюгационные системы можно рассматривать как аппарат секреции белка, приспособленный для экспорта ковалентно связанной с белком ДНК. Показано, что конъюгационные системы могут транспортировать и белки, не связанные с ДНК. Так, Tra аппарат плазмиды RP4 секретирует белок RecA [93], а система переноса Т-ДНК способна секретировать VirE2 (SSB белок) и еще один фактор вирулентности, VirF, у патогена человека Yersinia pestis [94].
В отличие от T3SS информация о роли T4SS в симбиозе весьма малочисленна. В ризобиях T4SS характеризуются высокой степенью гомологии с субъединицами VirB/VirD4, обнаруженными в Agrobacterium. Транскрипция T4SS контролируется двухкомпонентным регулятором VirA/VirG [52]. Белок VirA является мембранно-связанной киназой, фосфорилирующей VirG в ответ на внешние факторы [95]. А белок VirG, который связывается с боксами vir, активирует транскрипцию. У Rhizobium эти регуляторы индуцируются флавоноидами, активирующими VirG [96]. Продолжая аналогию с A. tumefaciens, можно отметить, что у обоих видов бактерий найден в С-концевой части одинаковый необходимый для транслокации мотив, заряженный положительно и присутствующий в эффекторных белках [97]. Для транспорта через канал белок VirD4 взаимодействует с этой положительно заряженной сигнальной последовательностью, что обеспечивает бо̀льшую специфичность в транспорте белков [97]. В частности у Mesorhizobium loti она идентифицирована в эффекторных белках Msi059 и Msi061, которые являются у микросимбионтов белками-субстратами SS4T, единственными известными в настоящее время [96]. Msi059 обладает частичным сходством с цистеиновой пептидазой C48. Семейство этих пептидаз содержит белок XopD растительного патогена X. campestris [98]. Ее мишенями в растениях являются конъюгированные белки SUMO [98], разрушение которых мешает им регулировать экспрессию специфических защитных белков. Msi061 имеет также сходство с эффектором VirFA. A. tumefaciens, который взаимодействует с Skp1 (ключевой компонент убиквитинлигазы E3, осуществляющей деградацию белка) растения-хозяина для облегчения деградации эффекторных белков VirE2 и Vip1, участвующих в последующем освобождении Т-ДНК в клетки хозяина [99, 100]. Следует отметить, что у патогенов животных специфические сигнальные последовательности более разнообразны и более сложно устроены.
Система секреции V типа(SS5T). Она называется также системой автотранспортеров и первоначально была описана у N. gonorrhoeae при изучении продукции IgA1-протеазы [101]. Однако несколько позже было показано, что SS5T участвует в секреции целого ряда имеющих отношение к патогенности и вирулентности белков, в частности адгезинов [102]. В этой системе С-концевая последовательность секретируемого белка определяет экспорт N-терминального домена через наружную мембрану, что и послужило основанием для названия этой системы [103]. Большинство автотранспортеров несет N-концевую сигнальную последовательность, разрешающую выход в периплазматическое пространство с использованием Sec-пути. N-концевая сигнальная последовательность примыкает к домену α, за которым следует С-концевой домен β [104]. Последний предназначен для формирования пронизывающей мембрану бочкообразной (barrel) структуры, стенки которой состоят из 10–14 петель полипептидной цепи α-домена. Через такой “бочонок” перемещается домен α, называемый также “пассажирский домен”. После выхода на наружную поверхность клетки судьба “пассажиров” оказывается различной в зависимости от того, что секретируется. Два домена могут оставаться ковалентно или нековалентно связанными, или же α-домен может протеолитически отщепляться и функционировать самостоятельно уже во внеклеточном пространстве. Отщепление может происходить как автокаталитически, поскольку α‑ домены являются протеазами, так и с участием протеаз, локализуемых во внешней мембране. В том случае, когда пассажирский домен остается ковалентно связанным с α-доменом, он в большинстве случаев играет роль адгезина, т.е. фактора патогенности, обеспечивающего прикрепление к тканям организма-хозяина [103, 105, 106]. Такие белки–автотранспортеры присутствуют у широкого круга патогенов животных: AIDA-I – у E. coli, Ag43 и YadA – у Y. enterocolitica и Y. pseudotuberculosis соответственно, адгезин Hia – у H. influenzae и Prn – у Bordetella pertussis. Помимо уже упоминавшейся IgA1-протеазы N. gonorrheae и N. meningitidis, показано участие в патогенезе ряда сериновых протеаз энтеробактерий. Они даже получили собственное название – SPATE (the serine protease autotransporters of Enterobacteriaceae) [107]. К ним относятся токсин у SepA S. flexneri, PrtS у клинических штаммов S. marcescens и протеазы-муциназы Pic и SepA у различных видов шигелл. Из не относящихся к энтеробактериям патогенов, способных секретировать протеазы по пятому пути секреции, можно назвать Haemophilus influenzae (Hap-белок) [108] и Bordetella pertussis (протеаза SphB1) [109]. Белками–аутотранспортерами также являются эстераза EstA P. aeruginosa [110] и токсин VacA Helicobacter pylori [111]. Следует отметить, что все эти белки обладают достаточно большими молекулярными массами, превышающие массы белков, секретируемых через системы I и II типов. В последние годы по мере продвижения исследований получены новые уточняющие данные о компонентах этой системы и, в соответствии с этим, выделены следующие ее подвиды.
Va секреторная система. Секретируемые белки обладают всеми необходимыми признаками автотранспортеров, описанных выше, а автотранспортная субъединица в С-концевой области выполняет роль хелпера, необходимого для секреции через наружную мембрану. Таким образом, белок–эффектор начинает секретироваться в виде предшественника, и в процессе транспорта созревает. Например, предшественник протеазы IgA1 гонококков имеет молекулярную массу 169 кДа, а зрелая протеаза – 106 кДа [112]. Примерами секретируемых таким путем факторов патогенности являются цитолизин ShlA S. marcescens (соответствующий ему TpsВ назван ShlB), цитолизин EthA E. tarda (TpsВ – EthB), адгезины HMW1 и HMW2 H. influenzae (TpsВ – HMW1В и HMW2B соответственно), адгезины LspA1 и LspA2 H. Ducreyi (TpsВ – LspB1 и LspB2 соотвественно) и филаментный гемагглютинин FHA B. pertussis (TpsВ – FhaC) [106]. Все эти белки обладают большими молекулярными массами и состоят из не менее чем 3000 аминокислотных остатков. Наибольший из секретируемых таким путем белков PAO1 был обнаружен при протеомном анализе P. aeruginosa, его молекулярная масса составляла 361 594 Дa, а количество аминокислотных остатков – 5627 [113].
Тип Vb системы. На первый взгляд тип Vb-системы подобен Va-системе, поскольку она содержит пассажирский домен и транслокационный домен, которые представлены двумя отдельными белковыми цепями, но экспрессируются из одного оперона. Этот тип называется также двупартнерской системой (Tps) [106]. Транслоцируемый белок обозначен как TpsB белок, а транспортируемые белки (соответствующие пассажирскому домену типа Va–автотранспортеров) – как TpsA белки. Следует отметить, что белок TpsB, образующий транслокационную пору, содержит дополнительные периплазматические домены, не представленные в классических автотранспортерах.
Тип Vc система. Эта система является наиболее сложной и представляет собой тример, который функционирует в основном как бактериальные адгезины. Это позволило дать им альтернативное название тримерные автотранспортные адгезины (ТАА) [114]. Пассажирские домены Vc весьма разнообразны, тогда как их транслокационный домен высоко консервативен. Vc-система является единственной из разновидностей систем V-типа, у которой транслокационная пора представлена олигомером.
Тип Vd системы. Этот тип был описан недавно и в деталях изучен только на нескольких примерах [115]. Существует мнение, что они относятся к пататин-подобным белкам из P. aeruginosa, обозначенные как PlpD. Их “пассажирский” домен обладает липолитической активностью и разрушается автокаталитически после завершения автотранспорта [115]. Основная структура таких белков сходна со структурой белков Va-системы, содержащих C-концевой транслокационный домен и N-концевой “пассажирский” домен. Однако в отличие от классических автотранспортеров эти домены взаимодействуют через дополнительный периплазматический домен, гомологичный Vb транслокационной поре.
Тип Ve системы. Другое название системы обратный автотранспортер, поскольку он имеет обратный порядок доменов, его C-концевая часть включает в себя “пассажирский” домен, а N-концевая часть образует транслокационную пору [115]. Белок интимин из E. coli и инвазин из Yersinia spp. являются адгезинами с внеклеточным Ig доменом, закрепленным в бактериальной внешней мембране [116]. Интимин осуществляет тесную связь патогенной кишечной палочки с клетками хозяина, что приводит к формированию основания. Для этого второй белок (Tir) секретируется в клетку-хозяина и встраивается в ее плазматическую мембрану изнутри. Секреция и инъекция Tir опосредуется системой секреции 3 типа. Затем Tir действует как специфический рецептор для связывающего домена интимина в клеточной мембране хозяина [117]. Инвазин напрямую связывается с b1-интегринами, которые расположены на поверхности клетки хозяина. На Yersinia spp показано, что “пассажирский” домен является типичной Ig-протеазой и лектино-подобным доменом, который не обнаружен в других подвидах V типа секреции, но который широко распространен в адгезинах из грамположительных бактерий [118]. В N-концевой части полипептидной цепи присутствует маленький периплазматический домен, но он не гомологичен периплазматическому домену из Vb- и Vd-систем и в некоторых случаях содержит пептидогликан-связывающий мотив [118].
В настоящее время не установлено участие T5SS в вирулентности фитопатогенов и микросимбионтов, хотя имеется несколько примеров, описывающих предполагаемые адгезины. У D. dadantii белок, состоящий из более 3800 аминокислотных остатков и гомологичный T5bSS, необходим для бактериальной адгезии к поверхности листьев, агрегации бактерий и стимуляции гибели клеток хозяина [2]. Этот адгезин также содержит каталитический остаток в С-концевой части полипептидной цепи и гомологичен цитотоксической рибонуклеазе, которая является целевой для 16S рибосомальной РНК бактерий. Это предполагает его потенциальную роль в контактно-зависимом торможении роста [119]. Белки-кандидаты T5bSS, состоящие из более чем 3000 аминокислотных остатков, были обнаружены в Xanthomonas axonopodis и Xylella fastidiosa. Такие субстраты также участвуют в адгезии [120, 121]. Предполагается, что XatA T5aSS X. fastidiosa представляет собой большой поверхностный белок [122]. Его роль в адгезии подтверждается тем, что дефицитный мутант xatA не обеспечивал успешную агрегацию бактерий этого вида. Данные in silico также обнаружили белки, связанные с T5SS у E. chrysanthemi и D. dadantii. На основе C-концевых последовательностей, гомологичных белкам, токсичным для бактерий, эти предполагаемые T5SSs субстраты были отнесены к белкам, подавляющим конкурирующие бактерии, но не участвующие в вирулентности [123].
Система секреции 6 типа (T6SS). Эта система была описана относительно недавно. В начале 2000 годов при анализе сиквенсов островков патогенности бактериальных возбудителей болезней животных и растений было зафиксировано консервативное семейство генов, функции которых на тот момент были не ясны [124]. Но к 2006 г. удалось установить, что продукты этих генов формируют специализированную секреторную систему, функционально сходную с системами III и IV типов, которая обеспечивает доставку секретируемого белка непосредственно в цитоплазму хозяйских клеток [125, 126].
В основном эта система представлена в клетках представителей Proteobacteria, в частности Vibrio cholerae, Edwardsiella tarda, P. aeruginosa, Francisella tularensis и Burkholderia mallei, сходные группы генов обнаружены у фитопатогенов, микросимбионтов и почвенных бактерий Agrobacterium tumefaciens, Dickeya dadanti, R. leguminosarum, Pectobacterium atroseptium, Ralstonia solanacearum, P. syringae, P. fluorescens и P. protegens [127]. Предполагают, что у непатогенных бактерий T6SS может участвовать в образовании биопленок, а патогены могли получить гены, кодирующие эту систему, в результате горизонтального переноса [128]. Количество генов, входящих в T6SS-кластеры, колеблется от 15 до 25, и пока еще не все продукты этих генов охарактеризованы структурно и функционально. В структурную часть этой системы входят белки Hcp (hemolysin coregulated protein) [129] и VgrG (valine–glycine repeat protein G) [130], описанные для патогенного штамма E. coli. Гомологи этих белков или кодирующие их гены у других видов бактерий могут иметь другие обозначения, но их наличие считается критерием присутствия у данных бактерий системы T6SS [131].
Было установлено, что выделенный и очищенный белок Hcp способен образовывать кольцевые олигомеры из шести молекул, которые затем объединяются в трубочки с внутренним диаметром 40 Å и длиной около 100 нм [125]. Белок VgrG имеет ярко выраженное структурное сходство с белками бактериофага Т4, которые формируют так называемую “иглу” (the needle complex) на конце хвостового отростка. С помощью этого комплекса бактериофаг прокалывает мембрану и вводит свою ДНК в цитоплазму бактериальной клетки. В белках Gp27 и Gp5, образующих “иглу” бактериофага, в С-концевом домене имеются характерные α-спиральные участки, от которых зависит способность белков проходить через мембрану. Именно такие участки обнаружены в белке VgrG и его аналогах у других бактерий. Как бактериофаг, так и белок VgrG образуют комплекс из трех молекул (тример), который собирается в “иглу” [125]. Последовательность T6SS была определена у пяти разных видов ризобий R. leguminosarum, B. japonicum, M. loti, S. saheli и S. fredii [132, 133], однако ее функциональная роль еще не установлена.
Система секреции 7 типа (T7SS). Существование 7 системы секреции было предсказано в конце 1990 гг. при анализе полной последовательности генома типового штамма возбудителя туберкулеза Mycobacterium tuberculosis H37 Rv и далее подтверждено в ходе экспериментальной работы с другими штаммами M. tuberculosis и другими видами рода Mycobacterium – M. bovis, M. avium и M. marinum [134]. В настоящее время функционирование данной системы показно у многих грамположительных патогенов млекопитающих: Corynebacterium diphtheria, S. aureus, S. agalactiae, Bacillus subtilis, Listeria monocytogenes и Streptomyces coelicolor. Это предполагает, что система секреции типа 7 должна рассматриваться как общая секреторная система для грамположительных бактерий из таксонов Actinobacteria и Firmicutes [4].
Особенностью всех микобактерий является необычное строение их клеточных стенок. По химическому составу, структуре и архитектуре слоев, расположенных над цитоплазматической мембраной эти бактерии относятся к грамположительным, поскольку у них имеется примыкающий к цитоплазматической мембране пептидогликановый слой, но отсутствуют периплазматическое пространство и наружная липопротеиновая мембрана, характерные для грамотрицательных бактерий. Однако в отличие от других грамположительных бактерий у микобактерий, относящихся к отделу Actinobacteria, в клеточной стенке муреиновые комплексы чередуются с комплексами из арабиногалактана, к которым присоединены молекулы миколовых кислот, образующие слой толщиной 8−10 нм [2]. В связи с этим эту часть клеточной оболочки называют микомембраной [135].
Белки, секретируемые T7SS системой, отличаются наличием повторяющихся сочетаний либо остатков пролина и глутаминовой кислоты (РЕ–белки), либо двух остатков пролина и глутаминовой кислоты (РРЕ – белки, где Е – остаток глутаминовой кислоты) [135]. Изучение геномов микобактерий показало, что они имеют кластеры генов, которые кодируют белки, ассоциированные с мембраной и обладающие АТФазной активностью, обычно участвующие в секреции у других бактерий. Они обозначаются как ESX по сокращенной аббревиатуре первого известного представителя этого класса белков ESAT-6 (early secreted antigenic target of 6 kDa ESAT-6 secretion system Х). Соответствующие кластеры генов ESX могут повторяться до 5 раз, сочетаясь с генами различных белков из семейств РЕ и РРЕ, поэтому их назвали ESX-1-ESX-5 [136]. В 2007 г. было предложено все эти варианты относить к секреции 7 типа [137].
Существенно, что количество ESX-систем достаточно хорошо коррелирует с уровнем патогенности микобактерий. Самые патогенные виды рода Mycobacterium (так называемые, медленнорастущие M. avium, M. bovis, M. marinum, M. tuberculosis и M. leprae) имеют в своем геноме все пять кластеров ESX. Быстрорастущие и менее патогенные M. smegmatis и M. gilvum имеют только три таких кластера. Было показано, что только совокупность всех пяти вариантов ESX-систем делает штаммы высоко патогенными [137].
Механизм действия и строение секреторного аппарата типа 7 изучены пока недостаточно, присутствие последнего обнаружено только у патогенов животных и не будет более подробно рассматриваться в данном обзоре. Однако следует отметить, что вредоносные фитопатогены вида Clavibacter michiganensis, относящиеся к грамположительным бактериям, имеют подвиды, паразитирующие на томатах (C. michiganensis subsp. michiganensis) и картофеле (C. michiganensis subsp. sepedonicus) и являются близкими родственниками рода Corynebacterium. Это дает основание надеяться, что компоненты S7TT будут со временем также обнаружены и у данных фитопатогенов.
Система секреции 8 типа (S8TT). Эта система секреции, а также 9 система были обнаружены в начале 2000 гг. [138] и имеют весьма локальное распространение. Они найдены только у некоторых видов патогенов животных. Поскольку они пока не обнаружены у фитопатогенов, их характеристики будут описаны кратко. Более подробно о структурных особенностях системы 8 можно прочитать в работах [139–141].
Различные бактерии способны продуцировать уникальный класс функциональных амилоидов, называемых curl (завиток), которые важны для адгезии, образования биопленок и колонизации поверхностей клеток хозяина [141]. Наиболее подробно структура curl изучена у E. coli, у которой обнаружен – липопротеиновый компонент, CsgG, формирующий канал секреции в бактериальной наружной мембране для транспортировки ее субъединиц. Структурный анализ субъединицы таких амилоидов показал, что секреция через бактериальную внешнюю мембрану осуществляется в развернутой форме с помощью CsgG.
Система секреции 9 (T9SS). Эта система была обнаружена намного позже, чем T8SS и только у некоторых видов грамположительных бактерий Bacteroidetes. T9SS играет различные роли в зависимости от условий обитания бактерий. Она обеспечивает либо тип передвижения (так называемое скользящее движение) у свободноживущих почвенных и водных бактерий (Flavobacterium johnsoniae), либо поставляет факторы вирулентности патогенам (Porphyromonas gingivalis, возбудитель периодонтита человека). У P. gingivalis с помощью этой системы транслоцируются факторы вирулентности через внешнюю мембрану клетки [142]. Белки, предназначенные для секреции, несут консервативный С-концевой домен (CTD), который направляет белок в транслокон мембраны. По крайней мере 18 белков участвуют в этом все еще загадочном процессе, при этом некоторые из них – в посттрансляционной модификации белков T9SS. После транслокации через внешнюю мембрану CTD удаляется протеазой, обладающей сортазоподобной активностью (сортазы – ферменты, ковалентно пришивающие белки к клеточной стенке после того, как они пройдут внешнюю клеточную мембрану). В результате белок-груз может секретироваться во внеклеточную среду или ковалентно прикрепляться к поверхности бактерий. В настоящее время известно, что T9SS регулируется двухкомпонентной системой, однако точные обстоятельства этого процесса еще не изучены. Более подробные сведения об этой системе можно найти в обзоре [142].
Подводя итог рассмотрению систем секреции факторов вирулентности фитопатогенов и мутуалистов, следует отметить, во-первых, значительно меньшую их изученность по сравнению с системами патогенов животных. Во-вторых, обращает на себя внимание тот факт, что наибольшее разнообразие субстратов для систем секреции сосредоточено у фитопатогенов и мутуалистов в системах S2TT и S3TT (табл. 1). Важно также отметить, что, несмотря на принципиальные различия в специализации, основы построения систем секреции, похожи как у патогенов животных, так и фитопатогенов и мутуалистов растений (рис. 1). Проблема дальнейшего изучения особенностей уже известных систем, а также поиск новых, вероятно, долгое время еще будет оставаться актуальной, поскольку полученные знания могут помочь в поисках способов борьбы с фитопатогенами и, возможно, повысить эффективность симбиоза бактерий с растениями.
Таблица 1.
Субстраты, секретируемые системами секреции фитопатогенов и мутуалистов
| Вид | Система секреции бактерий | Специализация бактерий | Ссылка | |||||
|---|---|---|---|---|---|---|---|---|
| S1TT | S2TT | S3TT | S4TT | S5TT | S6TT | |||
| Erwinia sps. | Металлопротеазы | Целлюлаза,
пектатлиаза, полигалактуроназа |
Аvr- и Dsp-белки харпин hrpJ-белок | Адгезин HecA; |
Возбудитель мягкой гнили овощей и фруктов | [7], [25], [57] | ||
| Rhizobium sps. | Глюконазы | Целлюлазы пектиназы; белки PrsDE; | NopA–C, NopJ, NopL, NopMNopPNopT, NopX | Аналоги VgrG; Hcp |
Симбиотический азотфиксатор бобовых | [33], [34], [51], [71], [83], [84], [127] | ||
| Sinorhizobi-um meliloti | Полигалак-туроназы | Симбиотический азотфиксатор бобовых | [35] | |||||
| Bradyrizobium japonicum | Гем цитохрома С | Белки симбиоза:NopP GunA, Blr1806, Blr1649 | Аналоги VgrG; Hcp |
Симбиотический азотфиксатор бобовых | [85], [86] | |||
| Agrobacterium tumefaciens | Β-1,2-глюкан | Бактериальная ДНК; | Аналоги VgrG; Hcp |
Возбудитель рака картофеля |
[2] | |||
| Xanthomonas sps. | Полигалак- туроназы РghAxc PghBxc амилаза, эндоглюконаза | Эффектор
XopXccN; Эффектор AvrBs3 |
Транс-гликози-даза НpaH; эффекторы AvrBs2, XopD, XopN, XopX | Филамент- ный белок XacFhaB Адгезины XadA, XadBYapH |
Возбудитель мягкой гнили | [63], [64], [98], [127] | ||
| Pseudomonas syringae | Эффекторы AvrRpm1; AvrD; HopPtoE; AvrB; | Аналоги VgrG Hcp |
Возбудитель бактериального ожога растений |
[66], [127] | ||||
| Clavibacter michiganensis sps. | Целлюлаза CelA; Пектиназа |
Предполагаемые харпины | Возбудитель мокрой гнили кар-тофеля и томатов | [69], [70] | ||||
| Dickeya Dadantii | Целлюлаза,
пектатлиаза, Полигалак- туроназа |
Предполагаемые адгезины | Аналоги VgrG Hcp |
Возбудитель гнили картофеля | [2], [39], [127] |
|||
| Xylella fastidiosa | Адгезины | Бактериоз винограда | [120–122] | |||||
Список литературы
Зигангирова Н.А., Нестеренко Л.Н., Тиганова И.Г., Кост Е.А. // Молекулярная генетика, микробиология, вирусология. 2012. № 3. С. 1–13.
Chang J.H., Desveaux D., Creason A.L. // Annu. Rev. Phytopathol. 2014. V. 52. № 8. P. 317–345.
Green E.R., Mecsas J. // Microbiol. Spectr. 2016. V. 4. № 1. P. 1–32.
Schneewind O., Missiakas D. // Biochim. Biophys. Acta. 2014. V. 1843. № 8. P. 1687–1698.
Collmer A., Badel J.L., Charkowski A.O., Deng W-L., Derrick E. Fouts D.E., Ramos A.R., Rehm A.H, Anderson D.M., Schneewind O., van Dijk K., Alfano J.R. // Proc. Natl. Acad. Sci. USA. 2000. V. 97. № 16. P. 8770–8777.
Schmeisser C., Liesegang H., Krysciak D., Bakkou N., Le Que ´re A., Wollherr A., Heinemeyer I., Morgenstern B., Pommerening-Röse R., Flores M., Palacios R., Brenner S., Gottschalk G., Schmitz R.A., Broughton W.J., Perret X., Strittmatter A.W., Wolfgang R., Streit W.R. // Appl. Environ. Microbiol. 2009. V. 75. № 12. P. 4035–4045.
Akatsuka H., Binet R., Kawai E., Wandersman C., Omori K. // J. Bacteriol. 1997. V. 179. № 15. P. 4754–4760.
Henderson I.R., Navarro-Garcia F., Desvaux M., Fernandez R.C., Ala’Aldeen D. // Microb. Mol. Biol. Rev. 2004. V. 68. № 4. P. 692–744.
Johnson T.L., Jan Abendroth J., Hol W.C.J., Sandkvist M. // FEMS Microbiol. Lett. 2005. V. 255. № 2. P.175–186.
Soto M., Sanjua’n J., Olivares J. // Microbiol. 2006. V. 152. № 10. P. 3167–3174.
Houben E.N.G., Korotkov K.V., Bitter W. // Biochim. Biophys. Acta. 2014. V. 1843. № 3. P. 1707–1716.
Ho B.T., Tao G. Dong T.G., Mekalanos J.J. // Cell Host. Microbe. 2014. V. 15. № 1. P. 9–21.
Patel R., Sarah M., Smith S.M., Robinson C. // Biochim. Biophys. Acta. 2014. V. 1843. № 8. P. 1620–1628.
Bensing B.A., Seepersaud R., Yen Y.T., Sullam P.M. // Biochim. Biophys. Acta. 2014. V. 1843. № 8. P. 1674–1686.
Tsukazaki T. // FEMS Microbiol. Lett. 2018. V. 365. № 18. P. 1–9.
Wu G., Su P., Bo Wang B., Zhang Y., Guoliang Qian G., Liu F. // Phytopathol. 2017. V. 107. № 9. P. 1011–1021.
Chatzi K.E., Sardis M.F., Economou A., Karamanou S. // Biochim. Biophys. Acta. 2014. V. 1843. № 8. P. 1466–1474.
Kuhn A., Kosh H.-G., Dalby R.E. // EcoSal. Plus. 2017. V. 7. № 2. P. 1–8.
Catipovic M.A., Benedikt W., Bauer B.W., Loparo J., Rapoport T.A. // EMBO J. 2019. V. 15. pii: e101140.
Rosch J.W., Caparon M.G. // Mol. Microbiol.2005. V. 58. № 4.P. 959–968.
Poquet I., Saint V., Seznec E., Simoes N., Bolotin A., Gruss A. // Mol. Microbiol. 2000. V. 35. № 5. P. 1042–1051.
Patel R., Smith S.M., Robinson C. // Biochim. Biophys. Acta. 2014. V. 1843. № 8. P. 1620–1628.
Khezri J., Ahmadpour F., Talebi S. // J. Appl. Biotech.Rep. 2016. V. 3. № 3. P. 437–440.
Morgan J.L.W., Acheson J.F., Zimmer J. // Structure. 2017. V. 25. № 3. P. 522–529.
Thanassi D.G., Hultgren S.J. // Cur. Opin. Cell. Biology. 2000. V. 12. № 4. P. 420–430.
Fath M.J., Kolter R. // Microbiol. Rev. 1993. V. 57. № 4. P. 995–1017.
Fauvart M., Michiels J. // FEMS Microbiol. Lett. 2008. V. 285. № 1. P. 1–9.
Krehenbrink M., Downie J.A. // BMC Genomics. 2008. V. 29. № 1. P. 9–55.
Ghigo J-M., Wandersman C.B. // J. Biol. Chem. 1994.V. 269. № 12. P. 8979–8985.
Palacios J.L., Zaror I., Martinez P., Ribe F., Opazo P., SociI’as T., Gidekel M., Venegas A. // J. Bacteriol. 2001. V. 183. № 4. P. 1346–1358.
Binet R., Le’toffe S., Ghigo J.M., Delepelaire P., Wandersman C. // Gene. 1997. V. 192. № 3. P. 7–11.
Linhartova I., Osicka R., Bumba L., Sebo P. Microbial Toxins / Eds. Gopalakrishnakone P., Stiles B., Alape-Girón A., Dubreuil J.D., Mandal M. Dordrecht: Springer Sci., Business Media B.V., 2018. 502 p.
Finnie C., Hartley N.M., Findlay K.C., Downie J.A. // Mol. Microbiol. 1997. V. 25. № 1. P. 135–146.
Robledo M., Rivera L., Jiménez-Zurdo J., Rivas R., Dazzo F., Velázquez E., Martínez-Molina E., Hirsch A.M., Mateos P.F. // Microb. Cell Factories. 2008. V. 11. № 3. P. 125–133.
Hu C.Y., Lin L.P. // World J. Microb. Biot. 2003. V. 19. № 5. P. 515–522.
Korotkov V., SandkvistM., Hol W.G.J. // Nat. Rev. M-icrobiol. 2013. V. 10. № 5. P. 336–351.
Nivaskumar M., Francetic O. // Biochim. Biophys. A-cta. 2014. V. 1843. № 8. P. 1568–1577.
Lallemand M., Login F.H., Guschinskaya N., Pineau C., Effantin G., Robert X., Shevchik V.E. // PLoS ONE. 2013. V. 8. № 11. P. e79562.
Expert D., Patrit O., Shevchk V.E., Perino C., Boucher V., Creze C., Wenes E., Fagard M. // Mol. Plant. Pathol. 2018. V. 19. № 2. P. 313–327.
Corbett M., Virtue S., Bell K., Birch P., Burr T., Hyman L., Lilley K., Poock S., Toth I., Salmond G. // Mol. Plant. Microbe. Interact. 2005. V. 18. № 4. P. 334–342.
Crabill E., Joe A., Block A., van Rooyen J.M., Alfano J.R. // Plant Physiol. 2010. V. 154. № 1. P. 233–244.
Ozturk G., LeGrand K., Zheng Y., Young GM. // FEMS Microbiol. Lett. 2017. V. 364. № 20. P. 1–7.
Alfano J.R., Collmer A. // Annu. Rev. Phytopathol. 2004. V. 42. № 1. P. 385–414.
Koo J., Ryan P., Lamers R.P., John L. Rubinstein J.L., Burrows L.L., Howell P.L. // Structure. 2016. V. 24. № 10. P. 1778–1787.
Terashima H., Kawamoto A., Tatsumi C., Namba K., Minamino T., Imada K. // mBio. 2018. V. 9. № 3. P. 1–13.
Мокрякова М.В., Абдеева И.А., Пирузян Э.С., Шаад Н.В., Игнатов А.Н. // Микробиология. 2010. Т. 79. № 1. С. 63–71.
Николайчик Л.Л., Хомская Е.И., Игнатенко Е.И. // Труды БГУ. 2009. Т. 4. С. 1–7.
Collmer A., Badel J.L., Charkowski A.O, Deng W-L., Derrick E., Fouts D.E., Ramos A.R., Rehm A.H., Anderson D.M., Schneewind O., van Dijk K., Alfano J.R. // Proc. Natl. Acad. Sci. USA. 2000. V. 97. № 16. P. 8770–8777.
Puigvert M., Guarischi-Sousa R., Zuluaga P., Coll N.S., MachoA.P., João C., Setubal J.C.,Valls M. // Fron. Plant Sci. 2017. V. 8. Article 370. https://doi.org/10.3389/fpls.2017.00370
Saad M.M., Staehelin C., Broughton W.J., Deakin W.J. // J. Bacteriol. 2008. V. 190. № 2. P. 750–754.
Nelson M. S., Sadowsky M.J. // Front. Plant Sci. 2015. V. 6. P. 491–496.
Büttner D. // Microbiol. Mol. Biol. Rev. 2012. V. 76. № 2. P. 262–310.
Hauck P., Roger Thilmony R., He S.Y. //. Proc. Natl. Acad. Sci. USA. 2003. V. 100. № 14. P. 8577–8582.
Bergeron J.R.C., Fernández L., Wasney G.A., Vuckovic M., Reffuveille F., Hancock R.E.W., Natalie C.J., Strynadka N.C.J. // J. Biol. Chem. 2016. V. 291. № 4. P. 1676–1691.
Choi M.S., Kim W., Lee C., Oh C.S. // Mol. Plant. Microbe. Interact. 2013. V. 26. № 10. P. 1115−1122.
Лагоненко А.Л., Овчинникова Т.В., Николайчик Е.А., Евтушенков А.Н. // Докл. HAH Беларуси. 2004. Т. 48. № 5. С. 74−78.
Лагоненко А.Л., Николайчик E.А., Евтушенков A.H. // Докл. HAH Беларуси. 2006. Т. 50. № 1. С. 70−75.
Casper-Lindley C., Dahlbeck D., Clark E.T., Staskawicz B.J. // Proc. Natl. Acad. Sci. USA. 2002. V. 99. № 12. P. 8336−8342.
Schechter L.M., Roberts K.A., Jamir Y. // J. Bacteriol. 2004. V. 186. № 2. P. 543−549.
Block A., Alfano J.R. // Curr. Opin. Microbiol. 2011. V. 14 № 1. P. 39–46.
Ma K-W., Jiang S., Hawara E., Lee D.H., Pan S., Coaker G., Song J., Ma W. // New. Phytol. 2015 V. 208. № 4. P. 1157–1168.
Han S.W., Hwang B.K. // Planta. 2016. V. 245. № 2. P. 237−253.
Mou S., Gao F., Shen L., Yang S., He W., Cheng W., Wu Y., He S. // BMC Plant Biol. 2019. V. 19. № 1. P. 28−33.
Roden J.A., Belt B., Ross J.B., Tachibana T., Vargas J., Mudgett M.B. //Proc. Natl. Acad. Sci. USA. 2004. V. 101. № 47. P. 16624–16629.
Wroblewski T., Caldwell K.S., Piskurewicz U., Cavanaugh K.A., Xu H., Kozik A., Ochoa O., McHale L.K., Lahre K., Jelenska J., Castillo J.A., Blumenthal D., Vinatzer B.A, Greenberg J.T., Michelmore R.W. // Plant Physiol. 2009. V. 150. № 4. P. 1733–1749.
Madden J.C., Ruiz N., Caparon M. // Cell. V. 104. № 12. P. 143−152.
Gauthier A., Finlay B.B. // Cur. Biol. 2001. V. 11. № 3. P. 264−267.
Бояркина С.В., Омеличкина Ю.В., Волкова О.Д., Еникеев А.Г., Верхотуров В.В., Шафикова Т.Н. // Известия ВУЗОВ. Прикл. хим. биотехнол. 2016. Т. 6. № 3. С. 42−49.
Nissinen R., Lai F-M., Laine M.J., Metzler M.C. // Phytopathol. 1997. V. 87. № 7. P. 678−684.
Deakin W.J., Broughton W.J. // Nat. Rev. Microbiol. 2005. V. 7. № 4. P. 313−316.
Soto M. J., Sanjua’n J., Olivares J. // J. Microbiol. 2006. V. 152. № 11. P. 3167–3174.
Songwattana P., Noisangiam R., Teamtisong K., Prakamhang J., Tittabutr P., Piromyou P., Boonkerd N., Eric Giraud E., Teaumroong N. // Front. Microbiol. 2017. V. 8. article 707. https://doi.org/10.3389/fmicb.2017.01810
Gazi A.D., Sarris P.F., Fadouloglou V.E., Charova S.N., Mathioudakis N., Panopoulos N.J. // BMC Microbiol. 2012. V. 12. № 188. P. 1–15.
Tampakaki A. // Front. Plant Sci. 2014. V. 5. article 114. https://doi.org/10.3389/fpls.2014.00114
Kobayashi H., Naciri-Graven Y., Broughton W.J., Perret X. // Mol. Microbiol. 2004. V. 51. № 2. P. 335–347.
Wassem R., Kobayashi H., Kambara K., Le Quéré A., C. Walker G.C., Broughton W.J., Deakin W.J. // Mol. Microbiol. 2008. V. 68. № 3. P. 736–748.
Skorpil P., Saad M.M., Boukli N.M., Kobayashi H., Ares-Orpel F., Broughton W.J., Deakin W.J. // Mol. Microbiol. 2005. V. 57. № 5. P. 1304–1317.
López-Baena F.J., Ruiz-Sainz J.E., Rodríguez-Carvajal M.A., Vinardell J.M. // Int. J. Mol. Sci. 2016. V. 17. № 5. P. 755−777.
Bartsev A.V., Deakin W.J., Boukli N.M., McAlvin C.B., Stacey G., Malnoë P., Broughton W.J., Staehelin C. // Plant Physiol. 2004. V. 134. № 2. P. 871–879.
Ge Y-Y., Qi-WangD., Xiang Q-W., Wagner C., Zhang D., Zhi-Ping O-Y., XieZ-P., Staehelin C. // J. Exp. Bot. 2016. V. 67. № 7. P. 2483–2494.
Pedley K.F., Martin G.B. // Curr. Opin. Plant Biol. 2005. V. 8. № 5. P. 541−547.
Fotiadis T.D., Dimou M., Geograkopoulos D.G., KatinakisP., Tampakaki A.P. // FEMS Microbiol. Letter. 2012. V. 327. № 1. P. 66−77.
Xin D.W., Liao S., Xie Z.P., Hann D.R., Steinle L., Boller T. // PLoS Pathog. 2012. 8:e1002707. https://doi.org/10.1371/journal.ppat.1002707
Bigeard J., Colcombet J., Hirt H. // Mol. Plant. 2015. V. 8. № 4. P. 521–539.
Brauer E.K., George V. Popescu G. V., Dharmendra K. Singh D.K.,Calviño N., Gupta K., Gupta B., Chakravarthy S., Popescu S.C. // PLoS Biol. 2018. V. 16. № 12. P. 1−24.
Calviño N., Gupta K., Gupta B., Chakravarthy S., Popescu S.C. // PLoS Biol. 2018. V. 16. № 12. P. 1−24.
Melville S., Craig L. // Microb. Mol. Biol. Rev. 2013. V. 77. № 3. P. 323–341.
Oki H., Kawahara K., Maruno T. // Proc. Natl. Acad. Sci. USA. 2018. V. 115. № 12. P. 7421−7427.
Hamilton H.L., Dillard J.P. // Mol. Microbiol. 2006. V. 59. № 2. P. 376–385.
Fronzes R., Peter J., Christie P.J., WaksmanG. // Nat. Rev. Microbiol. 2009. V. 7. № 1. P. 703−714.
Atmakuri K., Cascales E., Christie P.J. // Mol. Microbiol. 2004. V. 54. № 5. P. 1199–1211.
Waksman G. // EMBO Rep. 2019. V. 20. № 3. P. 1−16.
Smillie C., Garcillán-Barcia M.P., Francia V., Rocha P.C., de la Cruz F. // Microbiol. Mol. Biol. Rev. 2010 V. 74. № 3. P. 434–452.
Thoerner C.I., Bin Kingombe K., Bögli-Stuber B., Bissig-Choisat B., Wassenaar T.M., Frey J., Jemmi E.T. // Appl. Environ. Microbiol. 2003 V. 69. № 3. P. 1810–1816.
Lin Y.H., Gao R., Binns A.N., Lynn D.G. // Adv. Exp. Med. Biol. 2008. V. 631. № 1. P. 161−177.
Hubber A.M., Sullivan J.T., Ronson C.W. // Mol. Plant Microbe. Interact. 2007. V. 20. № 3. P. 255–261.
Vergunst A.C., van Lier M.C.M., den Dulk-Ras A., Stüve T.A.G., Ouwehand A., Hooykaas P.J.J. // Proc. Natl. Acad. Sci. USA. 2005. V. 102. № 3. P. 832–837.
Hotson A., Chosed R., Shu H., Orth K., Mudgett M.B. // Mol. Microbiol. 2003. V. 50. № 2. P. 377–389.
Schrammeijer B., Risseeuw E., Pansegrau W., Regensburg-Tuïnk T.J., Crosby W.L., Hooykaas P.J. // Curr. Biol. 2001. V. 11. № 4. P. 258–262.
Shi Y., Lee L.Y., Gelvin S.B. // Plant J. 2014. V. 79. № 5. P. 848−860.
Jahnig J.J.F., Meyer T.F. // Mol. Microbiol. 1995. V. 18. № 2. P. 378−380.
Leo J.C., Grin I., Linke D. // Philos. Trans. R. Soc. Lond. B. Biol. Sci. 2012. V. 367. № 4. P. 1088–1101.
Henderson I.R., Navarro-Garcia F., Desvaux M., Fernandez R.C., Ala’Aldeen D. // Microbiol. Mol. Biol. Rev. 2004. V. 68. № 12. P. 692–744.
Szabady R.L., Peterson J.H., Skillman K.M., Bernstein H.D. // Proc. Natl. Acad. Sci. USA. 2005. V. 102. № 1. P. 221–226.
Ulsen P., Rahman S.,Wouter S.P., Jong W.S.P., Daleke-Schermerhorn M.H., Luirink J. // Biochim. Biophys. Acta. 2014. V. 1843. № 3. P. 1592–1611.
Fan E., Chauhan N., Udatha D.B.R.K., Leo J.C., Linke D. // Microbiol. Spectrum. 2016. V. 4. № 1. P. 1−24.
Andrade F.B., Abreu A.G., Nunes K.O., Gomes T.A.T., Piazza R.M.F., Elias W.P. // Infect. Genet. Evol. 2017. V. 50. № 6. P. 83−86.
Spahich N.A., Geme J.W.St. // Front. Cell Infect. M-icrobiol. 2011. V. 1. Article 5. https://doi.org/10.3389/fcimb.2011.00005
Coutte L., Antoine R., Drobecq H., Jacob-Dubuisson F. // EMBO J. 2001. V. 20. № 8. P. 5040−5048.
Wilhelm S.., Gdynia A., Tielen P., Rosenau F., Jaeger K.E. // J. Bacteriol. 2007. V. 189. № 18. P. 6695−6703.
Foegeding N.J., Caston R.R., McClain M.S., Ohi M.D., Cover T.L. // Toxins (Basel). 2016. V. 8. № 5. P. 173−177.
Bernstein H.D. // Eco. Sal. Plus. 2019. V. 8. № 1. P. 10–15.
Ma Q., Zhai Y., Schneider J.C., Ramseier T.M., Saier M.N. // Biochim. Biophys. Acta. 2003. V. 1611. № 1−2. P. 223–233.
Nummelin H., Merckel M.C., Leo J.C., Lankinen H., Skurnik M., Goldman A. // EMBO J. 2004.V. 23. № 4. P. 701–711.
Salacha R., Kovacić F., Brochier-Armanet C., Wilhelm S., Tommassen J., Filloux A., Voulhoux R., Bleves S. // Environ. Microbiol. 2010. V. 12. № 6. P. 1498−1512.
Fairman J.W., Dautin N., Wojtowicz D., Liu W., Noina N.J., Barnard T.J., Udho E., Przytycka T.M., Cherezov V., Buchanan S.K. // Structure. 2012. V. 20. № 7. P. 1233–1243.
Lai Y., Rosenshine I., Leong J., Frankel G. // Cell. Microbiol. 2013. V. 15. № 11. P. 1796–1808.
Heinz E., Christopher J. Stubenrauch C.J., Grinter R., Nathan P. Croft N.P., Purcell A.V., Strugnell R.A., Dougan G., Lithgow T. // Genome. Biol. Evol. 2016. V. 8. № 6. P. 1690–1705.
Walker D., Lancaster L., James R., Kleanthous C. // Protein Sci. 2004. V. 13. № 1. P. 1603–1611.
Gottig N., Garavaglia B.S., Garofalo C.G., Orellano E.G., Ottado J. // PLoS ONE. 2009. V. 4. № 2. P. e4358. https://doi.org/10.1371/journal.pone.0004358
Voegel T.M., Warren J.G., Matsumoto A., Igo M.M., Kirkpatrick B.C. // Microbiol. 2010. V. 156. № 5. P. 2172–2179.
Katsumoto A., Huston S.L., Killiny N., Igo M.M. // Micro-biol. Open. 2012. V. 1. № 1. P. 33–45.
Ruhe Z.C., Low D.A., Hayes C.S. // Trends Microbiol. 2013. V. 21. № 2. P. 230–237.
Sarris P.F., Skandalis N., Kokkinidis M., Panopoluos N.P. // Mol. Plant Pathol. 2010. V. 11. № 6. P. 795–804.
Silverman J.M., Brunet Y.R., Cascales E., Mougous J.D. // Annu. Rev. Microbiol. 2012. V. 66. № 4. P. 453–472.
Ho B. T., Dong T. G., Mekalanos J.J. // Cell Host Microbe. 2014. V. 15. P. 9–21.
Ryu C.-M. // J. Microbiol. 2015. V. 53. № 3. P. 201–208.
Thomas J., Watve S.S., Ratcliff W.C., Hammer B.K. // mBio. 2017. V. 25. № 8. P. e00654-17. https://doi.org/10.1128/mBio.00654-17
Silverman J.M., Agnello D.M., Zheng H., Andrews B.T., Li M., Catalano C.E., Gonen T., Mougous J.D. // Mol. Cell. 2013. V. 51. № 5. P. 584−593.
Zong B., Zang Y., Wang X., Liu M., Zhang T., Zhu Y., Zheng Y., Hu L., Li P., Chen H., Tan C. // Virulence. 2019. V. 10. № 1. P. 118–132.
Decoin V., Barbey C., Bergeau D., Latour X., Feuilloley M.G.J., Orange N., Merieau A. // PLoS One. 2014. V. 9. № 2. P. e89411. https://doi.org/10.1371/journal.pone.0089411
Bladergroen M.R., Badelt K., Spaink H.P. // Mol. Plant Microbe. Interact. 2003. V. 16. № 1. P. 53–64.
Bingle L.E., Bailey C.M., Pallen M.J. // Curr. Opin. Microbiol. 2008. V. 11. № 1. P. 1–6.
Houben E. N.G., Korotkov K.V., Bitter W. // Biochim. Biophys. Acta. 2014. V. 1843. № 8. P. 1707–1716.
Dalekea M.H., Ummelsa R., Bawonoc P., Heringac J., Vandenbroucke-Graulsa C.M., Luirinkb J., Bittera W. // Proc. Natl. Acad. Sci. USA. 2012. V. 109. № 28. P. 11342–11347.
Bottai D., Gröschel M.I., Brosch R. // Curr. Top. Microbiol. Immunol. 2017. V. 404. № 4. P. 235−265.
Ates L.S., Houben E.N., Bitter W. // Microbiol. Spectrum. 2016. V. 4. № 1. P. 1−21.
Chapman M.R., Robinson L.S., Pinkner J.S., RothR., Heuser J., Hammar M., Norm S. // Science. 2002. V. 295. № 8. P. 851−855.
Gibson D.L., White A.P., Rajotte C.M., Kay W.W. // Microbiol. 2007. V. 153. № 4. P. 1131−1140.
Cao B., Zhaoa Y., Koua Y., Nia D., Zhanga X.C., Huanga Y. // Proc. Natl. Acad. Sci. USA. 2014. V. 111. № 50. P. E5439–E5444.
Depluverez S., Devos S., Devreese B. // Front. Microbiol. 2016. V. 7. article 1336–1339. https://doi.org/10.3389/fmicb.2016.01336
Lasica A.M., Ksiazek M., Madej K.M., Potempa M. // Front. Cell. Infect. Microbiol. 2017. V. 7. article 215. https://doi.org/10.3389/fcimb.2017.00215
Дополнительные материалы отсутствуют.
Инструменты
Прикладная биохимия и микробиология


