Журнал аналитической химии, 2020, T. 75, № 4, стр. 316-326
Большие данные в современном химическом анализе
Б. Л. Мильман a, *, И. К. Журкович b, **
a Институт экспериментальной медицины
197376 Санкт-Петербург, ул. Акад. Павлова, 12, Россия
b Институт токсикологии Федерального медико-биологического агентства России
192019 Санкт-Петербург, ул. Бехтерева, 1, Россия
* E-mail: bmilman@mail.rcom.ru
** E-mail: zhurkovich.i.k@toxicology.ru
Поступила в редакцию 27.11.2018
После доработки 09.02.2019
Принята к публикации 31.07.2019
Аннотация
Представлен обзор научных публикаций, в которых отражено получение и использование больших данных в современной аналитической химии. Такие данные характеризуются значительными объемами, потоками и разнообразием. Их генерация и манипуляции с ними сопровождают анализ биообразцов и образцов другого происхождения методами хроматографии и масс-спектрометрии. Большие данные, получаемые этими методами, обеспечивают мультианалитный анализ проб, хотя характеристики обнаружения, идентификации и количественного определения удовлетворительны не для всех аналитов. Применение простых аналитических систем также может быть сопряжено с получением большого количества данных. Огромный пласт информации содержится в больших химических базах данных, использование которых необходимо при нецелевом анализе. Отбор кандидатов на идентификацию учитывает распространенность (цитируемость) химических веществ; идентификация включает использование справочных библиотек масс-спектров. Методы обработки, анализа и представления данных (статистика, хемометрия) прогрессируют вместе с ростом объема информации. Технические характеристики компьютеров и их сетей растут опережающими темпами, создавая потенциал развития методов анализа информации и обеспечивая новые возможности межлабораторного сотрудничества.
Химический анализ давно рассматривают как специфический процесс получения новой информации о природе и составе вещества [1]. Характер и объем информации значительно изменяются вместе с развитием науки и технологии. Современная аналитика обеспечивает огромное количество новой информации, связанной с тем, что в последнее время принято называть “большими данными”1 (big data) [3–6].
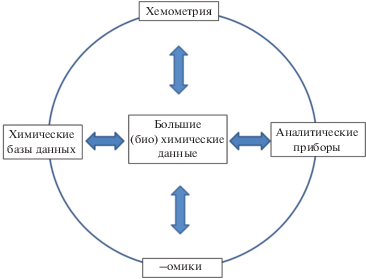
Первичный источник больших данных – результаты работы аналитических приборов (рис. 1), которые в наиболее сложном варианте генерируют многокомпонентные аналитические сигналы. Такого рода сигналы обычны для биологических/медицинских образцов в геномике, протеомике, метаболомике и других новых разделах биологии (биомедицины), объединяемых терминов “–омики”. Кроме того, вне зависимости от сложности самих сигналов, большие данные могут быть следствием большого потока анализируемых проб, обилия аналитических измерений. При обработке полученных данных, проводимой, например, с целью группировки и классификации анализируемых проб, широко применяют хемометрические методы. Они получили дополнительное развитие при росте количества информации. Хранение больших данных и полученной из них информации, их масштабная обработка – все это стало доступным при сопутствующем развитии хемоинформатики, выразившемся, в частности, в постоянной модернизации химических баз данных (рис. 1).
Проблемы, возникающие при переходе от небольших объемов данных к их большому количеству, затрагивает многие науки и их разделы. Современный химический (биохимический) анализ не является исключением. Между тем, взгляд на большие массивы информации под углом зрения профессиональных химиков этой специализации недостаточно отражен в научной литературе. Поэтому обзор работ, связанных с получением, обработкой и использованием больших данных в современной аналитике, представляется актуальным. Настоящая статья представляет собой краткий обзор такой направленности. Учтены преимущественно публикации последних пяти−семи лет. Необходимо отметить, что в литературе почти нет работ, полностью соответствующих теме, название которой вынесено в заголовок статьи. Тем не менее, релевантные фрагменты и выводы многих оригинальных публикаций хорошо дополняют друг друга, что позволило нам использовать “метод информационного синтеза” и очертить место и роль больших данных в химико-аналитических исследованиях и практическом анализе, включить актуальные примеры качественного и количественного определения. Другие трактовки затронутых вопросов, их более полное освещение можно найти в обзорных статьях [3–10].
ОБЩИЕ СВЕДЕНИЯ О БОЛЬШИХ ДАННЫХ
Большие данные характеризуются несколькими общими параметрами, английские названия которых начинаются на букву “v” [3–10].
Объем данных (volume). В табл. 1 охарактеризованы некоторые массивы больших данных (большие массивы информации). В аналитической и биоаналитической химии к ним относят данные, объем которых, например, измеряется в гигабайтах, если речь идет о единичном анализе, или в терабайтах в случае многих проб [6]. Изучение сложных объектов современными методами хроматомасс-спектрометрии порождает именно такое количество информации (табл. 1).
Таблица 1.
Примеры современных больших массивов данных/информации
| Массив данных | Объем данных* |
|---|---|
| Результаты секвенирования ДНК одного человека [3] | 750 Мб |
| Геномные данные населения США [3] | 222 Пб |
| Масс-спектрометрические базы данных в протеомике [3] | >1 Тб |
| Накопленные данные в метаболомике [11] | ~4 Тб |
| Химическая база данных, информация о десятках млн соединений [6] | ≥2 Тб |
| Хроматограмма с тандемными масс-спектрами ** [12] | 10–100 Мб |
| Масс-спектрометрическое изображение среза ткани, имиджинг (биообразцы) [13] | До сотен Гб |
| Библиотека 1 млн тандемных масс-спектров [14] | 1 Гб |
Примечание: Мб – мегабайт (106 байт); Гб – гигабайт (109 байт); Тб – терабайт (1012 байт); Пб – петабайт (1015 байт). *Для сравнения укажем, что “Война и мир” в оцифрованном виде – это 2 Мб [3]. **Ориентировочно запись информации об одном пике в масс-спектре (масса и интенсивность) занимает 8 байт [14].
Разнообразие (variety) данных и их сложность/многомерность. Типы информационных источников и виды информации (тексты, графики, изображения, видео и др.) разнообразны. Форматы данных, например, спектров, зарегистрированных приборами разных фирм, различаются. В целом стандартные форматы для спектральной информации (масс-спектров) отсутствуют.
Быстрота (или скорость – velocity). Аналитические приборы порождают большие потоки данных, что, в свою очередь, требует быстрой их обработки, согласованного учета справочной информации и своевременного принятия решений со стороны специалиста, участвующего в проведении анализа (получении данных).
Достоверность (veracity). Большие потоки информации могут включать данные плохого качества. Они связаны со слабыми аналитическими сигналами (малые отношения сигнал/шум), выбросом значений измеряемых величин, ошибочными и неполными записями в базах данных, неясными их источниками. При создании справочных спектральных баз данных необходимо контролировать качество исходной информации.
ПЕРВИЧНЫЕ АНАЛИТИЧЕСКИЕ ДАННЫЕ
Первичные данные генерируются аналитическими приборами в ходе соответствующих экспериментов. Масс-спектрометрия и ее сочетание с газовой или жидкостной хроматографией традиционно обеспечивают наибольшие объемы информации. Мерой количества данных в хроматографии (и других методах разделения) и масс-спектрометрии можно считать пиковую емкость – максимальное количество пиков на хроматограмме или в масс-спектре, оцениваемое теоретически при заданном разрешении. Оценки пиковой емкости и соответствующего быстродействия приборов [4] приведены в табл. 2. Видно, что такие варианты масс-спектрометрии, как масс-спектрометрия высокого разрешения и имиджинг, приводят к максимальным объемам данных. Массовые пики (прежде всего молекулярных ионов, протонированных или катионированных молекул) соответствуют отдельным соединениям, что обеспечивает возможности многокомпонентного (мультианалитного, мультиплексного) хромато-масс-спектрометрического анализа. Например, запись одной хроматограммы методом ВЭЖХ и сопутствующих масс-спектров высокого разрешения позволяет обнаружить более 100 тыс. пептидов [15].
Таблица 2.
Пиковая емкость и быстродействие аналитических приборов [4]
| Метод, прибор | Пиковая емкость, число пиков | Быстродействие, пик/c |
|---|---|---|
| Капиллярный электрофорез | 102–103 | 10–1 |
| ВЭЖХ/УЭЖХ | 102–103 | <1 |
| 2D гель-электрофорез | 103 | <1 |
| МС2 с тройным квадруполем | 103–104 | 104–105 |
| МС2 с ионной ловушкой | 104–105 | 103 |
| МСВР (времяпролетная МС) | 104–105 | 108–109 |
| МСВР (Орбитрэп) | 105–106 | 105 |
| МСВР (ионно-циклотронный резонанс) | 105–106 | 106 |
| ВЭЖХ–МС2 | 107–109 | 103–106 |
| Масс-спектрометрическая визуализация, имиджинг (биообразцы) | 109–1011 (пиксели) | 105–106 |
Рост быстродействия современных аналитических приборов (табл. 2, правый столбец) приводит к большим потокам данных, однако химику (биохимику) нельзя рассчитывать на симбатное увеличение полезной информации вследствие сложности и разнородности аналитических сигналов, трудности их аннотации (идентификации, интерпретации), наличия ошибок. В идеальной ситуации скорость компьютерной обработки, аннотации и интерпретации данных должна быть согласована с их первичными потоками.
Большие данные порождаются не только масс-спектрометрическими приборами, но и самыми простыми анализаторами, если они собраны в достаточно большие сети и/или обеспечивают большое количество измерений во времени/пространстве. Распространены три варианта таких аналитических систем.
1. Анализ объектов окружающей среды. Пример: проведен мониторинг загрязненности городского воздуха при использовании мобильных анализаторов (определение NO, NO2, сажи); на основе 3 млн наблюдений составлены карты загрязнений с разрешением 30 м [16].
2. Технологии, производство. Химические анализаторы/сенсоры, в том числе хроматографы и спектральные приборы, встраиваются наряду с другими датчиками в системы управления технологическими (производственными) процессами [10].
3. Медицина, здравоохранение. Быстро развивается методология слежения за состоянием здоровья людей в режиме реального времени при считывании данных носимых или имплантируемых сенсоров (детектирование глюкозы, лактатов, ионов металлов, NO и других аналитов) [8, 17, 18]. Прогресс в этой области поддерживается развитием нанотехнологий и соответствующими новыми разработками сенсорной техники [19].
АПРИОРНАЯ ХИМИЧЕСКАЯ ИНФОРМАЦИЯ
Результаты химического анализа попадают в научные публикации, справочники и другую литературу в виде описания новых соединений и данных о количественном содержании известных веществ в различных объектах/матрицах. Эта информация прямо и косвенно включается в химические базы данных (табл. 3). Они содержат те или иные сведения о структуре и свойствах химических соединений (веществ), путях их синтеза и методах анализа, применении, родственных веществах, биотестах и др.
Таблица 3.
Наиболее крупные химические базы данных
| Название | Массив соединений | Свободный доступ | Комментарии |
|---|---|---|---|
| Chemical Abstracts Service [20] | 155 млн веществ, включая индивидуальные соединения* и смеси** и 68 млн биополимеров (пептиды, белки, нуклеиновые кислоты и др.) | – | Главная ценность – полная библиографическая информация по химии и смежным наукам |
| PubChem [22] | 96 млн индивидуальных соединений*, 235 млн веществ*, ** | + | Биологически значимые соединения, биологическая активность, токсичность |
| ChemSpider [23] | 74 млн структур (индивидуальных соединений) | + | Компиляция из 262 источников с соответствующими ссылками |
| ZINC15 [24, 25] | >230 млн реактивов (химикатов), готовых к поставке | + | Сводный каталог компаний – поставщиков химической продукции |
Разные виды информации имеют различное значение для химико-аналитической работы. Аналитиков, занимающихся целевым определением (заранее заданные соединения), прежде всего интересуют методики анализа. Ссылки на них или их изложение в доступных базах данных (табл. 3) встречаются нечасто. Тем не менее, нетрудно обнаружить, что портал PubChem [22] аннотирует официальные и некоторые другие методики определения распространенных химических соединений.
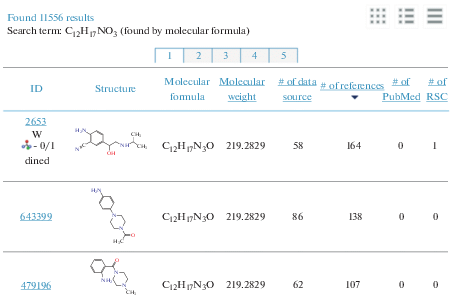
При подготовке аналитических экспериментов и истолковании их результатов химик может использовать и другую априорную информацию. Например, при разработке методик анализа часто требуются данные о точках кипения тех или иных соединений, их кислотности/основности и гидрофильности/гидрофобности. Далее, вещества, на которые часто ссылаются в литературе и базах данных (“высокоцитируемые” вещества), как правило, являются популярными/распространенными, и их следует учитывать в первую очередь, рассматривая список кандидатов на идентификацию при нецелевом анализе проб (анализе проб/образцов неизвестного состава) [25–30] (рис. 2).
Рис. 2.
Результаты поиска химических соединений с формулой C12H17N3O в базе данных ChemSpider. Результаты ранжированы по числу ссылок (# of References) на эти соединения в базе данных. Указаны три верхние позиции, на первом месте – лекарственный препарат циматерол (сimaterol).
В этой разновидности химического анализа на первый план выступает идентификация компонентов анализируемых образцов. В общем случае приходится учитывать все известные и даже возможные вещества (все химическое пространство) [25], поэтому уместен вопрос о количестве известных индивидуальных химических соединений. Что касается низкомолекулярных соединений (молекулярная масса в пределах сотен Дальтонов), понятно, что их количество исчисляется многими десятками миллионов (табл. 3). Точное же число не поддается простой оценке: в информационной системе Chemical Abstracts Service (табл. 3), наиболее авторитетной для нескольких поколений химиков, индивидуальные соединения и их смеси (например, смеси изомеров, в том числе рацематы) попадают в один и тот же регистр. Число различных биополимеров, например белков, также не поддается точной оценке. Тем не менее, отмечено большое разнообразие белков в организмах различных видов (>10 млн [31]).
При нецелевом анализе очень важны справочные спектральные данные. Они в наиболее полном объеме представлены в специализированных информационных системах, распространенным примером которых являются электронные библиотеки масс-спектров (табл. 4). Их полнота зависит от природы и свойств аналитов. Исторически первыми были библиотеки масс-спектров электронной ионизации, зарегистрированных для летучих соединений. К настоящему времени в этой области накоплены наиболее полные массивы масс-спектрометрической информации (дополненной хроматографическими характеристиками).
Таблица 4.
Большие библиотеки масс-спектров
| Название | Объем | Комментарии | |
|---|---|---|---|
| спектры | соединения | ||
| Низкомолекулярные соединения | |||
| Wiley Registry 11th, ЭИ-МС1 [32] | 775 тыс. | 599.7 тыс. | Летучие соединения |
| Wiley, 11 специализированных библиотек, ЭИ-МС1 [32] | >88 тыс. | Летучие лекарственные соединения, пестициды, биологически активные соединения, др. | |
| Wiley МС2 [32] | >17 тыс. | ≤6.2 тыс. | Лекарственные соединения |
| NIST 17, ЭИ-MС1 [32] | 306 622 | 267376 | Летучие соединения |
| NIST 17, MС2 [32] | 652 475 | ≤28 тыс. | |
| METLIN [32, 33] экспериментальные спектры in silico спектры | >72 тыс. >699.5 тыс. |
>14 тыс. >233 тыс. |
MС2, преобладают метаболиты |
| The Global Natural Product Social Molecular Networking (GNPS) [34] | 212 230 | 12 694 | MС2, природные соединения |
| MassBank of North America (MONA) [35] | >210 тыс. | 75 270 | Преобладают МС2 спектры биологически активных соединений. Присутствуют in silico масс-спектры |
| MassBank [36] | 50 998 | 16199 | MC1, MCn, биологически активные соединения |
| Spectral Database for Organic Compounds [37] | ~25 тыс. | ЭИ-MС1, летучие соединения | |
| HighChem Spectral Tree [38] | >13 тыс. | MСn, лекарственные соединения, метаболиты | |
| Пептиды, MС2 | |||
| PeptideAtlas [39] | 43.4 млн | 253 690 | 2012 г. |
| X!Hunter [40] | 20.7 млн | 6.0 млн | Оценка для 2014 г. |
| PRIDE [41] | 20.7 млн | 2013 г. | |
| NIST peptide [42] | >4.3 млн | <1.26 млн | |
Биологически активные соединения чаще всего являются нелетучими. Для получения их масс-спектров обычно применяют разработанный позднее метод – тандемную масс-спектрометрию с электрораспылением. Сегодня библиотеки нелетучих соединений недостаточно полны (табл. 4), но число охватываемых соединений постоянно растет. Появляются массивы теоретических (in silico) тандемных масс-спектров, хотя их предсказание не очень надежно.
Библиотеки масс-спектров биополимеров наиболее полно представлены в случае пептидов (табл. 4). Количество масс-спектров в них – наибольшее для обсуждаемых массивов информации. Тем не менее, и в этом случае имеется лишь малая доля необходимых данных; теоретически допустим синтез гораздо большего числа соединений данного класса.
В целом надежные справочные данные представлены для подавляющего меньшинства известных (био)химических соединений. Этот факт дополняет вывод о том, что химический анализ, по-существу, еще не касался большей части известного химического пространства [4].
ОБРАБОТКА И АНАЛИЗ ДАННЫХ
Большие данные, например хроматограммы в сочетании с тандемными масс-спектрами высокого разрешения или результаты спектральной визуализации, даже после начальной аннотации аналитических сигналов (им приписывают времена удерживания и массы ионов) представляют собой лишь “сырье”. Дальнейшая его обработка – в тривиальном случае сравнение со справочными данными (популярная процедура идентификации [29, 30, 43, 44]) и манипуляция интенсивностями сигналов (количественное определение) – приводит к требуемым результатам химического анализа. Методы многомерной статистики (хемометрии) способствуют получению этих и других результатов, касающихся природы определяемого вещества и родственных проблем. Хемометрия больших данных подробно рассмотрена в обзорах [5, 6], ниже укажем лишь отдельные примеры ее применения.
Для анализа сложных объектов типично наложение хроматографических или спектральных сигналов различных компонентов смесей. В такой ситуации используют программные подходы, основанные на многомерном разрешении сигналов (multivariate resolution) [6, 45]. При необходимости количественного определения проводят многомерную градуировку [6, 45].
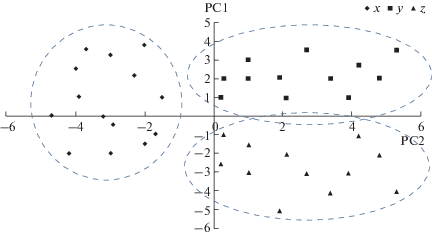
Хемометрия нашла широкое применение при решении задач качественного анализа II [30] – для группировки, типизации, классификации, идентификации анализируемых проб по спектральным, хроматографическим и другим характеристикам методами кластерного анализа и родственными методами [5, 6]. При этом часто сокращают число переменных и выделяют наиболее важные из них (метод главных компонент [46], рис. 3). Параллельно находят свое решение задачи визуализации (наглядного изображения) данных. Здесь учитываются особенности восприятия информации человеком, которому сложно охватить и осмыслить большие многомерные данные, если они не представлены “малоразмерными проекциями” (lower-dimensional projections) [4].
Методы статистики/хемометрии пригодны и для анализа информации в больших базах данных. Перспективно использование программной методологии text mining (интеллектуальный анализ текста), направленной на выявление в научной литературе (научных базах данных) неизвестных связей, зависимостей, закономерностей, которые можно использовать в дальнейших исследованиях [6, 47]. В качестве одного из примеров отметим работу [48], предлагающую способ извлечения аналитических методик из научных текстов в формализованном виде. Методы text mining полезны для выявления тенденций развития научных и технологических областей. Так, был проведен анализ большого количества патентов в области фармацевтики. Это исследование привело к выводу о том, что химики-синтетики перестали заботиться об увеличении выхода реакций синтеза лекарственных соединений, получив в свое распоряжение такой эффективный метод их выделения и очистки как препаративная хроматография [49].
Если отсутствует компьютерная программа необходимой манипуляции большими данными, возможна оценка их общих характеристик путем формирования небольших случайных выборок из начального массива. В нескольких работах мы использовали такой подход, например, для определения правильности результатов поисков в новых библиотеках масс-спектров [50, 51]. При всей простоте этого подхода, однако, возникает статистическая погрешность, связанная с ограниченным размером выборки. В табл. 5 даны примеры такой погрешности при оценке доли ПП в ходе поисков в соответствующих библиотеках масс-спектров. В этом случае предварительно формируется случайная выборка из этого же массива информации как подмассив спектров “неизвестных” соединений [50, 51]. Данные табл. 5 показывают, что при одной и той же правильности результатов погрешность зависит в наибольшей степени от размера выборки, а не от исходного массива спектров.
Таблица 5.
Погрешность оценки правильности идентификации при поисках в спектральной библиотеке (вычисления с калькулятором на сайте [52])
| Количество спектров в библиотеке | Выборка спектров | Заданная доля ПП*, % | Доверительный интервал, ± % |
|---|---|---|---|
| 100 000 | 1000 | 50 | 3.1 |
| 95 | 1.3 | ||
| 100 | 50 | 9.8 | |
| 95 | 4.3 | ||
| 10 000 | 1000 | 50 | 2.9 |
| 95 | 1.3 | ||
| 100 | 50 | 9.8 | |
| 95 | 4.3 |
КОМПЬЮТЕРЫ, ПРОГРАММЫ, СЕТИ
Постоянное увеличение производительности компьютеров и размера памяти запоминающих устройств способствует появлению больших данных [4]. Темпы роста памяти значительнее; отсюда следует, что, учитывая также быстродействие аналитических приборов (см. выше), накопление данных может опережать возможности их обработки, интерпретации, осмысления [4, 5]. Логично считать, что лимитирующей стадией развития химического анализа, связанного с большими данными, является разработка новых математических подходов и новых алгоритмов работы с данными, их программная реализация, а также стандартизация в области информатики. Проблемы компьютерной манипуляции большими данными и детали соответствующего программного обеспечения освещены в многочисленных публикациях (см. обзоры [6, 8, 53, 54]).
Развитие широкополосного интернета (высокая скорость передачи информации) [4] обеспечило работе с большими данными коллективный, взаимосвязанный, межлабораторный характер. Резко увеличивается масштаб обмена данными и сотрудничество при их обработке, для этого создаются специальные компьютерно-программные платформы (data sharing platforms [3], e-collaboration platforms [7]). Решение ряда задач, в том числе химической (биохимической) аналитики, проводится совместно представителями разных лабораторий (crowdsourcing – сотрудничество через интернет больших групп специалистов). Можно отметить, например, совместное создание масс-спектрометрических баз данных в области природных соединений [34] или кооперацию при истолковании хроматомасс-спектрометрических данных, полученных в одной из лабораторий [4]. Еще один пример работ в сети – облачные вычисления (cloud computing). Они реализуются при сетевом доступе к общим или посторонним вычислительным ресурсам – серверам, устройствам хранения данных, суперкомпьютерам. Известны примеры подобной обработки многомерных масс-спектрометрических изображений и протеомных данных [4].
АКТУАЛЬНЫЕ ПРИМЕРЫ КАЧЕСТВЕННОГО И КОЛИЧЕСТВЕННОГО АНАЛИЗА
Охарактеризованные выше методы и приборы, позволяющие генерировать данные большого (относительно большого) размера, и соответствующие информационные ресурсы широко применяются в мультианалитных качественных и количественных определениях (био)органических соединений многих классов в разных матрицах.
Прежде всего, это относится к разнообразным идентификационным процедурам. Для лучшего понимания их перспектив необходимо соответствующее сравнение в межлабораторных экспериментах, до последнего времени проводившихся преимущественно в отношении количественного анализа. Начиная с 2012 г., реализована серия сравнительных экспериментов по распознаванию низкомолекулярных соединений различными методами в разных лабораториях (critical assessment of small molecule identification, CASMI) [55, 56]. Здесь впервые широко оценены возможности предсказания тандемных масс-спектров. Эффективность предсказания для более 200 аналитов оказалась не очень высокой: при использовании in silico масс-спектров в качестве справочных данных получено приблизительно 30% ПП [55]. Объединение этих результатов с традиционным поиском в библиотеках масс-спектров экспериментального происхождения и поиском высокоцитируемых соединений в химических базах данных увеличило показатель ПП до 70% [55] и даже 87–93% [56]. Эти характеристики эффективности распознавания молекул в случае совокупности методов следует считать достаточно высокими, поскольку ПП – основной метрологический показатель качественного анализа – для классического/эталонного варианта масс-спектрометрии (ЭИ) и соответствующих больших библиотек масс-спектров составляет приблизительно 80% [29, 30].
Работа [57], рассматривающая обнаружение более 9000 метаболитов, которые присутствуют в моче человека, демонстрирует современные возможности и ограничения быстрой идентификации многих компонентов сложных биопроб в рамках их нецелевого анализа. Ограничения могут быть связаны, например, с неполнотой библиотек масс-спектров и недоступностью многих аналитических стандартов. Лишь 175 соединений идентифицировано с максимальной надежностью: при использовании стандартных образцов метаболитов с учетом их молекулярных масс, хроматографических характеристик и тандемных масс-спектров. Еще 578 аналитов идентифицированы относительно надежно методом тандемной масс-спектрометрии высокого разрешения (МС2ВР) при использовании справочных библиотек [57].
Наличие многопиковых хроматограмм и связанных с ними масс-спектров позволяет проводить целевое скрининговое (обнаружение и идентификация вредных веществ, соотнесенное с нормативными уровнями их содержания) или полностью количественное определение многих аналитов в одной пробе. Это важно для аналитического экспресс-контроля продуктов питания, объектов окружающей среды и других объектов. Все чаще появляются сообщения о таких мультианалитных определениях в отношении десятков и сотен компонентов. Описано, например, определение в продуктах питания 143 [58] и 160 [59] лекарственных соединений, 295 бактериальных и грибковых метаболитов [60], 389 [61] и свыше 625 [62] загрязнителей пищи различных классов, 475 запрещенных пищевых добавок [63]. Рассмотрен анализ осадков сточных вод с определением 148 лекарственных соединений, в том числе запрещенных к применению [64]. Часть предложенных методик относится к скрининговым [58, 59, 62, 63], в других работах [60, 61, 64] описан полноценный количественный анализ. Определения во многих случаях относятся к следам аналитов (содержание 1–100 мкг/кг). Использованные методы скрининга, как правило, основаны на использовании ВЭЖХ (УЭЖХ) и МСВР (МС2ВР) и могут быть распространены на нецелевой анализ.
Характеристики количественного мультианалитного определения, чаще всего базирующегося на применении МС2 (МС2ВР), неодинаковы для разных аналитов даже при оптимизации условий анализа. Это показывают, например, результаты одновременного определения более трехсот метаболитов в четырех пищевых матрицах [60]. Из 331 целевых аналитов 36 соединений (11%) не были определены. Одна из причин этого – невысокая чувствительность метода по отношению к некоторым компонентам проб, ослабленная к тому же матричными эффектами. Требуемая нормативными документами степень извлечения различных аналитов из исходных образцов (70–120%) реализована лишь для 21–74% соединений (при их содержании в анализируемых образцах в интервале 0.3–630 мкг/кг). Что касается погрешностей количественного определения, выраженных в относительном стандартом отклонении средних значений, для большинства аналитов этот показатель не превышал границы 20%, но зависел от вида матрицы [60].
Таким образом, в “эру больших данных” неизбежно стремление к увеличению числа соединений, одновременно определяемых в одних и тех же пробах, что приводит, несомненно, к более полному обнаружению отдельных компонентов, но необязательно сопровождается хорошими показателями правильности идентификации и характеристиками количественного определения всех аналитов.
* * *
Тема данной статьи – большие данные – не отражает существование какой-либо самостоятельной науки или ее раздела. Скорее, это часть современной аналитики, объединяющая ее с биологией, информатикой и статистикой, обусловливающая модернизацию образования в химии [3, 9]. Проблемы, связанные с большими данными, возникают в химическом анализе, если работа лаборатории не ограничивается определением одного или небольшого числа аналитов традиционными методами. Постоянный нецелевой анализ биологических проб методами хроматографии и масс-спектрометрии (особенно МС2ВР) неизбежно затрагивает манипуляции с большими данными. Они появляются в ходе мультианалитных анализов, проводимых с целью большей полноты обнаружения отдельных компонентов сложных проб. Это, однако, не обязательно сопровождается высокими показателями правильности идентификации и хорошими характеристиками количественного определения отдельных аналитов. Применение сенсорных устройств и других анализаторов (в мультисигнальных, мультианалитных вариантах) также может быть сопряжено с получением больших данных. Использование априорной информации, представленной в больших химических базах данных, полезно для химиков всех специализаций. Аналитикам, тем не менее, следует учесть, что доступные сведения о способах, методах и методиках анализа могут быть фрагментарными. Использование больших массивов информации особенно важно при решении задач нецелевого анализа. Отбор кандидатов на идентификацию связан с особыми физико-химическими свойствами химических веществ и их популярностью (высокой цитируемостью), надежная идентификация предполагает доступ к справочным библиотекам спектров (масс-спектров). Методы обработки, анализа и представления данных, прежде всего методы хемометрии и визуализации, прогрессируют вместе с ростом количества и сложности информации. Полный анализ данных может быть заменен выборочным. Технические характеристики компьютеров и их сетей растут опережающими темпами, создавая потенциал развития интеллектуальных методов анализа информации. Прогресс интернета обеспечивает новые возможности использования посторонних вычислительных/информационных ресурсов, кооперации при создании электронных справочных данных и онлайнового межлабораторного сотрудничества в решении аналитических задач, первоначально затрагиваемых одну из лабораторий.
Список литературы
Eckschlager K., Danzer K. Information Theory in Analytical Chemistry. N.Y.: Wiley, 1994. 275 p.
Харрис Д. Данные, информация и управление знаниями, 2011. https://www.prj-exp.ru/dwh/data_information_knowledge.php (01.10.2018).
Williams A.J., Pence H.E. The future of chemical information is now // Chem. Int. 2017. V. 39. № 3. P. 9.
May J.C., McLean J.A. Advanced multidimensional separations in mass spectrometry: Navigating the big data deluge // Annu. Rev. Anal. Chem. 2016. V. 9. P. 387.
Szymańska E. Modern data science for analytical chemical data – A comprehensive review // Anal. Chim. Acta. 2018. V. 1028. P. 1.
Tauler R., Parastar H. Big (bio) chemical data mining using chemometric methods: A need for chemists // Angew. Chem. Int. Ed. Engl. 2018. March 23. https://doi.org/10.1002/anie.201801134
Kalidindi S.R., De Graef M. Materials data science: Current status and future outlook // Annu. Rev. Mater. Res. 2015. V. 45. P. 171.
Andreu-Perez J., Poon C.C., Merrifield R.D., Wong S.T., Yang G.Z. Big data for health // IEEE J. Biomed. Health Inform. 2015. V. № 4. P. 1193.
Pence H.E., Williams A.J. Big data and chemical education // J. Chem. Educ. 2016. V. 93. № 3. P. 504.
Chiang L., Lu B., Castillo I. Big data analytics in chemical engineering // Annu. Rev. Chem. Biomol. Eng. 2017. V. 8. P. 63.
Haug K., Salek R.M., Steinbeck C. Global open data management in metabolomics // Curr. Opin. Chem. Biol. 2017. V. 36. P. 58.
Pluskal T., Yanagida M. Metabolomic analysis of Schizosaccharomyces pombe: sample preparation, detection, and data interpretation // Cold Spring Harbor Protocols. 2016. V. 2016. № 12. P. 1044.
Veselkov K., Sleeman J., Claude E., Vissers J.P., Galea D., Mroz A., Laponogov I., Towers M., Tong R., Mirnezami R., Takats Z., Nicholson J., Langridge J.I. BASIS: High-performance bioinformatics platform for processing of large-scale mass spectrometry imaging data in chemically augmented histology // Sci. Rep. 2018. V. 8. № 1. P. 4053.
Wright D. A. Automatic reconstruction of MS-2 spectra from all ions fragmentation to recognize previously detected compounds. U.S. Patent Appl. No. 13/682,443, 20.11.2012. Pub. No. US 2014/0142865 A1, 22.5 2014.
Michalski A., Cox J., Mann M. More than 100,000 detectable peptide species elute in single shotgun proteomics runs but the majority is inaccessible to data-dependent LC–MS/MS // J. Proteome Res. 2011. V. 10. № 4. P. 1785.
Apte J.S., Messier K.P., Gani S., Brauer M., Kirchstetter T.W., Lunden M.M., Marshall J.D., Portier C.J., Vermeulen R.C.H., Hamburg S.P. High-resolution air pollution mapping with Google street view cars: exploiting big data // Environ. Sci. Technol. 2017. V. 51. № 12. P. 6999.
Bandodkar A.J., Jeerapan I., Wang J. Wearable chemical sensors: Present challenges and future prospects // ACS Sensors. 2016. V. 1. № 5. P. 464.
Koydemir H.C., Ozcan A. Wearable and implantable sensors for biomedical applications // Annu. Rev. Anal. Chem. 2018. V. 11. № 1. P. 127.
Мильман Б.Л., Журкович И.К. Новые рапорты с фронтов науки: наноматериалы, микрофлюидика, протеомика // Аналитика. 2017. № 5. С. 30.
CAS content. https://www.cas.org/about/cas-content (31.07.2019).
Substance identity in REACH. EU Final Report, 2016. https://www.cas.org/about/cas-content (02.10.2018).
PubChem. https://pubchem.ncbi.nlm.nih.gov/search (31.07.2019).
ChemSpider. http://www.chemspider.com (31.07.2019).
ZINC15. http://zinc15.docking.org (31.07.2019).
Milman B.L., Zhurkovich I.K. The chemical space for non-target analysis // Trends Anal. Chem. 2017. V. 97. P. 179.
Milman B.L., Kovrizhnych M.A. Identification of chemical substances by testing and screening of hypotheses II. Determination of impurities in n-hexane and naphthalene // Fresenius’ J. Anal. Chem. 2000. V. 367. № 7. P. 629.
Milman B.L. A procedure for decreasing uncertainty in the identification of chemical compounds based on their literature citation and cocitation. Two case studies // Anal. Chem. 2002. V. 74. № 7. P. 1484.
Milman B.L. Literature-based generation of hypotheses on chemical composition using database co-occurrence of chemical compounds // J. Chem. Inf. Model. 2005. V. 45. № 5. P. 1153.
Мильман Б.Л. Введение в химическую идентификацию. СПб: ВВМ, 2008. 180 с.
Milman B.L. Chemical identification and its quality assurance. Berlin: Springer, 2011. 281 p.
How many proteins exist in human body? http://www.innovateus.net/health/how-many-proteins-exist-human-body (02.10.2018).
Mass spectral libraries (NIST 17 and Wiley libraries). https://www.sisweb.com/software/ms/wiley.htm (02.10.2018).
Guijas C., Montenegro-Burke J.R., Domingo-Almenara X., Palermo A., Warth B., Hermann G., Koellensperger G., Huan T., Uritboonthai W., Aisporna A.E., Wolan D.W., Spilker M.E., Benton H.P., Siuzdak G. METLIN: a technology platform for identifying knowns and unknowns // Anal. Chem. 2018. V. 90. № 5. P. 3156.
The Global Natural Product Social Molecular Networking (GNPS). https://gnps.ucsd.edu/ProteoSAFe/gnpslibrary.jsp?library=all (03.10.2018).
MONA − MassBank of North America. http://mona.fiehnlab.ucdavis.edu (17.11.2018).
MassBank. https://massbank.eu/MassBank (03.10.2018).
Spectral Database for Organic Compounds. https://sdbs.db.aist.go.jp/sdbs/cgi-bin/cre_index.cgi (17.11.2018).
HighChem Spectral Tree. http://www.highchem.com/index.php/81-massfrontier (17.11.2018).
PeptideAtlas overview. http://www.peptideatlas.org/overview.php (3.10.2018).
X!HUNTER Annotated Spectrum Library. http://thegpm.org/HUNTER/index.html (03.10.2018).
Griss J., Foster J.M., Hermjakob H., Vizcaíno J.A. PRIDE Cluster: Building a consensus of proteomics data // Nat. Methods. 2013. V. 10. № 2. P. 95.
NIST Libraries of Peptide Tandem Mass Spectra. https://chemdata.nist.gov/dokuwiki/doku.php?id= peptidew:start (17.11.2018).
Kind T., Tsugawa H., Cajka T., Ma Y., Lai Z., Meh-ta S.S., Wohlgemuth G., Barupal D.K., Showalter M.R., Arita M., Fiehn O. Identification of small molecules using accurate mass MS/MS search // Mass Spectrom. Rev. 2017. V. 37. № 4. P. 513.
Blaženović I., Kind T., Ji J., Fiehn O. Software tools and approaches for compound identification of LC−MS/MS data in metabolomics // Metabolites. 2018. V. 8. № 2. P. 31.
Dumancas G.G., Bello G.A., Hughes J., Murimi R., Viswanath L.C., Orndorff C.O., Dumancas G.F., O’Dell J.D. Visualization tools for big data analytics in quantitative chemical analysis: A tutorial in chemometrics / Handbook of Research on Big Data Storage and Visualization Techniques / Eds. Segall R., Cook J. Hershey, PA: IGI Global, 2018. P. 873. https://doi.org/10.4018/978-1-5225-3142-5.ch030
Дубров А.М., Мхитарян В.С., Трошин Л.И. Многомерные статистические методы. М.: Финансы и статистика, 1998. 352 с.
Krallinger M., Rabal O., Lourenco A., Oyarzabal J., Valencia A. Information retrieval and text mining technologies for chemistry // Chem. Rev. 2017. V. 117. № 12. P. 7673.
Postma G.J., Kateman G. A systematic representation of analytical chemical actions // J. Chem. Inf. Comput. Sci. 1993. V. 33. № 3. P. 350.
Schneider N., Lowe D. M., Sayle R.A., Tarselli M.A., Landrum G.A. Big data from pharmaceutical patents: a computational analysis of medicinal chemists’ bread and butter // J. Med. Chem. 2016. V. 59. № 9. P. 4385.
Мильман Б.Л., Гостев В.В., Дмитриев А.В. Сравнение “низкомолекулярного” и устоявшегося подходов к видовой идентификации бактерий методом масс-спектрометрии МАЛДИ // Масс-спектрометрия. 2016. Т. 13. № 4. С. 252.
Milman B.L., Zhurkovich I.K. Identification performance of low-molecular compounds by searching tandem mass spectral libraries with simple peak matching // Mass Spectrom. Lett. 2018. V. 9. № 3. P. 73.
Sample size calculator. https://www.surveysystem.com/sscalc.htm (03.10.2018).
Назипова Н.Н., Исаев Е.А., Корнилов В.В., Первухин Д.В., Морозова А.А., Горбунов А.А., Устинин М.Н. Большие данные в биоинформатике // Математическая биология и биоинформатика. 2017. Т. 12. № 1. С. 102.
Alyass A., Turcotte M., Meyre D. From big data analysis to personalized medicine for all: challenges and opportunities // BMC Med. Genomics. 2015. V. 8. № 1. P. 33.
Schymanski E.L., Ruttkies C., Krauss M., Brouard C., Kind T., Dührkop K., Allen F., Vaniya A., Verdegem D., S. Böcker, Rousu J., Shen H., Tsugawa H., Sajed T., Fiehn O., Ghesquiére B., Neumann S. Critical assessment of small molecule identification 2016: automated methods // J. Cheminform. 2017. V. 9. P. 22.
Blaźenović I., Kind T., Torbašinović H., Obrenović S., Mehta S.S., Tsugawa H., Wermuth T., Schauer N., Jahn M., Biedendieck R., Jahn D., Fiehn O. Comprehensive comparison of in silico MS/MS fragmentation tools of the CASMI contest: database boosting is needed to achieve 93% accuracy // J. Cheminform. 2017. V. 9. P. 32.
Blaženović I., Kind T., Sa M.R., Ji J., Vaniya A., Wancewicz B., Roberts B.S., Torbašinović H., Lee T., Mehta S.S., Showalter M.R. Song H., Kwok J., Jahn D., Kim J., Fiehn O. Structure annotation of all mass spectra in untargeted metabolomics // Anal. Chem. 2019. V. 91. № 3. P. 2155. https://doi.org/10.1021/acs.analchem.8b04698
Dasenaki M.E., Bletsou A.A., Koulis G.A., Thomaidis N.S. Qualitative multiresidue screening method for 143 veterinary drugs and pharmaceuticals in milk and fish tissue using liquid chromatography quadrupole-time-of-flight mass spectrometry // J. Agric. Food Chem. 2015. V. 63. № 18. P. 4493.
Robert C., Gillard N., Brasseur P.Y., Pierret G., Ralet N., Dubois M., Delahaut, P. Rapid multi-residue and multi-class qualitative screening for veterinary drugs in foods of animal origin by UHPLC−MS/MS // Food Addit. Contam. A. 2013. V. 30. № 3. P. 443.
Malachová A., Sulyok M., Beltrán E., Berthiller F., Krska R. Optimization and validation of a quantitative liquid chromatography–tandem mass spectrometric method covering 295 bacterial and fungal metabolites including all regulated mycotoxins in four model food matrices // J. Chromatogr. A. 2014. V. 1362. P. 145.
Dzuman Z., Zachariasova M., Veprikova Z., Godula M., Hajslova J. Multi-analyte high performance liquid chromatography coupled to high resolution tandem mass spectrometry method for control of pesticide residues, mycotoxins, and pyrrolizidine alkaloids // Anal. Chim. Acta. 2015. V. 863. P. 29.
Pérez-Ortega P., Lara-Ortega F.J., García-Reyes J.F., Gilbert-López B., Trojanowicz M., Molina-Díaz A. A feasibility study of UHPLC-HRMS accurate-mass screening methods for multiclass testing of organic contaminants in food // Talanta. 2016. V. 160. P. 704.
Fu Y., Zhou Z., Kong H., Lu X., Zhao X., Chen Y., Chen J., Wu Z., Xu Z., Zhao C., Xu G. Nontargeted screening method for illegal additives based on ultrahigh-performance liquid chromatography–high-resolution mass spectrometry // Anal. Chem. 2016. V. 88. № 17. P. 8870.
Gago-Ferrero P., Borova V., Dasenaki M.E., Thomaidis N.S. Simultaneous determination of 148 pharmaceuticals and illicit drugs in sewage sludge based on ultrasound-assisted extraction and liquid chromatography–tandem mass spectrometry // Anal. Bioanal. Chem. 2015. V. 407. № 15. P. 4287.
Дополнительные материалы отсутствуют.
Инструменты
Журнал аналитической химии