Вестник Военного инновационного технополиса «ЭРА», 2023, T. 4, № 3, стр. 249-255
Связь гликозилирования белков S-слоя штаммов рода Lactobacillus с характерным мотивом в домене самосборки
С. В. Рыков 1, *, А. А. Войтенок 1, 2, А. И. Селимзянова 1, 3, О. В. Березина 1
1 Национальный исследовательский центр “Курчатовский институт”
Москва, Россия
2 МИРЭА – Российский технологический университет
Москва, Россия
3 Московский физико-технический институт (национальный исследовательский университет)
Москва, Россия
* E-mail: enterru@yandex.ru
Поступила в редакцию 23.10.2023
После доработки 27.10.2023
Принята к публикации 27.10.2023
Аннотация
Белки, формирующие поверхностный слой (S-слой) штаммов Lactobacillus brevis ATCC 367, ATCC 8287 и ATCC 14869, проверены на наличие гликозилирования. Впервые обнаружено гликозилирование белка S-слоя штамма ATCC 367. В составе поверхностного S-слоя штамма ATCC 367 имеются две формы белка SlpE, основная из которых с массой 56 кДа гликозилирована. Аминокислотная последовательность белка SlpE содержит мотив SASSAS, необходимый для гликозилированния и присутствующий у известных гликозилированных белков S-слоя лактобацилл. У двух других штаммов L. brevis гликозилирования белков S-слоя не обнаружено, а их последовательности не содержат характерного мотива.
ВВЕДЕНИЕ
Лактобактерии – грамположительные облигатные или факультативные анаэробные бактерии, роль которых в жизни человека трудно переоценить. Многие виды лактобактерий издревле используются в качестве заквасок и являются эффективными пробиотиками. Они неинвазивны, непатогенны и способны размножаться в организмах человека и животных, формируя нормальную сбалансированную микрофлору. Прикрепляясь к слизистым оболочкам, лактобактерии исключают или уменьшают адгезию патогенных микроорганизмов [1, 2]. Некоторые виды лактобактерий являются важной составляющей микрофлоры верхних дыхательных путей человека, могут ингибировать рост патогенов, вызывающих синусит [3, 4], и облегчают аллергические заболевания, например симптомы круглогодичного аллергического ринита [5].
Известно, что лактобактерии могут взаимодействовать с иммунными клетками слизистой оболочки или эпителиальными клетками, выстилающими слизистую оболочку, модулируя специфические функции иммунной системы [2]. У лактобактерий было выявлено несколько факторов, влияющих на иммунный ответ in vitro и in vivo, включая гликаны клеточной поверхности, ферменты, модифицирующие структуру липотейхоевых кислот и метаболиты [1, 2].
Лактобактерии повышают иммуногенность перорально вводимых вакцин, таких как ротавирусная [6], полиомиелитная [7], холерная [8] и вакцина против вируса гриппа [9]. Некоторые штаммы лактобацилл стимулируют выработку секреторных и сывороточных IgA-антител [10]. Адъювантное и иммуномодулирующее действие лактобактерий на иммунитет также очевидно из многих исследований, в которых молочнокислые бактерии использовались в качестве средств доставки вакцины для иммунизации слизистых оболочек [11, 12].
Поверхностный белковый слой (S-слой) лактобактерий – упорядоченная ячеистая белковая структура, состоящая из белков с малой молекулярной массой (25–71 кДа), обладающих уникальной способностью к самосборке на поверхности клеток [13]. S-слой не только способен к самосборке на растущей клеточной поверхности, но и может быть реконструирован на поверхности клеток того же или другого вида бактерий, с которого он был ранее удален. Гетерологичная реконструкция S-слоя возможна в случае совпадения механизмов взаимодействия между связывающими доменами Slp-белков и компонентами клеточной стенки у донорного и акцепторного штаммов. При этом механизмы адсорбции и состав полисахаридных рецепторов белков S-слоя у разных штаммов лактобацилл и лактококков могут различаться [14, 15].
Некоторые белки S-слоя лактобактерий, а также другие поверхностные белки, прикрепленные к клеточной стенке, гликозилированы [16, 17]. Гликановые структуры, связанные с Slp-белками, могут связываться с рецепторами распознавания образов и таким образом участвовать в активации иммунного ответа, поэтому исследование структурных и биохимических особенностей гликанов S-слоя, а также молекулярных механизмов гликозилирования белков S-слоя является фундаментальной основой для разработки нового инструментария для молекулярного конструирования на основе S-слоя.
Лактобактерии Lactobacillus brevis – перспективная платформа для экспонирования модифицированных белков на поверхности клеток с перспективой создания новых живых вакцин [13]. Цель настоящей работы – поиск гликозилированных белков S-слоя у штаммов L. brevis.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Штаммы лактобацилл Lactobacillus brevis В-КПМ В-5563 (=ATCC 367), L. brevis ВКПМ В‑10 405 (=ATCC 8287) и L. brevis ВКПМ В-10 903 (=ATCC 14 869) получены в Национальном биоресурсном центре “Всероссийская коллекция промышленных микроорганизмов” НИЦ “Курчатовский институт”.
Культуры штаммов лактобацилл выращивали на среде MRS (HiMedia Laboratories) при температуре 28°С без качания в течение 1–2 сут.
Белки S-слоя экстрагировали с помощью додецилсульфата натрия. Клетки штаммов лактобактерий осаждали из жидкой культуры центрифугированием на 3000 об./мин. Осадок ресуспендировали в растворе, содержащем 2% додецилсульфата натрия и 1 мМ меркаптоэтанола. Суспензию инкубировали при 70°С в течение 10 мин, после чего клетки собирали центрифугированием при 13 000 об./мин и анализировали супернатант на присутствие белков S-слоя.
Препараты белков S-слоя анализировали методом электрофореза в 12%-ном полиакриламидном геле (ПААГ) по Лэммли в денатурирующих условиях.
Идентификацию белков S-слоя проводили по масс-спектрометрическим пептидным картам специфического протеолитического гидролиза. Трипсинолиз проводили во фрагментах геля, содержащих полосы целевых белков. Масс-спектры регистрировали на времяпролетном MALDI масс-спектрометре BRUKER Ultraflex TOF/TOF MS (Bruker Daltonics Gmbh, Германия) в режиме положительных ионов. Регистрацию MALDI масс-спектров осуществляли в ЦКП Научно-исследовательского института биомедицинской химии имени В.Н. Ореховича. Анализ полученных спектров проводили с помощью программного обеспечения FlexAnalysis 3.0 (Bruker Daltonics Gmbh, Германия). Идентификацию белков по спектру проводили с помощью базы Mascot Server (Matrix Science).
Анализ гликозилирования белков S-слоя штаммов лактобацилл проводили методом окрашивания гликопротеинов в реакции с перйодной кислотой и основанием Шиффа с помощью набора Pierce Glycoprotein Staining Kit (№ 24562, Thermo Scientific) в соответствии с инструкцией производителя.
Последовательности полных геномов штаммов L. brevis ATCC 367, ATCC 8287 и ATCC 14869 получены из базы данных GenBank. Модели пространственных структур белков S-слоя L. brevis, сконструированные с помощью алгоритма глубокого машинного обучения AlphaFold2, получены из базы данных UniProt. Выравнивание аминокислотных последовательностей и филогенетический анализ выполняли в программе ClustalX версии 2.1.
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
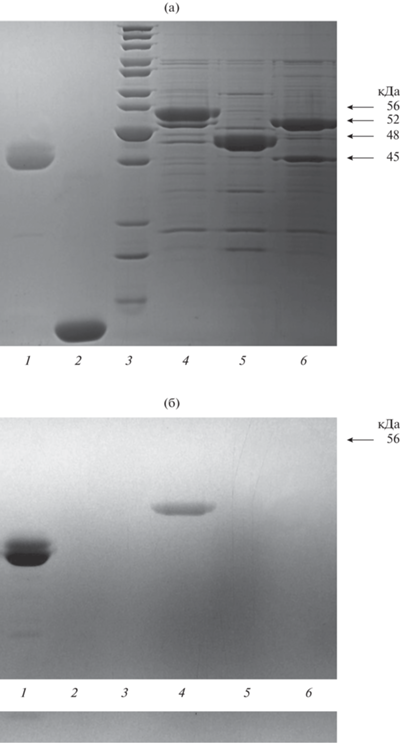
Препараты белков S-слоя были получены из клеток штаммов L. brevis ВКПМ В-5563 (ATCC 367), L. brevis ВКПМ В-10405 (ATCC 8287) и L. brevis ВКПМ В-10903 (ATCC 14869), предоставленных коллекцией ВКПМ. С помощью электрофореза в денатурирующем ПААГ экспериментально подтверждено наличие поверхностных белков в данных штаммах. Сравнительный анализ препаратов белков S-слоя показал значительное различие их состава у разных штаммов одного вида L. brevis. Так, на электрофореграмме препарата штамма В-5563 обнаружены полосы размером 48, 52 и 56 кДа, тогда как препарату штамма В-10903 соответствуют белковые полосы размером 45 и 52 кДа, а препарату штамма В‑10405 – только одна полоса размером 48 кДа (рис. 1а).
Рис. 1.
Электрофореграмма экстрактов белков поверхностного S-слоя клеток штаммов L. brevis: а – окрашивание красителем Кумасси голубой, б – окрашивание перйодной кислотой и основанием Шиффа. Номера дорожек: белок пероксидазы хрена, положительный контроль на гликозилирование (1), белок соевого ингибитора трипсина, отрицательный контроль на гликозилирование (2), маркер PageRuler Unstained Protein Ladder (Thermo Scientific) (3), экстракты белков поверхностного S-слоя клеток штаммов Lactobacillus brevis ATCC 367 (4), ATCC 8287 (5) и ATCC 14 869 (6).
Анализ геномов L. brevis ATCC 367, ATCC 8287 и ATCC 14869 и сопоставление с результатами [13] позволили идентифицировать гены белков S‑слоя этих штаммов (табл. 1). Расчетная масса белков S-слоя штаммов ATCC 8287 и ATCC 14869 соответствовала экспериментальной (табл. 2). Однако мажорный белок S-слоя штамма ATCC 367 имел массу 56 кДа, что не соответствует ни одному из белков, обнаруженных в этом штамме в результате анализа генома. Тем не менее данный белок идентифицирован пептидным фингерпринтом как SlpE и, по-видимому, является его формой, которая образуется в процессе посттрансляционной модификации.
Таблица 1.
Идентификация генов, кодирующих белки S-слоя, в геномах штаммов L. brevis ATCC 367, L. brevis ATCC 8287 и L. brevis ATCC 14869
| Штамм ATCC | Slp-ген | Slp-белок (UniProt), кДа | Slp-ген | Slp-белок (UniProt), кДа | Slp-ген | Slp-белок (UniProt), кДа |
|---|---|---|---|---|---|---|
| 367 | LVIS_2083 | Q03NT3 48 |
KY273133 | SlpE 52 |
||
| 8287 | slpA | Q05044 48 |
||||
| 14 869 | slpB | Q8GFE5 52 |
slpC | Q8GFE4 49 |
slpD | Q8GFE3 45 |
Таблица 2.
Экспериментальная идентификация белков S-слоя в клетках штаммов L. brevis ВКПМ В-5563 (ATCC 367), L. brevis ВКПМ В-10405 (ATCC 8287) и L. brevis ВКПМ В-10903 (ATCC 14869) из коллекции ВКПМ
| В-5563 (ATCC 367) | В-10405 (ATCC 8287) | В-10903 (ATCC 14869) | |||
|---|---|---|---|---|---|
| Белок | ММ, кДа | Белок | ММ, кДа | Белок | ММ, кДа |
| SlpE_glyc | 56 | SlpB | 52 | ||
| SlpE | 52 | ||||
| LVIS_2083 | 48 | SlpA | 48 | SlpD | 45 |
Впервые проведен анализ гликозилирования белков S-слоя штаммов L. brevis ATCC 367, ATCC 8287 и ATCC 14 869. Устойчивое специфическое окрашивание в геле приобретал только белок с молекулярной массой 56 кДа штамма L. brevis ATCC 367 (рис. 1б). Таким образом, различие расчетной и фактической массы белка SlpE действительно объясняется гликозилированием.
Известны случаи гликозилирования белков S‑слоя среди штаммов рода Lactobacillus. Достоверно определено и детально исследовано гликозилирование белков S-слоя у штаммов L. buchneri 41 021/251, CD034, NRRL B-30929 [18] и L. kefiri CIDCA 83111 [19].
Основной белок S-слоя SlpB штамма L. buchneri CD034 гликозилирован по O-типу по четырем сериновым остаткам в последовательности: S(152)-A-S(154)-S(155)-A-S(157). Каждый сериновый остаток содержит гликановый хвост, состоящий в среднем из семи глюкоз, связанных последовательно α(1-6)-гликозидной связью. Белок S-слоя SlpN штамма L. buchneri NRRL B-30 929 содержит идентичный мотив и также гликозилирован по типу штамма CD034. Было обнаружено, что белки LbGH25B (L. buchneri CD034) и LbGH25N (L. buchneri NRRL B-30 929) содержат тот же мотив S-A-S-S-A-S и гликозилируются подобным образом.
Гликозилирование белка S-слоя L. kefiri CI-DCA 83 111 происходит схожим с L. buchneri образом: четыре сериновых остатка в последовательности SSASSASSA гликозилируются по O-типу. Гликановый хвост состоит из пяти–восьми остатков глюкозы и оканчивается галактуроновой кислотой (до трех остатков). Кроме того, обнаружен гликозилированный пептид с последовательностью TTTSAE и характерным гликановым хвостом в пять–восемь остатков глюкозы и галактуроновой кислотой (1–2 остатка). Также открыто гликозилирование белка S-слоя L. kefiri CIDCA 83 111 по N-типу.
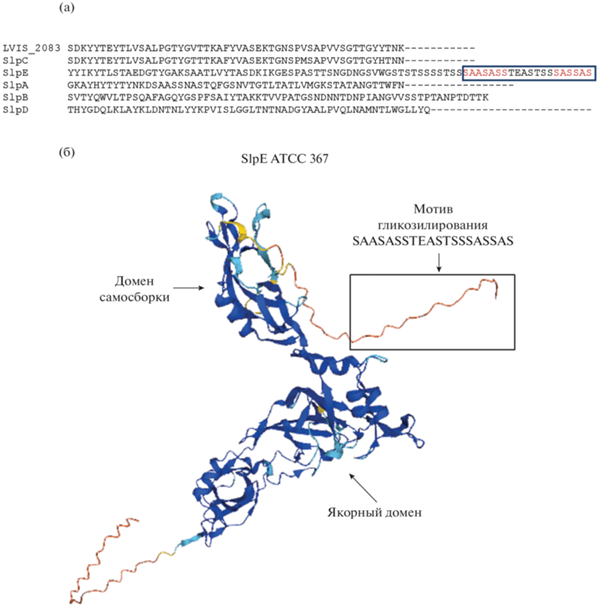
Филогенетический анализ последовательностей белков штаммов L. brevis LVIS_2083, SlpE (ATCC 367), SlpA (ATCC 8287), SlpB, SlpC и SlpD (ATCC 14 869) подтверждает низкий уровень гомологии в данной группе (рис. 2). Гомологичными оказываются только N-концевые последовательности, приходящиеся на консервативный сигнальный пептид и домен присоединения к клеточной стенке. C-концевые последовательности, которые формируют домен самосборки, существенно различаются. Мотив SASSAS, специфичный для гликозилированных белков L. buchneri и L. kefiri, присутствует только у SlpE штамма ATCC 367 (рис. 3а). Множественное выравнивание последовательностей белков SlpE L. brevis ATCC 367, SlpB L. buchneri CD034 (NCBI Accession number – AFS00631.1) и Slp L. kefiri CIDCA 83111 (NCBI Accession number – SCA78675.1) указывает на разное положение мотива SASSAS. У SlpE мотив располагается на краю C-конца последовательности, в то время как у SlpB и Slp мотив располагается ближе к N-концу в районе 150 остатка (рис. 3б). Такое расположение сайтов гликозилирования может быть связано с различной архитектурой белков S-слоя, для которых положение домена самосборки может быть как у L. brevis с C-конца, так и как у L.crispatus и L. acidophilus с N-конца. Таким образом, присутствие мотива SASSAS в последовательности белка S-слоя ассоциировано с посттрансляционным гликозилированием.
Рис. 2.
Филогенетический анализ последовательностей белков штаммов L. brevis LVIS_2083, SlpE (ATCC 367), SlpA (ATCC 8287), SlpB, SlpC и SlpD (ATCC 14869).
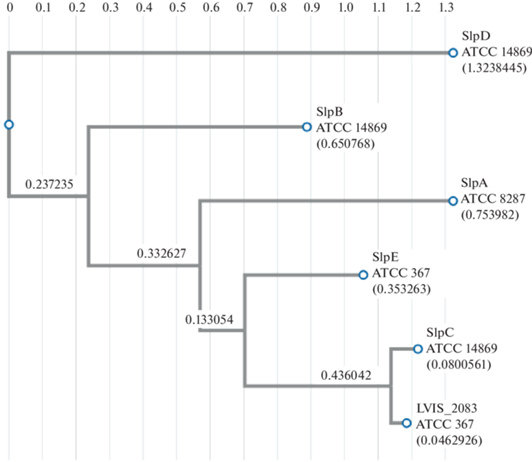
Рис. 3.
Положение мотива гликозилирования у белка SlpE штамма Lactobacillus brevis ATCC 367: а – множественное выравнивание С-концевых последовательностей белков штаммов L. brevis LVIS_2083, SlpE (ATCC 367), SlpA (ATCC 8287), SlpB, SlpC и SlpD (ATCC 14869), мотив гликозилирования выделен цветом и обведен в рамку, б – пространственная модель белка SlpE штамма L. brevis ATCC 367 и позиция мотива гликозилирования.
Функционализация белков S-слоя с помощью гликоинженерии является перспективным направлением фундаментальных и прикладных исследований белков S-слоя и может быть использована как для анализа процесса гликозилирования белков S-слоя, так и для их искусственного оснащения функциональными мотивами гликозилирования для (нано)биотехнологических или биомедицинских целей [20]. Успешность стратегии закрепления гликозилированных белков S-слоя на клеточной поверхности была продемонстрирована на примере E. coli, на поверхности которой были реконструированы рецепторы токсина Шига, способного адсорбировать и нейтрализовать этот токсин с очень высокой эффективностью [21]. Возможность рекристаллизации S-слоя in vitro на широком спектре подложек позволяет воспользоваться уникальным преимуществом кристаллической, регулярной “иммобилизационной матрицы”, обеспечивающей контролируемое отображение на поверхности “функциональных” мотивов гликозилирования [20].
Использование гликозилированных белков S‑слоя создает новую стратегию для функционализации поверхностей. Эта стратегия обладает большим биотехнологическим потенциалом, включающим в себя разработку аффинных матриц, биосовместимых поверхностей и мукозальных вакцин. Экспонирование гликозилированных белков S‑слоя на клеточной стенке бактерий можно использовать для адресной доставки антигенов, скрининга библиотек пептидов, разработки биокатализаторов и диагностики [22–24]. Белки S‑слоя лактобацилл, в том числе впервые обнаруженный нами белок SlpE из L. brevis, являются потенциальными кандидатами для использования в системах экспонирования и доставки антигенов. Применение пробиотических лактобацилл, несущих гликозилированные белки S-слоя, для направленной доставки антигенов является перспективным способом разработки новых вакцин.
ЗАКЛЮЧЕНИЕ
Проведен анализ гликозилирования белков S‑слоя штаммов L. brevis ATCC 367, ATCC 8287 и ATCC 14 869 методом окрашивания гликопротеинов в реакции с перйодной кислотой и основанием Шиффа. Впервые обнаружено гликозилирование белка S-слоя штамма L. brevis ATCC 367. В составе поверхностного S-слоя штамма L. brevis ATCC 367 имеются две формы белка SlpE, основная из которых с массой 56 кДа гликозилирована. Гликозилирования белков S-слоя у двух других штаммов L. brevis не обнаружено. Анализ аминокислотных последовательностей белков S-слоя штаммов L. brevis и известных гликозилированных белков штаммов L. buchneri CD034 и L. kefiri CIDCA 83 111 показал, что наличие гликозилирования ассоциировано с присутствием мотива SASSAS в последовательности белка.
Работа выполнена в рамках государственного задания НИЦ “Курчатовский институт”.
Список литературы
Новокшонов А.А., Соколова Н.В. // Эффективная фармакотерапия. Эпидемиология и инфекции. 2013. Т. 54. № 2. С. 20.
Peters V.B.M., van de Steeg E., van Bilsen J., Meije-rink M. // Benef. Microbes. 2019. V. 10. № 3. P. 225. https://doi.org/10.3920/BM2018.0066
De Boeck I., van den Broek M.F.L., Allonsius C.N. et al. // Cell Rep. 2020. V. 31. № 2. P. 1. https://doi.org/10.1016/j.celrep.2020.107674
Lee J.T., Frank D.N., Ramakrishnan V. // Am. J. Rhinol. Allergy. 2016. V. 30. № 1. P. 3. https://doi.org/10.2500/ajra.2016.30.4255
Ishida Y., Nakamura F., Kanzato H. et al. // Biosci. Biotech. Biochem. 2005. V. 69. № 9. P. 1652. https://doi.org/10.1271/bbb.69.1652
Isolauri E., Joensuu J., Suomalainen H. et al. // Vaccine. 1995. V. 13. № 3. P. 310. https://doi.org/10.1016/0264-410X(95)93319-5
De Vrese M., Rautenberg P., Laue C. et al. // Eur. J. N-utrition. 2005. V. 44. № 7. P. 406. https://doi.org/10.1007/s00394-004-0541-8
Paineau D., Carcano D., Leyer G. // FEMS Immunol. Med. Microbiol. 2008. V. 53. № 1. P. 107. https://doi.org/10.1111/j.1574-695X.2008.00413.x
Kobayashi N., Saito T., Uematsu T. et al. // Int. Immunopharmacol. 2011. V. 11. № 2 P. 199. https://doi.org/10.1016/j.intimp.2010.11.019
Kotani Y., Shinkai S., Okamatsu H. et al. // Immunol. Ageing. 2010. V. 7 № 11. P. 1. https://doi.org/10.1186/1742-4933-7-11
Wells J.M., Mercenier A. // Nat. Rev. Microbiol. 2008. V. 6. № 5. P. 349. https://doi.org/10.1038/nrmicro1840
De Vuyst L., Degeest B. // FEMS Microbiol. Rev. 1999. V. 23. № 2. P. 153. https://doi.org/10.1111/j.1574-6976.1999.tb00395.x
Rykov S.V., Yegorov Y.E., Vishnyakova H.S., Berezi-na O.V. // Russ. J. Bioorg. Chem. 2018. V. 44. № 2. P. 199. https://doi.org/10.1134/S1068162018010156
Åvall-Jääskeläinen S., Kylä-Nikkilä K., Kahala M. et al. // Appl. Environ. Microbiol. 2002. V. 68. № 12. P. 5943.
Avall-Jääskeläinen S., Palva A. // FEMS Microbiol. Rev. 2005. V. 29. № 3. P. 511. https://doi.org/10.1016/j.femsre.2005.04.003
Mobili P., Serradell M. de L., Trejo S.A. et al. // Antonie Van Leeuwenhoek. 2009. V. 95. № 4. P. 363. https://doi.org/10.1007/s10482-009-9322-y
Francius G., Lebeer S., Alsteens D. et al. // ACS Nano. 2008. V. 2. № 9. P. 1921. https://doi.org/10.1021/nn800341b
Anzengruber J., Pabst M., Neumann L. et al. // Glycoconjugate J. 2014. V. 31. № 2. P. 117. https://doi.org/10.1007/s10719-013-9505-7
Cavallero G.J., Malamud M., Casabuono A.C. et al. // J. Proteomics. 2017. V. 162. P. 20. https://doi.org/10.1016/j.jprot.2017.04.007
Messner P., Steiner K., Zarschler K., Schäffer C. // Carbohydr. Res. 2008. V. 343. № 12. P. 1934. https://doi.org/10.1016/j.carres.2007.12.025
Paton A.W., Morona R., Paton J.C. // Nat. Med. 2000. V. 6. № 3. P. 265. https://doi.org/10.1038/73111
Leenhouts K., Buist G., Kok J. // Antonie Van Leeuwenhoek. 1999. V. 76. № 1–4. P. 367.
Yu Q., Zhu L., Kang H., Yang Q. // Human Vaccin. Immunother. 2013. V. 9. № 4. P. 805. https://doi.org/10.4161/hv.23302
Hynönen U., Palva A. // Appl. Microbiol. Biotechnol. 2013. V. 97. № 12. P. 5225. https://doi.org/10.1007/s00253-013-4962-2
Дополнительные материалы отсутствуют.
Инструменты
Вестник Военного инновационного технополиса «ЭРА»


