Российская археология, 2023, № 3, стр. 57-71
Индивид волынцевского времени из Куриловки: первые археогенетические данные
Т. В. Андреева 1, 2, 3, *, А. Б. Малярчук 2, 3, **, В. Е. Родинкова 4, ***, А. Д. Сошкина 2, 3, ****, Е. В. Рождественских 1, *****, М. В. Добровольская 4, ******, Е. И. Рогаев 1, 3, 5, *******
1 Центр генетики и наук о жизни, Университет “Сириус”
Сочи, Россия
2 Институт общей генетики им. Н.И. Вавилова РАН
Москва, Россия
3 Центр генетики и генетических технологий, Московский государственный университет им. М.В. Ломоносова
Москва, Россия
4 Институт археологии РАН
Москва, Россия
5 Медицинская школа Чан Массачусетского университета, департамент психиатрии
Шрусбери, США
* E-mail: an_tati@mail.ru
** E-mail: sasha-m98@mail.ru
*** E-mail: vlasta2004@mail.ru
**** E-mail: anna.soshkina91@gmail.com
***** E-mail: l.i.r.o@mail.ru
****** E-mail: mk_pa@mail.ru
******* E-mail: rogaev.ei@talantiuspeh.ru
Поступила в редакцию 11.06.2022
После доработки 11.06.2022
Принята к публикации 11.10.2022
- EDN: KGTTIT
- DOI: 10.31857/S0869606323030030
Аннотация
Несмотря на достижения последних лет в области палеогеномных исследований, генетические особенности и разнообразие раннеславянского населения остаются неисследованными в связи с распространенным у древних славянских племен обрядом трупосожжения. Поэтому ценна каждая находка антропологического материала из раннеславянских памятников. Таким уникальным объектом является череп из жилища, отнесенного к волынцевской культуре, памятника Куриловка 2 (Курская обл.), который датируется концом VII – первой половиной/серединой VIII в. Из зуба была выделена ДНК и использована для проведения генетического анализа. Мы реконструировали полную последовательность митохондриальной ДНК и определили, что она принадлежит к европейской гаплогруппе H1b. Результаты филогенетического анализа свидетельствуют об общих материнских линиях индивида из Куриловки со средневековыми и современными европейскими образцами и позволяют предположить общность раннеславянских и северо-западных европейских митохондриальных линий.
История возникновения и расселения славянских народов продолжает оставаться актуальным дискуссионным вопросом, несмотря на результаты анализа археологических, исторических, палеоантропологических и лингвистических источников. Достижения последних лет в области палеогеномики свидетельствуют об эффективности новых методов в исследовании происхождения и миграций древних народов, однако генетические особенности раннеславянского населения остаются практически не известны. Основная проблема палеогенетического изучения славян до X в. связана с распространением обряда трупосожжения. В ряде работ было показано, что непродолжительное воздействие высоких температур не приводит к полной деградации ДНК (Rogaev et al., 2009; Grela et al., 2021; Ottoni et al., 2009), однако погребальные традиции в культурах славян предполагали долговременное высокотемпературное горение (Клещенко, Решетова, 2019, Сыроватко, Клещенко, 2017), поэтому обнаружить не поврежденные огнем скелеты можно крайне редко. Любые скелетные останки, происходящие из раннеславянских памятников, имеют большую ценность. Таким уникальным объектом является череп человека из жилища волынцевской археологической культуры многослойного памятника Куриловка 2.
Современная палеогеномика вводит в научный оборот сотни образцов. Особое внимание предлагается уделять формированию выборки для палеогеномного исследования (принадлежность к одному памятнику, единой или разным культурно-хронологическим периодам его существования и пр.), выявлению кровных связей, поскольку открытые структуры отражают самые разные стороны жизни социумов (Kristiansen, 2022). Но насколько информативен может быть анализ индивида? Обладает ли он самодостаточной информативностью? Этот вопрос актуален для ситуаций, которые по ряду причин (погребальный обряд, природно-климатические условия) не сулят надежды на получение больших серий образцов.
Поселение Куриловка 2 расположено в Суджанском р-не Курской обл., в северной части Днепровского лесостепного Левобережья, в месте впадения р. Суджа в р. Псел. Здесь выявлены несколько горизонтов заселения, связанных с носителями прото- и раннеславянских традиций. Наиболее поздний из них соотносится с волынцевской культурой.
Памятники волынцевского типа датируются в целом концом VII – VIII (началом IX) в. (Гавритухин, Щеглова, 1996; Комар, 2012. С. 140–151) и распространены на территории лесостепной и южной части лесной зон от Среднего Поднепровья (включая примыкающую к реке полосу правого берега) до поречья Северского Донца и от Верхнего Подесенья до бассейна Средней и Нижней Ворсклы. В северной части ареала они сменяют колочинскую культуру, в южной – пеньковскую, финал которых на данной территории приходится на вторую половину/третью четверть VII в. (Родинкова, 1996).
Исследователями высказывались различные мнения о происхождении волынцевских древностей. Все они сводятся к двум основным позициям. Согласно первой, волынцевская культура – результат трансформации колочинской и пеньковской раннеславянских общностей, существовавших на Днепровском Левобережье в третьей четверти I тыс. н.э. (Этнокультурная карта…, 1985. С. 121–124; и др.). В соответствии с другой точкой зрения, наиболее ранние волынцевские памятники по характеру керамики и особенностям домостроительства (в частности, по наличию в жилищах глинобитных печей) сближаются с так называемыми памятниками типа Сахновки в Поросье и появляются в результате миграции с правого на левый берег Днепра наследников пражских традиций. На Днепровском Левобережье переселенцы вступают во взаимодействие с местным колочинским и пеньковским населением, формируя “сахновско-волынцевский” горизонт волынцевской культуры, который датируется концом VII – первой половиной/серединой VIII в. (Обломский, Щеглова, 1996; Гавритухин, Щеглова, 1996; и др.).
Именно к этому горизонту принадлежит жилище 13 из раскопа 3 поселения Куриловка 2, в котором выявлены человеческие останки. От жилища сохранился четырехугольный котлован полуземлянки размерами 3.8 × 4.7 м, углубленный в материк на 0.09–0.40 м, и серия выкопанных внутри котлована и рядом с ним ям. В северном углу постройки исследована печь, сложенная из глиняных “вальков” или “конусов”. Из жилища происходят фрагменты характерных сосудов с бугристой от обильной примеси шамота поверхностью, расширением в верхней трети тулова, с отогнутыми и украшенными пальцевыми вдавлениями венчиками. Имеются также обломки лощеной посуды с вертикальными венчиками, подражающей волынцевской гончарной керамике, и слабопрофилированные формы, показательные для колочинской культуры. Материал в целом немногочислен, особенно в нижней части заполнения: здесь найдены всего 14 фрагментов керамики из 176, собранных в сооружении в целом. В процессе археологизации в придонном слое откладываются, как правило, предметы, оставшиеся во внутреннем пространстве помещения после того, как оно перестало функционировать и начало разрушаться. Отмеченное распределение находок свидетельствует, что жилище 13 было покинуто не внезапно, а в “штатном режиме”, люди ушли из него, забрав все, что считали ценным.
Обнаруженные в жилище антропологические останки представлены костями черепа: лобная кость – полностью, парные теменные – во фрагментах, парные верхнечелюстные с зубами в альвеолярном сочленении; парные скуловые, фрагментированная затылочная, клиновидная (рис. 1). Нижняя челюсть, а также кости посткраниального скелета отсутствовали.
Рис. 1.
Фрагменты черепа ребенка из жилища на селище Куриловка.
Fig. 1. Fragments of a child’s skull from a dwelling in Kurilovka

Останки зафиксированы на высоте 0.1–0.2 м над дном котлована, т.е. в нижней, практически лишенной находок части заполнения, формирование которой связано с разрушением самой полуземлянки и ее интерьера. Костные фрагменты образовывали два скопления на расстоянии 0.6–1.0 м к югу от устья печи. Еще несколько фрагментов этого же черепа найдены за пределами постройки, в культурном слое, на расстоянии около 4 м к северу от скоплений. Вероятно, они оказались перемещены в связи с деятельностью землероек, следы которых на памятнике весьма многочисленны.
Сохранность костной ткани хорошая, поверхностный слой практически не поврежден. Как правило, поверхность костной ткани скелетов из грунтовых погребений характеризуется различного рода нарушениями (шероховатости, отслаивания поверхностного слоя ткани, повреждения мелкими корнями растений и пр.), которых мы в данном случае не фиксируем. Заметны нарушения в виде разломов костей свода черепа. Все повреждения, судя по характеру разломов, были получены, когда кость утратила значительную часть органических соединений и влагу. Это значит, что был разрушен скелетированный череп, а не тело человека. Таким образом, сочетание сохранности и комплектности останков позволяют предполагать, что мы имеем дело с депонированием в пространстве дома черепа без нижней челюсти.
Возрастное определение может быть выполнено по оценке прорезывания и формирования зубов молочной и основной генерации. Осмотр позволяет оценить возраст как около 3–4 лет. Рентгеновское изображение подтверждает это возрастное определение по состоянию молочных зубов и закладок основной генерации, зубы без выраженных патологий (рис. 2). Определение пола методами физической антропологии не может быть выполнено для ребенка этого возраста.
Рис. 2.
Зубы ребенка из жилища на селище Куриловка: А – рентгенограмма зубов верхней челюсти (данные использованы для определения возраста ребенка); Б – зуб, использованный для выделения ДНК и проведения генетического анализа.
Fig. 2. Teeth of a child from a dwelling in Kurilovka
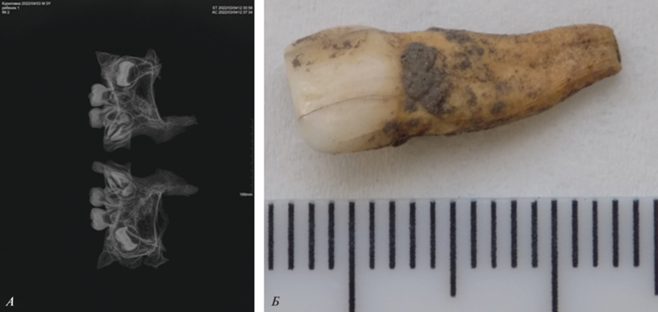
Экспериментальные работы проводили в специальных лабораторных помещениях, предназначенных для работы с древней ДНК. Фрагмент корня зуба весом около 90 мг (рис. 2, Б) после механической очистки от загрязнений измельчали в порошок и использовали для выделения ДНК по описанной ранее методологии (Andreeva et al., 2022). Геномную ДНК использовали для приготовления фрагментных геномных библиотек (Gansauge et al., 2017), которые секвенировали на платформе NovoSeq 6000 (Illumina). Для реконструкции последовательности митохондриальной ДНК (мтДНК) полученные прочтения после удаления адаптеров (Schubert, Lindgreen, Orlando, 2016) были картированы на референсную последовательность мтДНК человека (rCRS, NC_012920.1) и на референсный геном человека (сборка GRCh37) с использованием BWA (Li, Durbin, 2009). Аутентичность картированных на мтДНК последовательностей оценивали с помощью MapDamage (Jónsson et al., 2013). Для проведения дальнейшего анализа использовали только короткие прочтения, длиной до 50 пар нуклеотидов, чтобы исключить контаминацию фрагментами современной ДНК. Участки поли-С трактов в позициях 303–315 и 16183–16193 мтДНК проверяли визуально с использованием IGV (Robinson, Zemo jtel, 2018). Митохондриальную гаплогруппу определяли с использованием Haplogrep 2 (Weissensteiner et al., 2016), филогенетический анализ проводили с помощью программных пакетов mtPhyl (Eltsov.org) и MEGA Х (Kumar et al., 2018) с использованием последовательностей полных митохондриальных геномов из баз данных GenBank и AmtDB. Для построения филогенетических деревьев использовали полные последовательности мтДНК, которые выравнивали друг относительно друга с помощью MAFFT (Katoh, Standley, 2013), в качестве аутгруппы использовали последовательность мтДНК шимпанзе (NC001643). Полученные выравнивания использовали для эволюционного анализа методами минимальной эволюции (Minimum evolution) и метода присоединения ближайшего соседа (Neighbor joining) с поддержкой узлов бутстрепом с 2000 повторов в программе MEGAX (Katoh, Standley 2013).
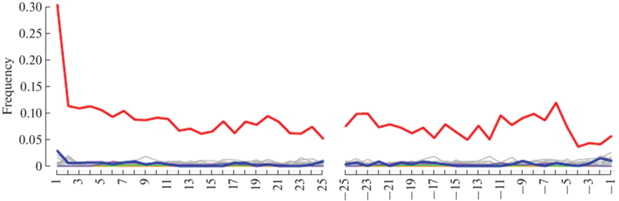
Тотальная геномная ДНК была выделена из фрагмента зуба и использована для приготовления фрагментных геномных библиотек. В результате их секвенирования было получено более 8 млрд прочтений, 3.79% которых картируются на геном человека, остальные представлены геномами почвенных микроорганизмов. Только 1631 прочтение было картировано на референсную последовательность мтДНК, что позволило реконструировать полную последовательность митохондриальной ДНК со средним покрытием x3.8. Профиль замен на концах фрагментов, картированных на мтДНК, соответствовал древней ДНК, так как имеет повышенную частоту замен цитозина на тимин (рис. 3).
Рис. 3.
Профиль нуклеотидных замен, полученный с использованием программы mapDamage 2.0 (Jónsson et al. 2013) для прочтений, картированных на мтДНК человека. Наблюдаются повышенный уровень замен C > T (красный) по всей длине фрагментов, а также значительное увеличение частоты таких замен на концах фрагментов, специфичное для древней ДНК и обусловленное постмортальными модификациями ДНК, что подтверждает подлинность древней ДНК, выделенной из зуба.
Fig. 3. Profile of nucleotide substitutions obtained using the mapDamage 2.0 software (Jónsson et al., 2013) for reads mapped to human mtDNA
Полученная последовательность мтДНК относится к гаплогруппе H1b, выявленные отличия ее от референсной последовательности мтДНК человека представлены в табл. 1.
Таблица 1.
Варианты, выявленные в последовательности мтДНК исследованного образца Table 1. Variants identified in the mtDNA sequence of the studied sample
| Координата (rCRS) | Референсный аллель | Вариант, выявленный в исследованном образце | Число прочтений, содержащих альтернативный аллель | Частота в современных популяциях (по базе данных GnomAD) |
|---|---|---|---|---|
| 263 | A | G | 9 | 0.991 |
| 315.1 | – | C | 2 | –* |
| 750 | A | G | 3 | 0.983 |
| 1438 | A | G | 7 | 0.956 |
| 3010 | G | A | 4 | 0.163 |
| 4769 | A | G | 3 | 0.984 |
| 8860 | A | G | 7 | 0.984 |
| 15 326 | A | G | 6 | 0.993 |
| 16 183 | A | C | 3 | 0.005 |
| 16 189 | T | C | 2 | 0.249 |
| 16 356 | T | C | 3 | 0.032 |
| 16 519 | T | C | 5 | 0.657 |
Гаплогруппа H1b представляет собой одну из 60 ветвей предковой гаплогруппы H1, которая, в свою очередь, принадлежит к самой распространенной в современной Европе гаплогруппе H (PhyloTree – mtDNA tree Build 17). Возникновение гаплогруппы H1b связано с тремя последовательными заменами в корневой гаплогруппе H. Первая из них (G3010A) сформировала подкладу H1, далее замены Т16189С и T16356С привели к появлению ветви, соответствующей гаплогруппе H1b, к которой принадлежит образец из Куриловки. При этом замена T16189C расположена в поли-С тракте и характеризуется высокой частотой спорадических мутаций, обусловленных ошибками репликации в повторяющемся участке, поэтому только вариант T16365C является маркером гаплогруппы H1b (Van Oven, Kayser, 2009).
Базальная гаплогруппа Н является самой распространенной (более 50% в некоторых популяциях) митохондриальной гаплогруппой в Европе. Примерно половина носителей гаплогруппы H (и до 27% от всех митохондриальных гаплогрупп) принадлежат ее дочерней ветви H1 (Roostalu et al., 2007), которая наиболее часто встречается в современных популяциях Иберийского полуострова, откуда эта материнская линия, вероятно, начала свое распространение по всей Европе (Ottoni et al., 2010). Ее клада H1b, к которой принадлежит образец из Куриловки, встречается во всех европейских популяциях и достигает максимальной частоты у населения современной Финляндии и Эстонии – до 7% от всех носителей гаплогруппы H (Loogväli et al., 2004).
Для более детального анализа мы провели прямое сравнение митохондриальной последовательности образца из Куриловки с полными митохондриальными геномами, представленными в открытых информационных источниках. Использованные для поиска базы данных включают в себя образцы как современных, так и древних индивидов (GenBank; AmtDB; Ian Logan – mt-DNA; YFull – MTree 1.02). Из них для анализа были отобраны все митохондриальные последовательности, принадлежащие гаплогруппе H1b, для которых указано географическое происхождение или этническая принадлежность. Также в анализ были включены три последовательности мтДНК древних образцов с территории России, принадлежащие гаплогруппе H1b, из собственной базы митохондриальных геномных последовательностей (лаборатории эволюционной геномики ИОГен РАН и НТУ “Сириус”), включающей более 400 образцов от палеолита до начала XX в.: из могильника Репная балка (среднедонская катакомбная культура, середина III тыс. до н.э., Воронежская обл.), из кургана “Чайка” (культура раннего железного века, Крым) и из захоронения Ксизово 17Б (гуннский период, Липецкая обл.). Кроме того, с использованием сервиса BLAST и полной митохондриальной последовательности образца из Куриловки был проведен поиск дополнительных последовательностей, присутствующих в GenBank, наиболее сходных с этим образцом, независимо от их принадлежности к митохонриальной гаплогруппе. По результатам отбора в анализ были включены все найденные с помощью BLAST последовательности мтДНК, отвечающие следующим критериям: Query cover – 100%; Percent Identity – не менее 99.99%. Так как не все митохондриальные последовательности, представленные в научных публикациях, размещаются в указанных выше базах данных, дополнительно мы провели анализ литературных источников, посвященным генетическим исследованиям древних образцов и отобрали образцы, для которых была показана принадлежность к гаплогруппе H1b. Несмотря на то что в литературе присутствует информация о носителях этой гаплогруппы у представителей различных древних культур с территории Европы, включая, например, фатьяновскую (Saag et al., 2021) и трипольскую культуры (Nikitin et al., 2017), алеманнов (O’Sullivan et al., 2018) и др., генетический анализ в этих исследованиях был проведен с использованием отдельных участков мтДНК или только гипервариабельного региона, а принадлежность к гаплогруппе H1b определялась по наличию единственной замены T16365C. Полные митохондриальные последовательности носителей гаплогруппы H1b по результатам анализа литературных данных были реконструированы для двух индивидов: венгерского короля Бела III (Olasz et al., 2019) и индивида из захоронения раннего булгарского периода (VIII–IX вв.) с территории Волго-Камского региона (Szeifert et al., 2022).
Таким образом, для проведения сравнительного анализа с мтДНК индивида из Куриловки были отобраны 58 полных митохондриальных последовательностей, включая 36 современных и 22 древних митогенома (табл. 2). С использованием последней версии дерева митохондриальных гаплогрупп (PhyloTree – mtDNA tree Build 17) были определены митохондриальные гаплогруппы для всех включенных в анализ образцов. Помимо гаплогруппы H1b, к которому принадлежит образец из Куриловки, а также другие 35 включенные в анализ образца, у оставшихся 23 были выявлены митохондриальные гаплогруппы H1, H1b1, H1b5, H1b3, H1ba, H1bs, H1bm, которые в соответствии с филогенетическим деревом www.phylotree.org отличаются от гаплогруппы H1b одной-тремя нуклеотидными заменами.
Таблица 2.
Полные митохондриальные последовательности, использованные для филогенетического анализа Table 2. Complete mitochondrial sequences used for phylogenetic analysis
| Обозначение | Происхождение, период | Гаплогруппа | Число отличий от образца из Куриловки (нт)* | Источник |
|---|---|---|---|---|
| JX153524 | Дания | H1b | 0 | GenBank |
| BelaIII | Венгрия, венгерский король Бела III (1148–1196 гг. н.э.) | H1b | 0 | Wang et al., 2021 |
| MN540478 | Финляндия, эпоха викингов, VII–XIII вв. н.э. | H1+16189 | 1 | Översti et al., 2019 |
| JQ324755 | Испания | H1b | 1 | GenBank |
| JQ324850 | Испания (баск) | H1b | 1 | –“– |
| JQ324924 | Испания (баск) | H1b | 1 | –“– |
| JX152783 | Финляндия | H1+16189 | 1 | –“– |
| JX152798 | Дания | H1b5 | 1 | –“– |
| JX153758 | Дания | H1b | 1 | –“– |
| JX154058 | Финляндия | H1+16189 | 1 | –“– |
| KF161163 | Дания | H1b | 1 | –“– |
| KF162082 | Дания | H1b | 1 | –“– |
| KF162092 | Дания | H1b | 1 | –“– |
| KF162415 | Дания | H1b | 1 | –“– |
| KF162418 | Дания | H1b | 1 | –“– |
| KY670892 | Россия (Псков) | H1b | 1 | –“– |
| KY671082 | Россия (Новгород) | H1+16189 | 1 | –“– |
| MG595757 | Финляндия | H1+16189 | 1 | –“– |
| MN046452 | Испания | H1b | 1 | –“– |
| MN411324 | Болгария | H1b | 1 | –“– |
| VK18 | Россия, Ладога, эпоха викингов, X–XII вв. н.э. | H1b1 | 2 | Margaryan et al., 2020 |
| DS77 | Россия, Репная балка, среднедонская катакомбная культура, середина III тыс. до н.э. | H1b | 2 | Собственные данные |
| MF114211 | Польша, неолит, культура шаровидных амфор, 3400–2800 гг. до н.э. | H1 | 2 | Tassi et al., 2017 |
| RC13 | Волго-Камский регион, ранний булгарский период, IX–X вв. н.э. | H1b | 2 | Szeifert et al., 2022 |
| JQ324649 | Испания (баск) | H1b | 2 | GenBank |
| JX153618 | Финляндия | H1+152+16189 | 2 | –“– |
| JX171107 | Финляндия | H1+16189 | 2 | –“– |
| KF161561 | Дания | H1b | 2 | –“– |
| KF162046 | Дания | H1b | 2 | –“– |
| KF162968 | Дания | H1b | 2 | –“– |
| KJ856753 | Россия (бурят) | H1b3 | 2 | –“– |
| KU867602 | Испания | H1b | 2 | –“– |
| KY496879 | Финляндия | H1+16189 | 2 | –“– |
| KY670910 | Россия | H1b | 2 | –“– |
| KY671008 | Россия (Владимир) | H1b5 | 2 | –“– |
| MK059536 | Дания | H1b | 2 | –“– |
| MN516591 | Финляндия | H1b | 2 | –“– |
| MZ920537 | Испания | H1b | 2 | –“– |
| MZ920740 | Испания | H1+16189 | 2 | –“– |
| KS6 | Россия, Ксизово 17Б, гуннское время | H1b | 3 | Собственные данные |
| VK313 | Дания, эпоха викингов, 850–900 гг. н.э. | H1b | 3 | Margaryan et al., 2020 |
| VK495 | Эстония, ранние викинги, VIII в. н.э. | H1b | 3 | Margaryan et al., 2020 |
| KF161743 | Дания | H1b | 3 | GenBank |
| MH120522 | Польша | H1b | 3 | –“– |
| MZ921145 | Испания (баск) | H1b | 3 | –“– |
| MF114216 | Польша, бронзовый век, культура шаровидных амфор, ок. III тыс. до н.э. | H1b | 4 | Tassi et al., 2017 |
| MN540478 (PCA0090) | Польша, готы, 200–400 гг. н.э. | H1cg | 4 | Stolarek et al., 2019 |
| MF114217 | Польша, неолит | H1b | 4 | Tassi et al., 2017 |
| VK398 | Швеция, викинги, X–XII вв. н.э. | H1b1 | 4 | Margaryan et al., 2020 |
| OBKR79 (MF498711) | Германия, бронзовый век | H1ba | 4 | Knipper et al., 2017 |
| MT588290 | Польша, неолит | H1b | 4 | Juras et al., 2021 |
| I5836 | Германия, медный век | H1b1+16362 | 5 | Olalde et al., 2018 |
| I2618 | Англия, бронзовый век | H1bs | 8 | Olalde et al., 2018 |
| AS16 | Крым, ранний железный век | H1b | 8 | Собственные данные |
| DA199** | Венгрия, средние века | H1ba | 9 | De Barros Damgaard et al., 2018 |
| Val107 | Испания, бронзовый век | H1bm | 9 | González-Fortes et al., 2019 |
| Val145 | Испания, бронзовый век | H1bm | 9 | –“– |
| Val146 | Испания, бронзовый век | H1bm | 9 | –“– |
Примечание. Серым цветом выделены древние образцы ДНК. *При анализе не учитывались позиции, в которых не определен нуклеотид (обозначен N в доступной последовательности из GenBank). **Образец DA199 исключен из филогенетического анализа в связи с большим количеством позиций в нем с неопределенными нуклеотидами.
Сравнение с современными индивидами позволило обнаружить полное совпадение митохондриальной геномной последовательности образца из Куриловки с современным индивидом датского происхождения (JX153524). Это свидетельствует об относительно недавнем (не ранее, чем I тыс. н.э.) существовании общего предка по материнской линии современных европейцев, носителей гаплогруппы H1b, и индивида из Куриловки. Наименьшее число отличий (1 нуклеотидная замена) было выявлено при сравнении полной последовательности мтДНК образца из Куриловки с последовательностями современных жителей Испании (включая басков), Дании, Финляндии и северо-западных регионов России (Псков и Новгород).
Сравнение с древними образцами показало полное совпадение мтДНК последовательности индивида из Куриловки с митохондриальной последовательностью венгерского короля Бела III (Olasz et al., 2019; Wang et al., 2021), матерью которого была Ефросинья Мстиславовна, дочь великого киевского князя Мстислава Владимировича.
Представитель эпохи викингов из захоронения c территории Финляндии (Луистари, 600–1200 гг. н.э.) отличается от образца из Куриловки одной нуклеотидной заменой в последовательности мтДНК. Следует отметить, что мтДНК этого образца из Финляндии содержит 12 позиций с неопределенными нуклеотидами, поэтому общее число различий с последовательностью мтДНК образца из Куриловки может быть больше, чем одно. Различия в две нуклеотидных замены выявлены между образцом из Куриловки и последовательностями мтДНК трех древних индивидов: представителем культуры шаровидных амфор с территории Польши (MF114211), среднедонской катакомбной культуры Воронежской области (DS77) и ранне-булгарского захоронения VIII–IX вв. Волго-Камского региона (RC13).
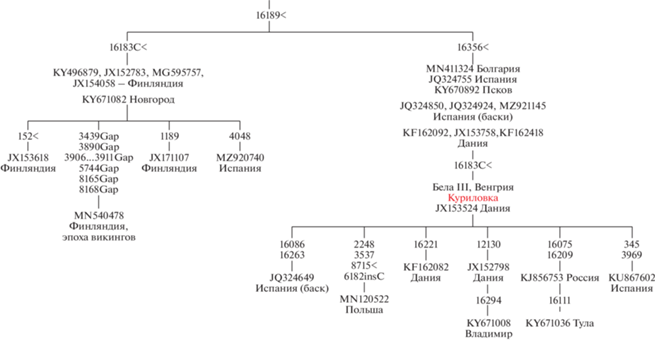
Дальнейший анализ полной последовательности мтДНК мы проводили с помощью построения филогенетических деревьев. Такой анализ позволяет найти вероятное положение последовательностей ДНК на филогенетическом дереве и определить возможное родство близких, но различающихся митотипов. Для определения положения ДНК индивида из Куриловки на дереве митохондриальных гаплогрупп мы провели филогенетический анализ полных последовательностей мтДНК всех отобранных по базам данных образцов 53 образцов и образца из Куриловки с использованием программы mtPhyl, в которой реализован метод максимальной экономии (Maximum parsimony) с учетом митохондриальных гаплогрупп человека. Анализ показал объединение мтДНК из Куриловки с современными жителями Испании, Дании, Польши и Северо-запада России (рис. 4).
Рис. 4.
Фрагмент филогенетического дерева гаплогруппы H1b, построенного с использованием программного пакета mtPhyl. Мутации на ветвях указаны относительно референсной последовательности мтДНК (rCRS); для транзиций указан только номер нуклеотидной позиции, для трансверсий приводится нуклеотидная замена; символом < обозначены позиции повторяющихся мутаций: Gap – неопределенные позиции в мтДНК. Для каждого образца указан его номер в GenBank и место его происхождения.
Fig. 4. A fragment of the phylogenetic tree of haplogroup H1b constructed with the mtPhyl software package
Дополнительный филогенетический анализ полных митохондриальных последовательностей был проведен с использованием метода минимальной эволюции (Minimum evolution) и метода присоединения ближайшего соседа (Neighbor joining). Топология полученных в результате анализа филогенетических деревьев, построенных двумя методами, оказалась сходной для ветвей, включающих образец из Куриловки (см. рис. 4). Было показано, что он имеет наибольшее сходство с современными образцами из Испании, Дании, Финляндии и России (рис. 5). Ближайшие к нему древние митотипы выявлены у древних жителей Центрального и Волго-Камского регионов Европейской равнины DS77 (среднедонская катакомбная культура) и RC13 (булгарский период, IX–X вв. н.э., Поволжье).
Рис. 5.
Филогенетическое дерево гаплогруппы H1b, построенное методом минимальной эволюции. Эволюционный анализ проведен с использованием MEGAX (Kumar et al., 2018). Все неоднозначные позиции были удалены для каждой пары анализируемых последовательностей. Консенсунсное дерево получено с поддержкой узлов бутстрепом с 2000 повторов. Древние митогеномы отмечены курсивом.
Fig. 5. Phylogenetic tree of haplogroup H1b constructed by the minimum-evolution method. Evolutionary analysis was conducted with MEGAX (Kumar et al., 2018)
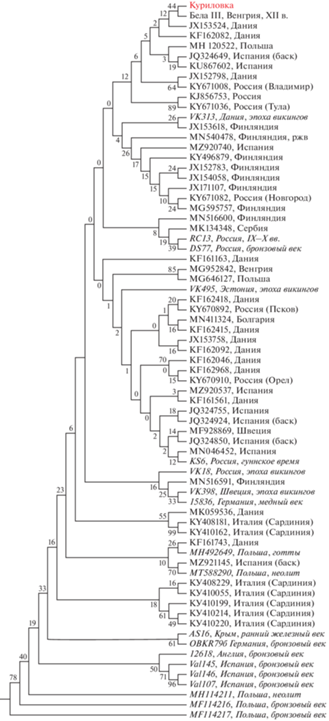
Следует отметить, что по результатам проведенного анализа (табл. 2 и рис. 5) митотип образца из Куриловки имеет максимальное сходство с митотипом венгерского короля Бэлы III (1148–1196). Митохондриальный геном он получил от матери – Ефросиньи Мстиславовны (1130–1193), которая в свою очередь унаследовала свой митохондриальный геном от Любавы Дмитриевны – дочери новгородского посадника Дмитрия Завидича (годы правления 1117–1118).
Митотип образца из Куриловки имеет большее сходство с современными и средневековыми (индивид эпоха викингов и венгерский король) европейскими образцами, а также с древними жителями восточной части Европы, включая Россию, чем с более древним населением неолита и бронзового века Западной Европы (Испании, Англии и Германии). Таким образом, возможны следующие сценарии происхождения данного митотипа индивида из жилища сахновского этапа волынцевской культуры.
Ранний этап волынцевской культуры связан с традициями пражской раннеславянской общности, которая существует в середине IV – VII в. Ее отличительная черта – высокая территориальная динамичность, экспансионистский характер на всех этапах развития. Уже в период формирования культуры ее носители осуществляют первые миграции из региона, где известны наиболее ранние памятники – бассейна Припяти, Центрального Полесья – в Южное Побужье и Поднестровье. В V в. они проникают в верховья Западного Буга и Вислы, бассейн Одера, Восточное Прикарпатье. В VII в. ареал пражской культуры охватывает огромную территорию от Эльбы до Днепра и от Южной Прибалтики до Подунавья и Балканского полуострова (Гавритухин, 1997).
Многочисленные исследования показывают, что пражское население вступало в активное взаимодействие с представителями окружающих культур и народов – лангобардами, гепидами, аварами, византийским населением дунайского лимеса и др. В частности, в низовьях Эльбы, где носители пражских традиций появились не позднее конца VII в., они встретились с саксами, плотные контакты с которыми привели к тому, что в ряде случаев трудно различить лепную керамику этих двух общностей (Шнеевайс, 2017).
Дальнейшее развитие пражских культурных традиций привело к формированию ряда славянских общностей последней четверти I тыс. н.э., таких, как культура Луки-Райковецкой на Правобережье Днепра и памятники сахновско-волынцевского типа, составляющие ранний горизонт волынцевской культуры, на Левобережье Днепра.
Сходство с современными северными европейскими индивидами (датчанами) и с представителями средневекового населения Пскова и Новгорода может отражать корни происхождения и миграции славянского компонента из западной части Восточной Европы. Следует, однако, отметить, что при сравнении с более древними образцами бронзового века и неолита мы обнаружили наибольшее совпадение индивида из Куриловки с индивидом из Репной Балки (~3000 лет до н.э) из той же географической местности, а не с древними западноевропейскими образцами. Коме того, полное совпадение с современным образцом датчанина свидетельствует об их общем ближайшем предке по материнской линии периода средневековья. Таким образом, возможным является и сценарий о генетическом потоке, по крайней мере данной материнской и предковой ей линии, от автохтонного населения с территории России на северо-запад Европы. Интересным также представляется сходство данного митотипа с гаплогруппой, встречающейся у современных басков, что может отражать определенные исторические и миграционные события при формировании населения Европы в период железного века и раннего средневековья.
Разумеется, однозначные выводы не могут быть сделаны на основе анализа единичного образца. Кроме того, низкое качество древней ДНК, сохранившейся в исследованном зубе, не позволяет провести анализ ядерных генетических маркеров. Исследования других костных фрагментов могут быть более перспективны для полногеномного анализа, необходимого для определения вероятной популяционной принадлежности данного индивида.
Палеогеномные исследования позволяют составить новую базу фактов, характеризующих происхождение людей из памятников ранних славянских культур. Впервые выполнен геномный анализ и определена полная последовательность митохондриального генома уникального образца, из памятника раннеславянской культуры VII–VIII вв. Полученные в результате палеогенетического анализа результаты показали принадлежность митохондриальной последовательности индивида из Куриловки европейской гаплогруппе H1b. Обнаружено полное сходство с митогеномом образца, представляющего венгерского короля Бэлу III. Этот митогеном унаследован им от матери Ефросиньи Мстиславовны, происхождение которой связано со средой новгородского боярства. Минимальное количества замен в мтДНК, по которым отличается образец из Куриловки от современных жителей Севера Европы, включая Северо-западный регион России, предполагает близкое родство материнских линий этих индивидов и свидетельствует о связи раннеславянских и северо-западных европейских митохондриальных линий.
Работа выполнена по проекту Минобрнауки России, системный номер No 075-10-2020-116 (No 13.1902.21.0023).
Список литературы
Гавритухин И.О. Хронология пражской культуры // Труды VI Международного Конгресса славянской археологии. Т. 3 / Отв. ред. В. В. Седов. М., 1997. С. 39–52.
Гавритухин И.О., Щеглова О.А. Хронология начальных фаз памятников волынцевского типа // Гапоновский клад и его культурно-исторический контекст. М.: ИА РАН, 1996 (Раннеславянский мир. Археология славян и их соседей; вып. 3). С. 133–135.
Клещенко Е.А., Решетова И.К. Палеоантропологические материалы в реконструкциях образа жизни и погребальной обрядности раннесредневекового населения Восточной Европы. М.: ИА РАН, 2019. 224 с.
Обломский А.М., Щеглова О.А. Некоторые особенности культуры памятников волынцевского типа и спорные вопросы их происхождения // Гапоновский клад и его культурно-исторический контекст. М.: ИА РАН, 1996 (Раннеславянский мир. Археология славян и их соседей; вып. 3). С. 131–133.
Комар А.В. Поляне и северяне // Древнейшие государства Восточной Европы. 2010 г. Предпосылки и пути образования Древнерусского государства. М.: Русский Фонд Содействия Образованию и Науке, 2012. С. 128–191.
Родинкова В.Е. Раннеславянские памятники Среднего Поднепровья и Днепровского Левобережья с датирующими находками // Гапоновский клад и его культурно-исторический контекст. М.: ИА РАН, 1996 (Раннеславянский мир. Археология славян и их соседей; вып. 3). С. 155–162.
Сыроватко А.С., Клещенко Е.А. Грунтовые погребения-кремации XII века: новые исследования курганного могильника Кременье // Археология Подмосковья: материалы науч. семинара. Вып. 13. М.: ИА РАН, 2017. С. 45–56.
Шнеевайс Й. Керамика как свидетельство идентичности и культурного трансфера? Саксонская и славянская керамика VIII–X вв. в Нижнем Поэльбье // Европа от Латена до Средневековья: варварский мир и рождение славянских культур / Отв. ред. В.Е. Родинкова, О.С. Румянцева. М.: ИА РАН, 2017 (Раннеславянский мир. Археология славян и их соседей; вып. 19). С. 90–102.
Этнокультурная карта территории Украинской ССР в I тыс. н.э. / Отв. ред. В.Д. Баран. Киев: Наукова думка, 1985. 184 с.
AmtDB [Электронный ресурс]. URL: https://amtdb.org/ (дата обращения: 15.05.2023).
Andreeva T.V., Manakhov A.D., Gusev F.E. et al. Genomic Analysis of a Novel Neanderthal from Mezmaiskaya Cave Provides Insights into the Genetic Relationships of Middle Palaeolithic Populations // Scientific Reports. 2022. 12. 13016. https://doi.org/10.1038/s41598-022-16164-9
De Barros Damgaard P., Marchi N., Rasmussen S. et al. 137 Ancient Human Genomes from across the Eurasian Steppes // Nature. 2018. V. 557. № 7705. P. 369–374.https://doi.org/10.1038/s41586-018-0094-2
BLAST [Электронный ресурс]. URL: https://blast.ncbi.nlm.nih.gov (дата обращения: 15.05.2023).
Eltsov.org [Электронный ресурс]. URL: http://eltsov.org (дата обращения: 17.01.1922).
Gansauge M.T., Gerber T., Glocke I et al. Single-Stranded DNA Library Preparation from Highly Degraded DNA Using T4 DNA Ligase // Nucleic Acids Research. 2017. V. 45. Iss. 10. P. e79. https://doi.org/10.1093/nar/gkx033
GenBank [Электронный ресурс]. URL: www.ncbi.nlm.nih.gov/genbank/
(дата обращения: 15.05.2023).
González-Fortes G., Tassi F., Trucchi E. et al. A Western Route of Prehistoric Human Migration from Africa into the Iberian Peninsula // Proceedings of the Royal Society. B: Biological Sciences. 2019. V. 286. Iss. 1895. 20182288. https://doi.org/10.1098/rspb.2018.2288
Grela M., Jakubczak A., Kowalczyk M., Listos P., Gryziń-ska M. Effectiveness of Various Methods of DNA Isolation from Bones and Teeth of Animals Exposed to High Temperature // Journal of Forensic and Legal Medicine. 2021. V. 78. 102131. https://doi.org/10.1016/j.jflm.2021.102131
Ian Logan – mtDNA [Электронный ресурс]. URL: http://www.ianlogan.co.uk (дата обращения: 15.05.2023).
Jónsson H., Ginolhac A., Schubert M., Johnson P.L.F., Orlando L. MapDamage2.0: Fast Approximate Bayesian Estimates of Ancient DNA Damage Parameters // Bioinformatics. 2013. 29. P. 1682–1684. https://doi.org/10.1093/bioinformatics/btt193
Juras A., Ehler E., Chyleński M. et al. Maternal Genetic Origin of the Late and Final Neolithic Human Populations from Present-Day Poland // American Journal of Physical Anthropology. 2021. V. 176. Iss. 2. P. 223–36. https://doi.org/10.1002/ajpa.24372
Katoh K., Standley D.M. MAFFT Multiple Sequence Alignment Software Version 7: Improvements in Performance and Usability // Molecular Biology and Evolution. 2013. V. 30. Iss. 4. P. 772–780. https://doi.org/10.1093/molbev/mst010
Knipper C., Mittnik A., Massy K. et al. Female Exogamy and Gene Pool Diversification at the Transition from the Final Neolithic to the Early Bronze Age in Central Europe // Proceedings of the National Academy of Sciences of the United States of America. 2017. V. 114. № 38. P. 10083–10088. https://doi.org/10.1073/pnas.1706355114
Kristiansen K. Archaeology and the genetic revolution in European prehistory. Cambridge: Cambridge University Press, 2022. 76 p.
Kumar S., Stecher G., Li M., Knyaz Ch., Tamura K. MEGA X: Molecular Evolutionary Genetics Analysis across Computing Platforms // Molecular Biology and Evolution. 2018. V. 35. Iss. 6. P. 1547–1549. https://doi.org/10.1093/molbev/msy096
Li H., Durbin R. Fast and Accurate Short Read Alignment with Burrows-Wheeler Transform // Bioinformatics. 2009. V. 25. Iss. 14. P. 1754–1760. https://doi.org/10.1093/bioinformatics/btp324
Loogväli E.-L., Roostalu U., Malyarchuk B.A. et al. Disuniting Uniformity: A Pied Cladistic Canvas of MtDNA Haplogroup H in Eurasia // Molecular Biology and Evolution. 2004. V. 21. Iss. 11. P. 2012–2021. https://doi.org/10.1093/molbev/msh209
Margaryan A., Lawson D.J., Sikora M. et al. Population Genomics of the Viking World // Nature. 2020. V. 585. № 7825. P. 390–396. https://doi.org/10.1038/s41586-020-2688-8
Nikitin A.G., Potekhina I., Rohland N. et al. Mitochondrial DNA Analysis of Eneolithic Trypillians from Ukraine Reveals Neolithic Farming Genetic Roots // PLOS ONE. 2017. V. 12. № 2. P. e0172952. https://doi.org/10.1371/journal.pone.0172952
O’Sullivan N., Posth C., Coia V. et al. Ancient Genome-Wide Analyses Infer Kinship Structure in an Early Medieval Alemannic Graveyard // Science Advances. 2018. V. 4. № 9. P. 1262. https://doi.org/10.1126/sciadv.aao1262
Olalde I., Brace S., Allentoft M.E. et al. The Beaker Phenomenon and the Genomic Transformation of Northwest Europe // Nature. 2018. V. 555. № 7695. P. 190–196. https://doi.org/10.1038/nature25738
Olasz J., Seidenberg V., Hummel S. et al. DNA Profiling of Hungarian King Béla III and Other Skeletal Remains Originating from the Royal Basilica of Székesfehérvár // Archaeological and Anthropological Sciences. 2019. V. 11. № 4. P. 1345–1357. https://doi.org/10.1007/s12520-018-0609-7
Ottoni C., Koon H.E.C., Collins M.J. et al. Preservation of Ancient DNA in Thermally Damaged Archaeological Bone // Naturwissenschaften. 2009. V. 96. № 2. P. 267–278.https://doi.org/10.1007/s00114-008-0478-5
Ottoni C., Primativo G., Kashani B.H. et al. Mitochondrial Haplogroup H1 in North Africa: An Early Holocene Arrival from Iberia // PLOS ONE. 2010. V. 5. № 10. P. e13378. https://doi.org/10.1371/journal.pone.0013378
Översti S., Majander K., Salmela E. et al. Human Mitochondrial DNA Lineages in Iron-Age Fennoscandia Suggest Incipient Admixture and Eastern Introduction of Farming-Related Maternal Ancestry // Scientific Reports. 2019. 9. 16883.https://doi.org/10.1038/s41598-019-51045-8
PhyloTree – mtDNA tree Build [Электронный ресурс]. URL: http://www.phylotree.org (дата обращения: 15.05.2023).
Robinson P., Zemo jtel T. Integrative Genomics Viewer (IGV): Visualizing Alignments and Variants // In Computational Exome and Genome Analysis. New York, 2018. P. 233–245. https://doi.org/10.1201/9781315154770-17
Rogaev E.I., Grigorenko A.P., Moliaka Y.K. et al. Genomic Identification in the Historical Case of the Nicholas II Royal Family // Proceedings of the National Academy of Sciences of the United States of America. 2009. V. 106. № 13. P. 5258–5263.https://doi.org/10.1073/pnas.0811190106
Roostalu U., Kutuev I., Loogväli E.L. et al. Origin and Expansion of Haplogroup H, the Dominant Human Mitochondrial DNA Lineage in West Eurasia: The Near Eastern and Caucasian Perspective // Molecular Biology and Evolution. 2007. V. 24. Iss. 2. P. 436–448. https://doi.org/10.1093/molbev/msl173
Saag L., Vasilyev S.V., Varul L. et al. Genetic Ancestry Changes in Stone to Bronze Age Transition in the East European Plain // Science Advances. 2021. V. 7. № 4. P. 6535. https://doi.org/10.1126/sciadv.abd6535
Schubert M., Lindgreen S., Orlando L. AdapterRemoval v2: Rapid Adapter Trimming, Identification, and Read Merging // BMC Research Notes (BioMed Central Research Notes). 2016. V. 9. P. 88. https://doi.org/10.1186/s13104-016-1900-2
Stolarek I., Handschuh L., Juras A. et al. Goth Migration Induced Changes in the Matrilineal Genetic Structure of the Central-East European Population // Scientific Reports. 2019. V. 9. P. 6737. https://doi.org/10.1038/s41598-019-43183-w
Szeifert B., Gerber D., Csáky V. et al. Tracing Genetic Connections of Ancient Hungarians to the 6th–14th Century Populations of the Volga-Ural Region // Human Molecular Genetics. 2022. V. 31. № 19. P. 3266–3280.https://doi.org/10.1093/hmg/ddac106
Tassi F., Vai S., Ghirotto S. et al. Genome Diversity in the Neolithic Globular Amphorae Culture and the Spread of Indo-European Languages // Proceedings of the Royal Society. B: Biological Sciences. 2017. V. 284. Iss. 1867. P. 20171540. https://doi.org/10.1098/rspb.2017.1540
Van Oven M., Kayser M. Updated Comprehensive Phylogenetic Tree of Global Human Mitochondrial DNA Variation // Human Mutation. 2009. V. 30. № 2. P. e386–e394. https://doi.org/10.1002/humu.20921
Wang Ch.Ch., Posth C., Furtwängler A. et al. Genome-Wide Autosomal, MtDNA, and Y Chromosome Analysis of King Bela III of the Hungarian Arpad Dynasty // Scientific Reports. 2021. V. 11. P. 19210. https://doi.org/10.1038/s41598-021-98796-x
Weissensteiner H., Pacher D., Kloss-Brandstätter A. et al. HaploGrep 2: Mitochondrial Haplogroup Classification in the Era of High-Throughput Sequencing // Nucleic Acids Research. 2016. V. 44. № W1. P. W58–W63. https://doi.org/10.1093/nar/gkw233
YFull – MTree 1.02 [Электронный ресурс]. URL: https://www.yfull.com/mtree/ (дата обращения: 15.05.2023).
Дополнительные материалы отсутствуют.
Инструменты
Российская археология


