Прикладная биохимия и микробиология, 2023, T. 59, № 1, стр. 3-16
Сурфактин: биологическая активность и возможность применения в сельском хозяйстве (обзор)
О. В. Кисиль 1, *, В. С. Трефилов 2, В. С. Садыкова 1, М. Э. Зверева 2, Е. А. Кубарева 3
1 Научно-исследовательский институт по изысканию новых антибиотиков им. Г.Ф. Гаузе
119021 Москва, Россия
2 Химический факультет Московского государственного университета им. М.В. Ломоносова
119991 Москва, Россия
3 Научно-исследовательский институт физико-химической биологии им. А.Н. Белозерского
Московского государственного университета им. М.В. Ломоносова
119991 Москва, Россия
* E-mail: olvv@mail.ru
Поступила в редакцию 12.08.2022
После доработки 30.08.2022
Принята к публикации 02.09.2022
- EDN: CPUHKX
- DOI: 10.31857/S0555109923010026
Аннотация
Обобщена и проанализирована актуальная информация о сурфактине – циклическом липопептиде, одном из наиболее изученных микробных биосурфактантов. Представлены механизм биосинтеза сурфактина, спектр его природных и синтетических изоформ, биологическая активность сурфактина и его роль в регуляции процессов жизнедеятельности продуцентов. Продемонстрирован потенциал использования сурфактина и биопрепаратов, полученных на основе бактерий рода Bacillus – продуцентов сурфактина, для защиты и стимуляции иммунитета растений.
Стратегии микробного контроля c использованием антибиотиков постоянно сталкиваются с проблемой появления резистентных бактериальных штаммов, в том числе встроенных в биопленочные образования. В последние годы внимание исследователей приковано к бактериальным поверхностно-активным веществам – биосурфактантам – гетерогенной группе амфифильных соединений: с гидрофильными (аминокислотными или пептидными; ди- или полисахаридными; анионными или катионными) и гидрофобными (остатками насыщенных или ненасыщенных алифатических кислот) фрагментами, которые, непосредственно взаимодействуя с компонентами матрикса патогенных бактерий и грибов, изменяют физические свойства поверхности биопленки и инициируют ее деградацию. Было показано, что диспергирующие свойства биосурфактантов в отношении бактериальных и дрожжевых биопленок, а также мембранных структур вирусов не уступают таковым современных фармпрепаратов. Это делает их потенциальными кандидатами для использования в качестве антимикробных агентов нового поколения и/или в качестве адъювантов для других антибиотиков. Микробные биосурфактанты имеют ряд преимуществ в сравнении с синтетическими поверхностно-активными веществами (ПАВ): биоразлагаемость, низкая токсичность и физико-химическая стабильность в условиях высоких температур или экстремальных значений рН среды [1]. Некоторые биосурфактанты в настоящее время используются в клинической, пищевой, фармацевтической и экологической сферах, в то время как другие пока остаются на стадии изучения и разработки.
Сурфактин является наиболее изученным представителем биосурфактантов, продуцируемых Bacillus spp. [2]. В 1968 г. Арима с соавт. [3] в процессе исследований, посвященных изучению влияния микробных метаболитов на фибринолитическую систему и систему свертывания крови, обнаружили мощный ингибитор свертывания, секретируемый в культуральную жидкость несколькими штаммами Bacillus subtilis. Ингибитор был выделен в виде белых игольчатых кристаллов и из-за его высокой поверхностной активности, превышающей таковую для лаурилсульфата натрия, был назван “Сурфактин” (“Surfactin” от английского surface-active). В Японии он запатентован как мощный биосурфактант, ингибирующий образование тромбов [4]. На сегодняшний день установлено, что сурфактин продуцируется несколькими видами рода Bacillus, включая B. amyloliquefaciens, B. subtilis, B. pumilus, B. mojavensi, B. licheniformis, B. circulan, B. natto, B. tequilensis, B. inaquosorum, B. spizizenii, B. vallismortis B. subtilis subsp. subtilis и B. velezensis (The National Center for Biotechnology Information, https://www.ncbi.nlm.nih.gov) [5–7]. Исследование 35 штаммов дикого типа и коммерческих продуцентов, проведенное Хсе и соавт. [8], показало, что среди всех культур штаммы дикого типа B. amyloliqufaciens и B. subtilis отличаются наиболее высоким уровнем производства сурфактина – 452.5 и 125.6 мг/л соответственно.
Сурфактин является амфифильной молекулой, что обуславливает его уникальные физико-химические свойства: способность к пенообразованию, эмульгированию, модификации гидрофобных поверхностей и хелатированию [9, 10]. Он может диспергировать нефтяные разливы на поверхности воды, увеличивая эффективность их утилизации аборигенными морскими микроорганизмами, а также улучшать адгезию бактерий к нефтяному пятну для повышения продуктивности биоремедиации [11]. Эмульгирующие свойства сурфактина предполагают возможность применения его в косметической промышленности и бифармацевтике [12]. Показано, что сурфактин индуцирует цитотоксичность в отношении клеточных линий многих типов злокачественных образований, таких как рак груди и толстой кишки, лейкемия и гепатома [2]. Амфифильная природа сурфактина позволяет легко вводить его в нанопрепараты (полимерные наночастицы, мицеллы, микроэмульсии, липосомы), что позволяет оптимизировать доставку сурфактина в злокачественные образования и повысить эффективность противоопухолевой терапии. К сожалению, коммерческий потенциал сурфактина в качестве терапевтического агента не может быть полностью реализован из-за его гематотоксичности, выражающейся в гемолизе эритроцитов.
Структурное разнообразие сурфактина. Первоначально структура сурфактина была установлена путем гидролиза молекулы на фрагменты с последующей их идентификацией и определением порядка следования сначала по аминокислотной последовательности, а затем по цепи алифатических кислот [13, 14]. Сурфактин состоит из замкнутой пептидной цепи, образованной семью остатками α-аминокислот, соединенной с остатком β-гидроксиалифатической кислоты. Типичная последовательность аминокислотных остатков в циклопептидном фрагменте (рис. 1а) следующая: L-Glu1-L-Leu2-D-Leu3-L-Val4-L-Asp5-D-Leu6-L-Leu7 [15]. Отметим, что в положениях 3 и 6 находятся аминокислотные остатки в D-форме. Полярная часть сурфактина состоит из двух отрицательно заряженных аминокислотных остатков Glu и Asp (в природном сурфактине).
Рис. 1.
Сурфактин, структура и взаимодействие с мембраной клетки: а – структурная формула сурфактина; б – предполагаемое строение мицеллы, образованной из молекул сурфактина (адаптировано из [2]); в – схематическая модель дестабилизации фосфолипидной мембраны (липиды показаны серым цветом) молекулами сурфактина (черный цвет), адаптировано из [16].
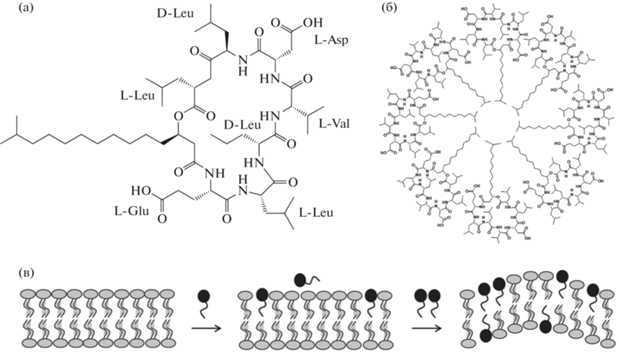
Изучение трехмерной структуры сурфактина методом 1H ЯМР показало наличие минорного полярного и основного гидрофобного доменов. Минорный домен образован основной цепью циклического пептида и двумя его отрицательно заряженными аминокислотными остатками – Glu1 и Asp5. Гидрофобный домен образован углеводородной цепью остатка жирной кислоты и аминокислотами Leu2, Leu3, Val4, Leu6, Leu7. Такая двудоменная структура обуславливает амфифильный характер сурфактина и, как следствие, высокую поверхностную активность [15]. Основываясь на данных спектроскопии кругового дихроизма и инфракрасной спектроскопии с преобразованием Фурье, Васс с соавт. [17] показали, что сурфактин в растворе обладает значительной способностью к самосборке с образованием мицелл и более крупных агрегатов, при этом на морфологию образующихся мицелл заметно влияют условия окружающей среды, такие как рН, ионы металлов и температура (рис. 1б).
Относительная сложность и вариабельность структуры определяет сосуществование большого количества изомеров сурфактина в образцах, выделяемых из культуральной жидкости бактерий [18]. Из-за широкого круга изомеров молекулярная масса сурфактинов варьируется в диапазоне 993–1049 Да [5]. Основные структурные аналоги сурфактина можно разделить на 2 группы: (1) изомеры, отличающиеся аминокислотной последовательностью гидрофильной “головы” и (2) изомеры, отличающиеся количеством атомов углерода в алифатической части жирной кислоты, то есть длиной “хвоста”. Компьютерный анализ кластеров генов вторичных метаболитов показал, что большинство вариантов сурфактина, кодируемых в геномах представителей рода Bacillus, различаются по положению 1 и 7 пептидного кольца. Гены биосинтеза сурфактина из B. subtilis и B. amyloliquefaciens – в положении 7 Leu, из B. atrophaeus – Ile, а из B. licheniformis – кодируют Gln и Ile в положениях 1 и 7 соответственно [19]. Были идентифицированы природные изоформы сурфактина с вариациями гидрофобных аминокислотных остатков в положениях 2 (Leu меняется на Val или Ile) и 4 (Val меняется на Leu, Ala или Ile) [5]. Гидрофильный остаток аспарагиновый кислоты в положении 5 может подвергаться модификации: он метилируется и превращается в относительно гидрофобный 4-метиловый эфир аспарагиновой кислоты [20]. Предположительной причиной такого широкого спектра вариаций могут быть аминокислотные замены в доменах аденилирования субъединиц сурфактинсинтетазы (см. раздел “Биосинтез сурфактина”) [21].
Длина гидрофобного “хвоста” сурфактина также подвержена изменению: она может составлять от 13 до 18 атомов углерода. Основные изоформы имеют 14 и 15 атомов углерода [5]. Варьироваться может не только длина углеродной цепи, но и ее разветвленность. Показано существование изомеров изо-C12, изо-C13, антеизо-C13, изо-C14, н-C14, изо-C15, н-C15, антеизо-C15, антеизо-C16 и антеизо-C17 [22].
На соотношение различных изомеров в синтезируемом бактериями сурфактине можно влиять путем изменения условий культивирования, а именно добавления в питательную среду различных низкомолекулярных соединений. Бартал и соавт. [5] показали, что изоформный состав сурфактина зависит от источника углерода в питательной среде, при этом наибольший эффект наблюдался при использовании фруктозы и ксилозы. Введение в культуральную жидкость ионов металлов Mn2+, Cu2+ и Ni2+ приводит к образованию как новых метилированных по аспартату форм сурфактина, так и изомеров с более длинными цепями жирных кислот: две трети молекул были гомологами С16, С17 или С18. Смежные исследования в этой области показали, что присутствие аминокислот в питательной среде также влияет на изомерный состав синтезируемого сурфактина [22]. Так, при добавлении в питательную среду B. subtilis TD7 аминокислот Arg, Gln или Val возрастает доля изомеров сурфактина с четной длиной гидрофобного “хвоста”, тогда как добавление Cys, His, Ile, Leu, Met, Ser, или Thr способствует увеличению доли изомеров с нечётной длиной “хвоста”.
Взаимосвязь структура – активность сурфактина. Различия в структуре – длине и строении углеводородного “хвоста”, а также аминокислотном составе – оказывают влияние на биологическую активность сурфактинов. Показано, что с увеличением длины цепи поверхностная и межфазная активность сурфактинов увеличивается, поэтому липопептид с короткой углеводородной цепью имеет тенденцию к образованию небольших мицелл, в то время как при увеличении длины цепи наблюдается тенденция к укрупнению мицелл и их агрегированию (рис. 1б) [15]. Степень проникновения сурфактина в фосфолипидную мембрану клетки прямо пропорциональна длине углеводородной цепи. Показано, что сурфактин с “хвостом” С15 обладает большей противоопухолевой активностью, чем с углеводородной цепью С13 и С14 [23]. Аналогичные данные были получены и при инактивации сурфактином оболочечных вирусов. Сурфактин С13 показал очень низкую противовирусную активность по сравнению с изоформами С14 и С15 [24]. К сожалению, с увеличением гидрофобности жирных кислот (длины “хвоста”) увеличивалась не только противовирусная активность, но и гемолитическое действие сурфактина.
Критичной для адсорбции сурфактина на липидной поверхности и его проникновения в липидную пленку оказывается и цикличность пептидного фрагмента. Показано, что линейный сурфактин, полученный путем химического расщепления цикла, обладает меньшей поверхностной активностью, чем природный сурфактин [25]. Что же касается роли природы аминокислотных остатков в биологической активности сурфактина, результаты экспериментов показали, что поверхностная активность повышается, а критическая концентрация мицеллообразования снижается при замене аминокислот на более гидрофобные [15]. Значимую роль в амфифильных свойствах сурфактина играют два отрицательно заряженных остатка Glu и Asp. Показано, что природный метилированый по Glu сурфактин С15 (С15-сурфактин-О-метиловый эфир) обладает более высокой поверхностной активностью и меньшим противоопухолевым действием, чем С15-сурфактин [26]. Если остатки Glu или Asp метилированы или амидированы, то активность сурфактина по снижению поверхностного натяжения увеличивается на 20%, несмотря на то, что растворимость липопептида в воде при этом уменьшается. При модификации остатков Glu1 и Asp5 аминометансульфоновой кислотой поверхностная активность сурфактина резко снижалась из-за электростатических и стерических факторов [26].
Исследования показывают, что структуру сурфактина можно модифицировать для усиления его желательных и ослабления нежелательных эффектов. Одним из основных недостатков использования сурфактина в качестве противоопухолевого и антибактериального препарата является его гемолитическая активность. Для ее преодоления, были разработаны линейные формы сурфактина, для которых, в отличие от циклических форм, не наблюдается значительного гемолиза [28]. Варианты сурфактина, у которых в кольце отсутствуют остатки Leu3 или Leu6, также демонстрируют пониженную гемолитическую активность. В то же время сурфактин без Asp5 сохраняет гемолитические свойства, но демонстрирует лучшие, по сравнению с природным сурфактином, антибактериальные свойства в отношении Staphylococcus aureus и Bacillus cereus. Минимальная ингибирующая концентрация (МИК), подавляющая развитие данных патогенов, составила 25 мкг/мл для сурфактина без Asp5 и 50 мкг/мл для нативного сурфактина [29].
Биосинтез сурфактина. Известно, что вторичный метаболический аппарат бактерий организован в кластеры генов, внутри которых в непосредственной близости друг от друга располагаются гены, отвечающие за биосинтез продуцируемых бактериями соединений [30]. Подобный кластер для сурфактина был описан параллельно разными группами ученых [31]. В базе данных (Minimum Information about a Biosynthetic Gene cluster, http://mibig.secondarymetabolites.org) зарегистрирован кластер генов биосинтеза сурфактина для Bacillus velezensis FZB42 (идентификатор MiBIG: BG0000433) [32].
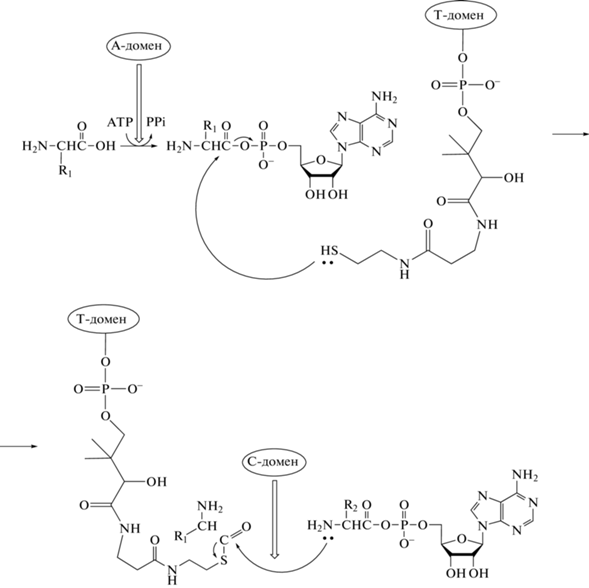
Биосинтез сурфактина, как и большинства других циклических липопептидов, осуществляется по нерибосомальному механизму с помощью специальных систем – нерибосомальных пептидных синтетаз (НРПС). НРПС представляют собой мультиферментные комплексы, которые условно можно разделить на модули – участки НРПС, отвечающие за встраивание определенной аминокислоты в пептидную цепь [2, 6]. В свою очередь, каждый модуль может быть разделен на несколько доменов: домен аденилирования (А), домен тиолирования (Т) и домен конденсации (С). А-домен отвечает за узнавание и активацию конкретной аминокислоты за счет образования аминоациладенилата (процесс аналогичен активации карбоксильной группы аминокислоты тРНК-синтетазой при рибосомальном пептидном синтезе). Далее подвижная и гибкая 4'-фосфопантетеиниловая часть Т-домена образует тиоэфирную связь с карбоксильной группой аминокислоты, и за счет своей относительно большой длины (20 Å) переносит аминокислотный остаток из активного центра Т-домена к С-домену. С-домен, в свою очередь, катализирует образование новой пептидной связи и перемещение удлиненного пептида к следующему модулю (рис. 2) [31]. Линейное расположение нескольких таких модулей в виде сборочной линии обеспечивает координированное удлинение пептидной цепи. После модулей 3 и 6 дополнительно имеются домены эпимеризации (Е-домены), отвечающие за изомеризацию остатков природного L-Leu, связанного с Т-доменом в 3 и 6 модулях, в D-изомеры, так как молекула сурфактина содержит именно D-Leu в этих положениях. В большинстве случаев нерибосомальный синтез пептида завершается макроциклизацией, при этом части молекулы, удаленные в построенной линейной пептидной цепи, ковалентно связываются друг с другом. Процесс циклизации чаще всего катализируется тиоэстеразными доменами на С-конце сборочной линии НРПС. Остаток β-гидроксиалифатической кислоты, содержащийся в сурфактинах и других микробных липопептидах, встраивается в молекулу в начале биосинтеза. В стартовом модуле НРПС на N-конце имеется дополнительный домен (C*), отвечающий за образование связи между CoA-активированным остатком β-гидроксиалифатической кислоты с первой аминокислотой.
Рис. 2.
Схема реакции биосинтеза сурфактина одним из модулей нерибосомальных пептидных синтетаз. R1 и R2 – боковые цепи аминокислотных остатков.
Гены, кодирующие субъединицы НРПС сурфактина – srfAA, srfAB, srfAC и srfAD – объединены в единый оперон srfA (рис. 3). Белки SrfAA и SrfAB состоят из трех модулей, а SrfAC – из одного модуля и тиоэстеразного (ТЕ) домена, отвечающего, как упоминалось выше, за высвобождение гептапептидной цепи и ее макроциклизацию. Кроме того, субъединица SrfAA содержит на N-конце также уже упоминавшийся дополнительный домен, катализирующий присоединение к первой аминокислоте (в случае сурфактина это чаще всего Glu) липофильного “хвоста”, остатка β-гидроксиалифатической кислоты. SrfAD представляет собой ТЕ-домен и отвечает за регенерацию ошибочно связанных Т-доменов в модулях первых трех субъединиц [31].
Рис. 3.
Кластер генов белков, ответственных за биосинтез сурфактина (а) и классическая схема сборочной линии сурфактина (б). Гены субъединиц НРПС выделены черным; М – модуль сборочной линии; остальные гены кластера обозначены серым. Масштаб размера генов кластера приведен в тысячах п.н. Регуляторный ген comS транскрибируется совместно с srfAB; (адаптировано из [31]).
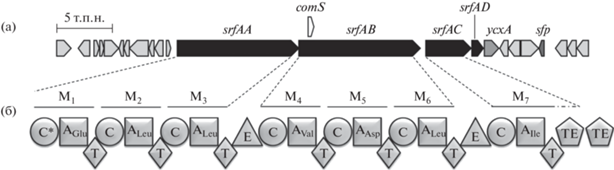
Все 4 гена, кодирующие НРПС сурфактина, включены в один оперон srfA, транскрипция которого контролируется промотором PsrfA. Активность PsrfA, в свою очередь, регулируется транскрипционным фактором ComA, являющимся частью двукомпонентной системы ComP/ComA. Когда концентрация бактериальных клеток B. subtilis достигает определенного значения, мембранная гистидиновая киназа ComP фосфорилирует белок ComA, в результате он активируется и индуцирует транскрипцию оперона srfA, инициируя биосинтез сурфактина. В работе [33] промотор PsrfA, активность которого, из-за зависимости от концентрации клеток в окружающей среде, непостоянна во времени, был заменен на нативный конститутивный промотор Pveg, содержащийся в геноме B. subtilis, с целью повышения количества синтезируемого сурфактина. Оказалось, что подобная замена приводит к повышению синтеза сурфактина только в малоэффективных штаммах-продуцентах, а в штаммах с повышенным природным производством сурфактина, его выход падает. В другой работе группа ученых заменила промотор PsrfA в штамме-продуценте B. subtilis THY-7 на другой “сильный” природный для штамма B. subtilis THY-7 промотор PgroE, однако получила тот же результат – производство сурфактина в модифицированном штамме заметно уменьшилось. Вместе с тем, при замене PsrfA на искусственный промотор Pg3, индуцируемый экзогенно изопропил-β-D-1-тиогалактопиранозидом (ИПТГ), производство сурфактина возросло в 18 раз по сравнению с нативным штаммом [34].
Помимо структурных генов сурфактинсинтетазы кластер биосинтетических генов сурфактина включает один встроенный и несколько смежных дополнительных генов, кодирующих транспортеры и регуляторные белки (рис 3, верхняя панель, серые стрелки) [32]. Среди них следует отдельно выделить гены sfp, ycxA, krsE, yerP и comS, транскрипция которых напрямую связана с эффективностью производства сурфактина. Особого внимания заслуживает ген sfp, кодирующий фосфопантетеинилтрансферазу – фермент, катализирующий перенос остатка фосфопантетеинила на неактивный Т-домен субъединиц НРПС и тем самым активирующий их [35]. Показано, что наличие интактного гена sfp в геноме бактерии строго необходимо для производства сурфактина. Так, штамм Bacillus subtilis 168, который систематически использовался в качестве модельной системы для грамположительных организмов, не производит сурфактин, при этом гены оперона srfA идентичны генам в штаммах-продуцентах сурфактина. Выяснилось, что у B. subtilis 168 вследствие мутации в гене sfp появился один лишний нуклеотид (А в позиции 634), что вызвало инактивацию белка Sfp и блокирование синтеза сурфактина. При этом его производство восстанавливается при внесении в штамм B. subtilis 168 вектора, содержащего интактный ген sfp, выделенный из штамма-продуцента [36]. Немаловажную роль как в процессе биосинтеза сурфактина, так и в механизме устойчивости клеток-продуцентов к нему, играет система экспорта сурфактина через мембрану, позволяющая избегать внутриклеточного накопления сурфактина. Были идентифицированы три гена, которые участвуют в оттоке сурфактина: ycxA, krsE, и yerP [37]. Показано, что основным экспортером является белок YerP, повышение экспрессии которого увеличивает количество сурфактина в культуральной жидкости на 145% [37].
Ген comS, расположенный внутри оперона srfA в открытой рамке считывания гена srfАB, кодирует белок ComS, имеющий несколько функций. С одной стороны, ComS участвует в положительной регуляции генетической компетентности клетки (способность захватывать экзогенный генетический материал и ассимилировать его), а с другой стороны, является частью системы comQXPA, отвечающей за распознавание окружающих клетку родственных бактерий (“quorum sensing”) и регулирующей биосинтез сурфактина [33, 38].
Биологическая активность сурфактина. Сурфактин как поверхностно-активное вещество. Биосурфактанты, обладая свойствами ПАВ, могут изменять условия на границах раздела двух фаз, различающихся полярностью и количеством водородных связей (например, вода/масло или вода/воздух) [2]. Существует два предполагаемых механизма влияния биосурфактантов на жизнеспособность клеток-продуцентов: (1) повышение способности к эмульгированию, что, в свою очередь, увеличивает доступность гидрофобных соединений в качестве источника питательных веществ и (2) помощь в прикреплении и отделении микроорганизмов от поверхностей [6]. Сурфактин известен как один из самых мощных ПАВ. Он снижает поверхностное натяжение воды с 72 до 27 мН/м при концентрации всего 10 мкМ, что намного ниже его критической концентрации мицеллообразования в воде (23 мг/л) и примерно на два порядка меньше, чем для большинства моющих средств [39]. Мозаичное распределение полярности и разветвленная кольцевая структура позволяют сурфактину принимать сферическую, мицеллярную структуру для облегчения плотной упаковки на границах раздела фаз (рис. 1б). В среднем масса мицеллы 179 000, число агрегации n = 173 [40]. Липидный “хвост”, свободно двигающийся в растворе, активно участвует в гидрофобных взаимодействиях в супрамолекулярных структурах на границе раздела вода/ воздух. Молекулы сурфактина на границе раздела фаз очень тесно выстроены, при этом адсорбционные свойства сурфактина зависят не только от концентрации, но и от длины углеводородной цепи [15].
Сурфактин в “quorum sensing”. Бактериальные сообщества могут чувствовать изменения окружающей среды и соответствующим образом корректировать свое поведение с помощью системы распознавания кворума. Этот процесс зависит от плотности клеток и регулируется сигнальными молекулами – аутоиндукторами. Концентрация последних повышается во внеклеточной среде по мере того, как увеличивается плотность бактериальных клеток, и, когда она достигает определенного порогового уровня, группа клеток начинает действовать синхронно. Сигнальные молекулы приводят к дифференцировке клеток в различные типы для адаптации к изменениям окружающей среды. Например, при неблагоприятных условиях сурфактин инициирует развитие субпопуляции B. subtilis, известной как каннибалы. Они выделяют особые токсины, которые разрушают соседние клетки, при этом сами клетки-каннибалы к токсинам невосприимчивы. Предполагается, что сурфактин является сигнальной молекулой в микробной коммуникации и участвует в активации гистидинкиназы сенсорного рецептора, ассоциированной с мембраной (KinC). В свою очередь, KinC активирует экспрессию гена белка раннего спорообразования Spo0A, что в конечном итоге активирует споруляцию, образование биопленок и колонизацию корней растений [41].
Сурфактин и дестабилизация биологических мембран. Биологическая активность сурфактина связана с его взаимодействием с липидной частью биологических мембран всех видов бактерий. Благодаря своей амфифильной структуре, молекулы сурфактина могут легко встраиваться в липидные слои и прочно закрепляться там: гидрофобная часть сурфактина взаимодействует с углеводородными цепями фосфолипидов мембраны, а пептидная часть – с полярными группами липидов (рис. 1в). Проникновение сурфактина в фосфолипидный бислой приводит к нарушениям и/или созданию каналов в цитоплазматической мембране, что, в свою очередь, может активировать каскад молекулярных событий, приводящих к защитным реакциям [23, 42 ]. Сурфактин дестабилизирует мембрану с помощью нескольких механизмов: (1) встраивание в липидные бислои, (2) образование каналов/пор или диффузия одно- и двухзарядных ионов через мембранный барьер и (3) солюбилизация мембраны подобно действию детергента. Какой именно механизм будет задействован, зависит от концентрации сурфактина [43]. Показано, что при низкой концентрации антимикробные пептиды склонны проникать в мембрану и встраиваться в липидный бислой [44]. Это вызывает образование однослойных везикул на внешней мембране, приводя к деформации клетки и, в конечном итоге, к ее гибели (рис. 1в) [45].
С увеличением концентрации сурфактин начинает образовывать агрегаты в липидном бислое, создавая поры в клеточной мембране. Образовавшиеся поры дают возможность для выхода нуклеиновых кислот, незаменимых ионов и АТФ из клетки, приводя к ее гибели. Показано, что ионы Са2+ помогают более глубокому проникновению сурфактина в мембрану за счет нейтрализации зарядов как сурфактина (экранирование отрицательных зарядов остатков Glu1 и Asp5 пептидного цикла), так и заряженных частей липидов [46, 47]. При высоких концентрациях преобладает детергентый механизм разрушения мембраны, основой которого является солюбилизация фосфолипидов молекулами сурфактина, при этом степень проникновения сурфактина прямо пропорциональна длине “хвоста”. Лю с соавт. [48] показали, что сурфактин ингибирует образование биопленки Staphylococcus aureus за счет снижения процентного содержания растворимых в щелочной среде полисахаридов и подавления экспрессии генов ica A и icaD, участвующих в формировании биопленки.
Сурфактин в образовании биопленок и колонизация корней растений. B. subtilis — это подвижная грамположительная спорообразующая факультативная аэробная почвенная бактерия. Для рода Bacillus миграция клеток по поверхности сред, роение, а также способность к колонизации корней растений путем образования на их поверхности биопленок напрямую связаны с производством сурфактина [6, 49]. В неподвижной субпопуляции B. subtilis сурфактин посредством взаимодействия с KinC активирует транскрипцию гена белка Spo0A, что приводит к изменению моторно-роторных жгутиковых механизмов [38, 50]. Сурфактин способствует смачивоемости гидрофобной кутикулы растения, что способствует не только увеличению подвижности бактериальных клеток, но также растворимости и диффузии субстратов для роста. Показано, что сурфактин может изменять вязкость поверхностей, тем самым влияя на подвижность клеток [51, 52]. Анализ генома штамма B. amyloliquefaciens subsp. plantarum FZB42, коммерчески используемого в качестве биоудобрения в сельском хозяйстве и представляющего собой модельную бактерию для изучения взаимодействий с растениями, показал, что почти 10% генома этого продуцента связано с синтезом антимикробных метаболитов [53]. При этом исследования на растениях демонстрируют, что, за исключением сурфактина, количество антимикробных метаболитов, обнаруживаемых вблизи корней растений, относительно невелико. Таким образом, именно сурфактины позволяют колониям бактерий р. Bacillus образовывать биопленки [54].
Бактерии инициируют образование биопленки в ответ на определенные сигналы окружающей среды, такие как доступность питательных веществ и кислорода. При переходе от свободноживущих организмов к неподвижным колониям в биопленке они претерпевают динамические изменения, включая специфическую продукцию вторичных метаболитов и значительное повышение устойчивости к биологическим, химическим и физическим воздействиям. Опыт успешного использования сурфактина для повышения устойчивость растений к патогенам в лабораторных условиях суммирован в табл. 1. Известно, что сурфактин влияет на способность B. subtilis стимулировать развитие растений через формирование биопленки [55]. Териен с соавт. [56] показали, что продукция сурфактина не является принципиальной для формирования биопленки, но отсутствие сурфактина снижает разрастание колоний B. subtilis .
Таблица 1.
Использование сурфактина для повышения устойчивости растений к патогенам
| Штамм | Действующий липопептид | Объект защиты | Способ обработки | Патоген | Ссылка |
|---|---|---|---|---|---|
| Защита растения от патогена вследствие прямого антимикробного действия | |||||
| B. subtilis RB14 | Смесь липопептидов: итурина и сурфактина | Томат (листья) | Инокуляция почвы до посадки проросших семян, обработка растений | Rhizoctonia solani | [57] |
| B. subtilis GA1 | Смесь липопептидов: итурина, фенгицина и сурфактина | Яблоки (плоды) | Обработка плодов | Botrytis cinerea | [58] |
| B. subtilis 6051 | Сурфактин | Арабидопсис (корни) | Инокуляция корней и почвенного грунта | Pseudomonas syringae pv tomato | [59] |
| B. subtilis UMAF6614 | Смесь липопептидов: бацилломицина, фенгицина и сурфактина. (Показано, что сурфактин основной детерминатор атипатогенного ответа) | Дыня (листья) | Опрыскивание листьев | Podosphaera fusca, Pectobacterium carotovorum subsp. carotovorum,Xanthomonas campestris pv. cucurbitae | [60] |
| B. subtilis 916 | Смесь изоформ сурфактина от C13 до C15 | Рис (стебель, листья) | Опрыскивание стеблей и листьев | Rhizoctonia solani | [61] |
| B. atrophaeus 176s, B. atrophaeus 1942, B. subtilis OKB105, B. amyloliquefaciens FZB42 | Сурфактины А, В и С. Смесь изоформ от C13 до C16 | Помидоры (корни), салат (корни), сахарная свекла (корни) | Обработка корней | Rhizoctonia solani | [21] |
| B. subtilis 9407 | Сурфактин А. Смесь изоформ от С13 до С16 | Дыня (листья) | Замачивание проросших семян | Acidovorax citrulli | [62] |
| B. velezensis Bvel1 | Смесь липопептидов: итурина A2, изоформ сурфактина C13 и C15, оксидиффицидина, бациллибактина, L-дигидроантикапсина | Виноград (грозди) | Обработка ягод | Botrytis cinerea | [63] |
| Стимулирование индуцированной системной устойчивости растений к заболеваниям | |||||
| B. subtilis 2500, B. subtilis 2508 | Смесь изоформ от C13 до C15 | Фасоль (листья), томат (листья) | Обработка семян. Добавление бактериальной суспензии в почву и прикорневая поливка через неделю | Botrytis cinerea | [42] |
| B. subtilis UMAF6639 | Смесь липопептидов, включая сурфактин. Показано, что сурфактин является основной детерминантой стимуляции иммунного ответа | Тыква (листья) | Инокуляция корней и прикорневая поливка через неделю | Podosphaera fusca | [64] |
| B. amylolequifaciens S499 | Смесь липопептидов: итурина, фенгицина и сурфактина | Сахарная свекла (корни) | Полив растений | Polymyxa betae | [65] |
| B. amyloliquefaciens S499, B. amyloliquefaciens FZB42, B. amyloliquefaciens QST713, B. subtilis 98S | Сурфактин | Томат (листья), табак (листья) |
Добавление сурфактина в гидропонный раствор | Botrytis cinerea | [66] |
| B. subtilis ABS-S14 | Сурфактин. (Показан синергизм смеси липопептидов: итурина, фенгицина и сурфактина) |
Апельсин (плоды) | Обработка плодов | Penicillium digitatum | [67] |
| B. amyloliquefaciens FZB42-AK3 | Смесь изоформ сурфактина от C12 до C15 | Пастбищный райграс (листья) | Замачивание семян в суспензии бактериальных клеток/ растворе сурфактина. Добавление бактериального инокулята/ раствора сурфактина в почву и прикорневая поливка через 3 недели | Magnaporthe oryzae | [68] |
| B. amyloliquefaciens S13-3 | Смесь липопептидов, включая сурфактин Коммерческий препарат сурфактина |
Земляника (листья) | Опрыскивание листьев | Colletotrichum gloeosporioides | [69] |
| B. subtilis | Коммерческий препарат сурфактина | Арахис (стебель) | Погружение корней проростков в раствор сурфактина | Sclerotium rolfsii | [70] |
| B. subtilis 26Д | Сурфактин | Пшеница (листья) | Замачивание семян | Septoria nodorum | [71] |
| B. subtilis GLB19.1 | Смесь липопептидов: сурфактина и фенгицина |
Виноград (листья) | Опрыскивание листьев | Plasmopara viticola | [72] |
| B. velezensis BBC023, B. velezensis BBC047 | Смесь изоформ сурфактина от C12 до C17 | Томат (корни, листья) | Инокуляция поверхности корней или листьев | Botrytis cinerea | [49] |
Базис с соавт. [59] продемонстрировали способность сурфактина из B. subtilis колонизировать корни арабидопсиса с образованием стабильной биопленки и, таким образом, защищать их от Pseudomonas syringae как in vitro, так и в почве МИК сурфактина в отношении P. syringae составила 25 мкг/мл, что является относительно высоким для противомикробного агента, но приемлемо для коммерческого биопестицида. В экспериментах с корнями Arabidopsis, которые были предварительно инокулированы суспензией B. subtilis, уровни сурфактина в промытых корнях были значительными: 151.6 мкг/мл на 50 мг сырого веса корней. Возможно, что на поверхности корня концентрация растворенного сурфактина значительно выше, чем МИК против P. syringae, определенная in vitro. Интересно, что после инокуляции патогенным штаммом P. syringae продукция сурфактина увеличивалась примерно в два раза. Авторы протестировали мутантный штамм с делецией в гене sfp, дефицитный по продукции сурфактина. Он был неэффективен в качестве средства биологической борьбы с P. syringae и не образовывал прочных биопленок ни на корнях арабибопсиса, ни на инертных поверхностях. Аналогичные результаты были получены и Луо с соавт. [61], которые получили мутантные штаммы B. subtilis 916 с делециями в гене первой субъединицы НРПС сурфактина srfАA.
B. subtilis 916 является ключевым компонентом популярного в Китае коммерчески доступного биофунгицида “Wenquning”. Мутантные дефицитные по продукции сурфактина штаммы продемонстрировали изменение роевой подвижности, снижение антагонистической активности и уменьшение эффективности образования биопленки [61]. При этом роевая подвижность восстанавливалась при добавлении сурфактина в дозе 10 мкг/мл, в то время как образование биопленок не удалось восстановить даже при добавлении дозы 50 мкг/мл. В работе Фан с соавт. [62] мутантный штамм B. subtilis, со сниженной выработкой сурфактина, показал уменьшение образование биопленки, снижение роевой подвижности и не проявлял способности ингибировать рост Acidovorax citrulli [62].
Изоформы сурфактина видоспецифичны и опосредуют конкретную для каждого вида передачу сигналов, приводящую к различному экологическому поведению. Мутантный штамм B. atrophaeus со сниженной выработкой сурфактина реагировал на экзогенный родственный сурфактин C, полученный из нативного штамма, образуя прочные биопленки, в то время как в присутствии сурфактина A, производимого B. subtilis, наблюдалась только разреженная биопленка [21]. Аналогично, родственный сурфактин А вызывал устойчивые биопленки у дефицитного по сурфактину штамму B. subtilis, в то время как сурфактин C, полученный из B. atrophaeus, индуцировал у B. subtilis разреженные биопленки.
В работе Дэбоиз с соавт. [73] показано, что сурфактин является основным бактериальным метаболитом, накапливающимся в растениях в течение первых часов взаимодействия бактерии с корнями растения. Cинтез сурфактина специфически стимулируется при взаимодействии бактерий с полимерами клеточных стенок растения: ксиланом или арабиногалактаном, что приводит к быстрому накоплению микромолярных количеств липопептида в корневой системе. При таких концентрациях сурфактин не только усиливает способность штамма-продуцента колонизировать корни, но и активирует индуцированную системную устойчивость растения-хозяина.
Сурфактин и индуцированная системная устойчивость растений. Помимо прямого антагонизма, некоторые бактерии могут защищать растения косвенно, стимулируя индуцируемые защитные механизмы, которые делают растение-хозяина более устойчивым к проникновению патогена. Такая индуцируемая защита проявляется на протяжении всего онтогенеза и по своей природе близка к естественным иммунным реакциям. Описываемое явление получило название индуцированной системной устойчивости (induced systemic resistance, ISR). Считается, что сурфактин не вызывает защитного ответа растений, связанного с существенным генетическим перепрограммированием и приспособлением, а скорее включает защитные механизмы хозяина для инициации системной устойчивости [73]. Потенциал сурфактина вызывать системную устойчивость растений был впервые показан на образцах фасоли и томата [42]. Применение липопептида в чистом виде в микромолярных концентрациях привело к значительному снижению заболеваемости растений, инфицированных Botrytis cinerea. Кавой с соавт. [66] было проведено исследование с большим количеством природных изолятов рода Bacillus, различающихся по способности продуцировать липопептиды в заданных условиях in vitro. Некоторые изоляты не продуцировали ни одного липопептида, другие продуцировали два или все три семейства липопетидов, включая итурины, фенгицины и сурфактины в разных пропорциях. Наблюдалась сильная корреляция между защитной активностью бактериальных экстрактов против Botrytis cinerea и количеством продуцируемого ими сурфактина.
Воздействием на растения в первые 15–20 сут онтогенеза или до посева путем обработки семян определенными биологически активными веществами, можно индуцировать изменение их метаболизма в сторону, неблагоприятную для патогенов [74]. Замачивание семян пастбищного райграса в суспензии бактериальных клеток B. amyloliquefaciens, продуцирующих сурфактин, привело к ативации индуцированной системной устойчивости растений против Magnaporthe oryzae [68].
Основой коммерческого препарата “Фитоспорин-М”, широко применяемого в России и содержащего бактерии B. subtilis, является штамм B. subtilis 26Д, продуцирующий сурфактин. В геноме штамма B. subtilis 26Д были обнаружены гены, кодирующие субъединицы сурфактинсинтетазы, при этом не было обнаружено генов, ответственных за синтез итуринсинтетазы, и фенгицинсинтетазы [71]. Авторы показали, что сурфактин из B. subtilis 26Д защищает растение от септориоза злаков, причем не только за счет прямого фунгицидного действия, но и опосредованно – через усиление экспрессии генов защитных белков растений и активации у растений-хозяев иммунного потенциала. Обработка корней пастбищного райграса сурфактином и суспензией клеток B. amyloliquefaciens FZB42-AK3 (штамм продуцирует только сурфактин, но не противогрибковые соединения бацилломицин D и фенгицин) значительно снижало заболеваемость растений M. oryzae [68]. Показано, что сурфактин вызывает многоуровневую активацию ISR у райграса за счет усиленного накопления перекиси водорода в корнях растений и протекания последующих H2O2-опосредованных защитных реакций. Наблюдалось быстрое повышение активности пероксидазы в межклеточной жидкости обработанных растений. Показано отложение каллозы и фенольных соединений на листовых пластинках райграса в местах заражения M. oryzae. H2O2-зависимая, опосредованная пероксидазой, взаимосвязь локализованных в клеточной стенке фенольных соединений играет значимую роль в укреплении клеточных стенок растений и ограничении проникновения патогенов. В работе Онгена с соавт. [42] инициация сурфактином ISR приводила к увеличению активности всех ферментов, разлагающих гидропероксиды. В работе Родригеса с соавт. [70] пероксидазная активность и отложение фенольных соединений под зоной грибковой инфекции были значительно выше у зараженных образцов арахиса, предварительно обработанных сурфакином, по сравнению с необработанными сурфакином образцами.
Точный механизм непосредственного действия сурфактина против бактериальных патогенов пока неизвестен. Опубликованные на настоящий день данные показывают, что ингибирующий эффект сурфактина не связан с его прямым воздействием на жизнеспособность клеток-мишеней, а обусловлен вмешательством в ключевые процессы развития патогена такие как образование биопленки Pseudomonas syringae [59] или развитие воздушных гиф, как показано для свободноживущей почвенной бактерии Streptomyces coelicolor [75]. При этом сурфактин не ингибирует рост субстратных гиф S. coelicolor, что можно было бы ожидать, если бы он действовал как антибиотик.
Почти все антибиотики, повышающие устойчивость растения к патогенам, глубоко проникают в ткани растений, увеличивая риск пагубного влияния плодов этого растения на человека. Однако сурфактин не распространяется в тканях растений. В работе Онгена с соавт. [42] живые клетки изолята, принадлежащего роду Bacillus, не были обнаружены в образцах листьев фасоли и томата после обработки корневой системы бактериальной суспензией, что свидетельствует о том, что бактерии не мигрируют в межклеточное пространство растений. Таким образом, для сурфактина ингибирование развития фитопатогенов происходит за счет индукции резистентности в растении-хозяине, поскольку бациллы и патоген остаются локализованными на разных частях растения. При этом бактериальная популяция на корнях находится в пределах концентраций, необходимых для инициирования индуцированной системной устойчивости.
В последние годы исследования возможности применения в сельском хозяйстве аэробных эндоспорообразующих бактерий привели к разработке различных продуктов на основе штаммов рода Bacillus для коммерческого использования в качестве микробных пестицидов, фунгицидов или удобрений [72]. Бациллы способны образовывать споры, которые позволяют им противостоять неблагоприятным условиям окружающей среды, их можно безопасно транспортировать, хранить и суспендировать в жидкости для удобства применения [76]. Несколько коммерческих продуктов на основе B. amyloliquefaciens, B. licheniformis, B. pumilus и B. subtilis продаются как биофунгициды.
Согласно данным литературы, разные штаммы рода Bacillus продуцируют разные типы липопептидов, и как следствие, обладают разной активностью даже в отношении одного и того же возбудителя. Молекулярные и физиологические механизмы, с помощью которых бациллы проявляют биофунгицидную активность, во многих случаях полностью не изучены. Предполагается, что биорегулирующая активность бацилл является результатом согласованного действия их антибактериальной активности и колонизации растений. Исследования последних лет, обобщенные в настоящем обзоре, доказывают ключевую роль сурфактина в колонизации корней среди ассоциированных с растениями видов Bacillus spp., образовании биопленки, а также в качестве сигнальной молекулы во время формирования внеклеточного матрикса. Для широкого спектра растений-хозяев показано, что малые концентрации сурфактина являются пусковым сигналом к включению сложного каскада защитных механизмов индуцированной системы устойчивости к фитопатогенам. Учитывая, что такой механизм защиты растений является более благоприятным для окружающей среды, чем применение пестицидов, стоит рассмотреть использование сурфактина в сельском хозяйстве в качестве природного биопестицида и стимулятора роста растений. Все представленные в настоящем обзоре эксперименты по использование сурфактина были выполнены в лабораторных условиях. В настоящее время речь не идет о широком применении сурфактина в сельском хозяйстве. Однако тот факт, что именно сурфактин является ведущим липопептидом среди липопептидов рода Bacillus, должен стимулировать исследователей к скринингу эффективных штаммов-продуцентов сурфактина среди бацилл, для выбора кандидатов для производства биопестицидов.
Работа выполнена при финансовой поддержке гранта Министерства науки и высшего образования Российской Федерации “Анализ микробиомов растений и беспозвоночных животных экстремальных мест обитания с целью разработки штаммов-продуцентов новых метаболитов и ферментов” № 075-15-2021-1396 от 26 октября 2021 г.
Список литературы
Fracchia L., Banat J.J., Cavallo M., Ceres C., Banat I.V. // AIMS Bioengineering. 2015. V. 2. № 3. P. 144–162. https://doi.org/10.3934/bioeng.2015.3.144
Wu Y.S., Ngai S.C., Goh B.H., Chan K.G., Lee L.H., Chuah L.H. // Front Pharmacol. 2017. V. 8. Art. 76. https://doi.org/10.3389/fphar.2017.00761
Arima K., Kakinuma A., Tamura G. // Biochem. Biophys. Res. Commun.1968. V. 31. P. 488–494. https://doi.org/10.1016/0006-291X(68)90503-2
Lilge L., Ersig N., Hubel P., Aschern M., Pillai E., Klausmann P., Pfannstiel J., Henkel M., Heravi K.M., Hausmann R. // Microorganisms. 2022. V. 10. № 4. P. 779. https://doi.org/10.3390/microorganisms10040779
Bartal A., Vigneshwari A., Boka B., Voros M., Takacs I., Kredics L., Manczinger L., Varga M., Vágvolgyi C., Szekeres A. // Molecules. 2018. V. 23. № 10 Art. 2675. https://doi.org/10.3390/molecules23102675
Stein T. // Mol. Microbiol. 2005. V. 56. № 4. P. 845–857. https://doi.org/10.1111/j.1365-2958.2005.04587.x
Caulier S., Nannan C., Gillis A., Licciardi F., Bragard C., Mahillon J. // Front. Microbiol. 2019. V. 10. Art. 302. https://doi.org/10.3389/fmicb.2019.00302
Hsieh F.C., Li M.C., Lin T.C., Kao S.S. // Curr. Microbiol. 2004. V. 49. P. 186–191. https://doi.org/10.1007/s00284-004-4314-7
Long X., He N., He Y., Jiang J., Wu T. // Bioresour. Technol. 2017. V. 241. P. 200–206. https://doi.org/10.1016/j.biortech.2017.05.120
Marcelino L., Puppin-Rontani J., Coutte F., Machini M.T., Etchegaray A., Puppin-Rontani R.M. // Amino Acids. 2019. V. 51. P. 1233–1240. https://doi.org/10.1007/s00726-019-02750-1
Banat I.M., Franzetti A., Gandolfi I., Bestetti G., Martinotti M.G., Fracchia L., Smyth T.J., Marchant R. // Appl. Microbiol. Biotechnol. 2010. V. 87. № 2. P. 427–444. https://doi.org/10.1007/s00253-010-2589-0
Varvaresou A., Iakovou K. // Lett. Appl. Microbiol. 2015. V 61. № 3. P. 214–223. https://doi.org/10.1111/lam.12440
Kakinuma A., Hori M., Isono M., Tamura G., Arima K. // Agric. Biol. Chem. 1969. V. 33. P. 971–972. https://doi.org/10.1080/00021369.1969.10859408
Kakinuma A., Sugino H., Isono M., Tamura G., Arima K. // Biol. Chem. 1969. V. 33. P. 973–976. https://doi.org/10.1080/00021369.1969.10859409
Liu J.F., Mbadinga S.M., Yang S.Z., Gu J.D., Mu B.Z. // Int. J. Mol. Sci. 2015 V. 16. № 3. P. 4814–4837. https://doi.org/10.3390/ijms16034814
Liu J., Zou A., Mu B. // Colloids and Surfaces A: Physicochemical and Engineering Aspects. 2010. V. 361. P. 90–95. https://doi.org/10.1016/j.colsurfa.2010.03.021
Vass E., Besson F., Majer Z., Volpon L., Hollosi M. // Biochem. Biophys. Res Commun. 2001. V. 282. № 1. P. 361–367. https://doi.org/10.1006/bbrc.2001.4469
Bonmatin J.-M., Laprevote O., Peypoux F. // Comb. Chem. High Throughput Screen. 2003. V. 6. № 6. P. 541–556. https://doi.org/10.2174/138620703106298716
Aleti G, Sessitsch A, Brader G. // Comput. Struct. Biotechnol. J. 2015. V. 13. P. 192–203. https://doi.org/10.1016/j.csbj.2015.03.003
Kecskemeti A., Bartal A., Boka B., Kredics. L, Manczinger L., Shine K., Alharby N.S., Khaled J.M., Varga M., Vagvolgyi C., Szekeres A. // Molecules. 2018. V. 23. Art. 2224. https://doi.org/10.3390/molecules23092224
Aleti G., Lehner S., Bacher M., Compant S., Nikolic B., Plesko M., Schuhmacher R., Sessitsch A., Brader G. // Environ. Microbiol. 2016. V. 18. № 8. P. 2634–2645. https://doi.org/10.1111/1462-2920.13405
Liu J.F., Yang J., Yang S.Z., Ye R.Q., Mu B.Z. // Appl. Biochem. Biotechnol. 2012. V. 166. № 8. P. 2091–2100. https://doi.org/10.1007/s12010-012-9636-5
Liu X., Tao X., Zou A., Yang S., Zhang L., Mu B. // Protein Cell. 2010. V. 1. № 6. P. 584–594. https://doi.org/10.1007/s13238-010-0072-4
Kracht M., Rokos H., Ozel M., Kowall M., Pauli G., Vater J. // J. Antibiot. (Tokyo). 1999. V. 52. № 7. P. 613–619. https://doi.org/10.7164/antibiotics.52.613
Eeman M., Berquand A., Dufrene Y.F., Paquot M., Dufour S., Deleu M. // Langmuir. 2006. V. 22. № 26. P. 11337–11345.
Liu X., Yang S., Mu B. // Process Biochemistry. 2009. V. 44. № 1. P. 1144–1151. https://doi.org/10.1016/j.procbio.2009.06.014
Morikawa M., Hirata Y., Imanaka T. // Biochim. Biophys Acta. 2000. V. 1488. № 3. P. 211–218. https://doi.org/10.1016/s1388-1981(00)00124-4
Dufour S., Deleu M., Nott K., Wathelet B., Thonart P., Paquot M. // Biochim. Biophys Acta. 2005. V. 1726. № 1. P. 87–95. https://doi.org/10.1016/j.bbagen.2005.06.015
Jiang J., Gao L., Bie X., Lu Z., Liu H., Zhang C., Lu F., Zhao H. // BMC Microbiol. 2016. V. 16. Art. 31. https://doi.org/10.1186/s12866-016-0645-3
Medema M.H., Kottmann R., Yilmaz P., Cummings M., Biggins J.B., Blin K., de Bruijn I., Chooi Y.H., Claesen J., Coates R.C. // Nat. Chem. Biol. 2015. V. 11. P. 625–631. https://doi.org/10.1038/nchembio.1890
Theatre A., Cano-Prieto C., Bartolini M., Laurin Y., Deleu M., Niehren J., Fida T., Gerbinet S., Alanjary M., Medema M.H., Leonard A., Lins L., Arabolaza A., Gramajo H., Gross H., Jacques P. // Front. Bioeng. Biotechnol. 2021. V. 9. Art. 623701. https://doi.org/10.3389/fbioe.2021.623701
Koumoutsi A., Chen X.H., Henne A., Liesegang H., Hitzeroth G., Franke P., Vater J., Borriss R. // J. Bacteriol. 2004. V. 186. № 4. P. 1084–1096. https://doi.org/10.1128/JB.186.4.1084-1096.2004
Willenbacher J., Mohr T., Henkel M., Gebhard S., Mascher T., Syldatk C., Hausmann R. // J. Biotechnol. 2016. V. 224. P. 14–17. https://doi.org/10.1016/j.jbiotec.2016.03.002
Jiao S., Li X., Yu H., Yang H., Li X., Shen Z. // Biotechnol. Bioeng. 2017. V. 114. P. 832–842. https://doi.org/10.1002/bit.26197
Quadri L.E., Weinreb P.H., Lei M., Nakano M.M., Zuber P., Walsh C.T. // Biochemistry. 1998. V. 37. № 6. P. 1585–1595. https://doi.org/10.1021/bi9719861
Nakano M.M., Corbell N., Besson J., Zuber P. // MGG Mol. Gen. Genet. 1992. V. 232. P. 313–321. https://doi.org/10.1007/BF0028001
Li X., Yang H., Zhang D., Li X., Yu H., Shen Z. // J. Ind. Microbiol. Biotechnol. 2015. V. 42. P. 93–103. https://doi.org/10.1007/s10295-014-1527-z
Rahman F.B., Sarkar B., Moni R., Rahman M.S. // Biotechnol. Rep. 2021. V. 32. P. e00686. https://doi.org/10.1016/j.btre.2021.e00686
Seydlova G., Svobodova J. // Cent. Eur. J. Med. 2008. V. 3. P. 123–133. https://doi.org/10.2478/s11536-008-0002-5
Ishigami Y., Osman M., Nakahara H., Sano Y., Ishiguro R., Matsumoto M. // Colloids Surf. B. 1995. V. 4. P. 341–348.
Chen B., Wen J., Zhao X., Ding J., Qi G. // Front. Microbiol. 2020 V. 11. Art. 631. https://doi.org/10.3389/fmicb.2020.00631
Ongena M., Jourdan E., Adam A., Paquot M., Brans A., Joris B., Arpigny J.L., Thonart P. // Environ Microbiol. 2007. V. 9. № 4. P.1084–1090. https://doi.org/10.1111/j.1462-2920.2006.01202.x
Deleu M., Lorent J., Lins L., Brasseur R., Braun N., El Kirat K., Nylander T., Dufrene Y.F., Mingeot-Leclercq M.P. // Biochim. Biophys. Acta. 2013. V. 1828. № 2. P. 801–815. https://doi.org/10.1016/j.bbamem.2012.11.007
Li T, Li L, Du F, Sun L, Shi J, Long M, Chen Z. // Molecules. 2021. V. 26. № 11. Art. 3438. https://doi.org/10.3390/molecules26113438
Tran C., Cock I.E., Chen X., Feng Y. // Antibiotics (Basel). 2022. V. 11. № 1. Art. 88. https://doi.org/10.3390/antibiotics11010088
Maget-Dana R., Ptak M. // Biophys. J. 1995. V. 68. P. 1937–1943. https://doi.org/10.1016/S0006-3495(95)80370-X
Maget-Dana R., Ptak M. // J. Colloid Interface Sci. 1992. V. 153. P. 285–291. https://doi.org/10.1016/0021-9797(92)90319-H
Liu J., Li W., Zhu X., Zhao H., Lu Y., Zhang C., Lu Z. // Appl. Microbiol. Biotechnol. 2019 V. 103. № 11. P. 4565–4574. https://doi.org/10.1007/s00253-019-09808-w
Stoll A., Salvatierra-Martínez R., Gonzalez M., Araya M. // Microorganisms. 2021 V. 9. № 11. Art. 2251. https://doi.org/10.3390/microorganisms9112251
Marahiel M.A., Nakano M.M., Zuber P. // Mol. Microbiol. 1993. V. 7. № 5. P. 631–636. https://doi.org/10.1111/j.1365-2958.1993.tb01154.x
Raaijmakers J.M., De Bruijn I., Nybroe O., Ongena M. // FEMS Microbiol. Rev. 2010. V. 34. № 6. P. 1037–1062. https://doi.org/10.1111/j.1574-6976.2010.00221.x
Sachdev D.P., Cameotra S.S. // Appl. Microbiol. Biotechnol. 2013. V. 97. P. 1005–1016. https://doi.org/10.1007/s00253-012-4641-8
Chowdhury S.P., Hartmann A., Gao X., Borriss R. // Front. Microbiol. 2015. V. 6. Art. 780. https://doi.org/10.3389/fmicb.2015.00780
Hofemeister J., Conrad B., Adler B., Hofemeister B., Feesche J., Kucheryava N., Steinborn G., Franke P., Grammel N., Zwintscher A., Leenders F., Hitzeroth G., Vater J. // Mol. Genet. Genomics. 2004. V. 272. № 4. P. 363–378. https://doi.org/10.1007/s00438-004-1056-y
Morikawa M. // J. Biosci Bioeng. 2006. V. 101. № 1. P. 1–8. https://doi.org/10.1263/jbb.101
Therien M., Kiesewalter H.T., Auria E., Charron-Lamoureux V., Wibowo M., Maroti G., Kovacs A.T., Beauregard P.B. // Biofilm. 2020. V. 2. Art. 100021. https://doi.org/10.1016/j.bioflm.2020.100021
Asaka O., Shoda M. // Appl. Environ. Microbiol. 1996. V. 62. № 11. P. 4081–4085. https://doi.org/10.1128/aem.62.11.4081-4085.1996
Toure Y., Ongena M., Jacques P., Guiro A., Thonart P. // J. Appl. Microbiol. 2004. V. 96. № 5. P. 1151–1160. https://doi.org/10.1111/j.1365-2672.2004.02252.x
Bais H.P., Fall R., Vivanco J.M. // Plant Physiol. 2004. V. 134. № 1. P. 307–319. https://doi.org/10.1104/pp.103.028712
Zeriouh H., de Vicente A., Perez-García A., Romero D. // Environ. Microbiol. 2014. V. 16. № 7. P. 2196–2211. https://doi.org/10.1111/1462-2920.12271
Luo C., Zhou H., Zou J., Wang X., Zhang R., Xiang Y., Chen Z. // Appl. Microbiol. Biotechnol. 2015. V. 99. № 4. P. 1897–1910. https://doi.org/10.1007/s00253-014-6195-4
Fan H., Zhang Z., Li Y., Zhang X., Duan Y., Wang Q. // Front. Microbiol. 2017. V. 8. Art. 1973. https://doi.org/10.3389/fmicb.2017.01973
Nifakos K., Tsalgatidou P.C., Thomloudi E.E., Skagia A. Kotopoulis D., Baira E., Delis C., Papadimitriou K., Markellou E. Venieraki A., Katinakis P. // Plants (Basel). 2021. V. 10. № 8. Art. 1716. https://doi.org/10.3390/plants10081716
García-Gutierrez L. Zeriouh H., Romero D., Cubero J., de Vicente A., Perez-García A. // Microb. Biotechnol. 2013. V. 6. № 3. P. 264–274. https://doi.org/10.1111/1751-7915.12028
Desoignies N., Schramme F., Ongena M., Legrève A. // Mol. Plant Pathol. 2013 V. 14. № 4. P. 416–421. https://doi.org/10.1111/mpp.12008
Cawoy H., Mariutto M., Henry G., Fisher C., Vasilyeva N., Thonart P., Dommes J., Ongena M. // Mol. Plant Microbe Interact. 2014. V. 27. № 2. P. 87–100. https://doi.org/10.1094/MPMI-09-13-0262-R
Waewthongrak W., Leelasuphakul W., McCollum G. // PLoS One. 2014. V. 9. № 10. Art. e109386. https://doi.org/10.1371/journal.pone.0109386
Rahman A., Uddin W., Wenner N.G. // Mol. Plant Pathol. 2015. V. 16. № 6. P. 546–558. https://doi.org/10.1111/mpp.12209
Yamamoto S., Shiraishi S., Suzuki S. // Lett. Appl. Microbiol. 2015. V. 60. № 4. P. 379–386. https://doi.org/10.1111/lam.12382
Rodriguez J., Tonelli M.L., Figueredo M.S., Ibanez F., Far A. // Eur. J. Plant Pathol. 2018. V. 152. P. 845–851. https://doi.org/10.1007/s10658-018-1524-6
Черепанова Е.А., Благова Д.К., Бурханова Г.Ф., Сарварова Е.С., Максимов И.В. // Экобиотех. 2019. Т. 2. № 3. С. 339–346. https://doi.org/10.31163/2618-964X-2019-2-3-339-346
Li Y., Heloir M.C., Zhang X., Geissler M., Trouvelot S., Jacquens L., Henkel M., Su X. Fang X., Wang Q., Adrian M. // Mol. Plant Pathol. 2019. V. 20. № 8. P. 1037–1050. https://doi.org/10.1111/mpp.12809
Debois D., Fernandez O., Franzil L. Jourdan E., de Brogniez A., Willems L., Clément C., Dorey S., De Pauw E., Ongena M. // Environ. Microbiol. Rep. 2015. V. 7. № 3. P. 570–582. https://doi.org/10.1111/1758-2229.12286
Поликсенова В.Д. // Вестник БГУ. Сер. 2. 2009. № 1. С. 48–60.
Straight P.D., Willey J.M., Kolter R. // J. Bacteriol. 2006 V. 188. № 13. P. 4918–4925. https://doi.org/10.1128/JB.00162-06
Pérez-García A., Romero D., de Vicente A. // Curr. Opin. Biotechnol. 2011. V. 22. № 2. P. 187–193. https://doi.org/10.1016/j.copbio.2010.12.003
Дополнительные материалы отсутствуют.
Инструменты
Прикладная биохимия и микробиология


