Журнал общей биологии, 2021, T. 82, № 2, стр. 143-154
Модели совместного распределения видов на примере донных сообществ малых рек волжского бассейна
В. К. Шитиков 1, Т. Д. Зинченко 1, *, Л. В. Головатюк 1
1 Институт экологии Волжского бассейна РАН, Филиал Федерального государственного
бюджетного учреждения науки Самарского федерального исследовательского центра РАН
445003 Тольятти, ул. Комзина, 10, Россия
* E-mail: zinchenko.tdz@yandex.ru
Поступила в редакцию 20.10.2020
После доработки 05.11.2020
Принята к публикации 23.12.2020
Аннотация
Рассмотрены теоретические и практические аспекты построения моделей совместного распределения видов, которые являются современным инструментом анализа экологических сообществ. Показано, что в случае, когда данные наблюдений составляют количественные показатели популяционной плотности (в частности, численности видов в гидробиологических исследованиях), представляется нецелесообразным использование метода MaxEnt и других, основанных на концепции точек “псевдоотсутствия”. Современные многомерные модели совместного распределения сообществ должны включать совокупность параметров, оценивающих влияние на встречаемость видов следующих групп фиксированных и случайных факторов: (а) ковариат и категориальных переменных, описывающих условия окружающей среды и характеристики биотопов, (б) основных показателей, характеризующих каждый вид и филогенетическую структуру сообществ, (в) функции пространственной автокорреляции данных в точках наблюдений, (г) остаточной (т.е. не обусловленной внешними факторами) ассоциативности видов. Анализ опубликованных материалов и примеры практической реализации показали, что перечисленным требованиям в целом удовлетворяет методическая платформа и R-пакет HMSC (Hierarchical Modelling of Species Communities), на основе которых выполняется построение многомерных иерархических обобщенных линейных моделей со смешанными параметрами, оцениваемых байесовской процедурой.Представлено описание основных концепций и блоков пакета HMSC и обсуждаются результаты построения моделей на основе данных авторов – многолетних гидробиологических исследований донных сообществ 132 малых и средних рек бассейна Средней и Нижней Волги. Приведен анализ параметров совокупности одномерных моделей-претендентов распределения численности подсемейства Prodiamesinae (Diptera, Chironomidae) и построена прогнозная карта его ареала в границах региона. Для иллюстрации многомерного случая построена модель совместного пространственного распределения 31 вида хирономид и выполнен анализ ее коэффициентов. Построен остаточный корреляционный граф статистически значимых межвидовых взаимодействий. Делается вывод, что метод и программный пакет HMSC может эффективно применяться для решения фундаментальных проблем экологии сообществ: как зависят ареалы отдельных популяций, структура их сообществ и характер межвидовых взаимодействий от условий окружающей среды, а также для прогнозирования будущих тенденций этих процессов в ответ на глобальные изменения.
Структура пространственного распределения сообществ и его связь с условиями обитания популяций являются важнейшими направлениями экологических исследований. После появления в 1980-х годах пакета BIOCLIM (Busby, 1991) моделирование распределения видов (SDM – Species Distribution Models) и экологических ниш (ENM – Environmental Niche Models) стало мощным инструментом (макро)экологических и биогеографических исследований и оценки роли факторов, влияющих на распространение видов (Peterson et al., 2011). Эти методы оказались также весьма эффективными в палеоэкологии, филогенетике, управлении биоресурсами и охране дикой природы (Araújo et al., 2019). Появилось огромное количество литературы по различным методам SDM/ENM, использование которых широко освещено в работах зарубежных экологов (Franklin, 2009; Guisan et al., 2017) и подробном обзоре российских коллег (Лисовский и др., 2020).
Анализ пространственного распределения видов основан на двух различных концептуальных подходах. Процессно-ориентированные SDM (также известные как ранговые модели динамики популяций; Zurell et al., 2016) включают в явной форме модельные структуры и параметры, описывающие механизмы основных экологических процессов в сообществах. Необходимость оценки коэффициентов интенсивности размножения, смертности, расселения и демографической стохастичности (Vellend, 2016; Розенберг и др., 2020), а также их зависимость от выборочных процессов получения данных, делают такой подход пока еще труднодоступным, хотя учет базовых процессов в сообществах должен приветствоваться в любых случаях (D’Amen et al., 2017).
Другой подход можно назвать коррелятивным, в том смысле, что он основан на нахождении статистических зависимостей между факторами окружающей среды и данными о встречаемости видов. Описаны десятки методов построения SDM (Norberg et al., 2019), которые различаются многими аспектами, включая состав исходных данных (“только присутствие” видов в точках отбора проб, “присутствие–отсутствие” или количественная оценка обилия), структурные допущения моделей (обобщенная линейная модель, опорные векторы или случайный лес), алгоритмы получения решения (использование максимума правдоподобия или байесовский подход) и техническая реализация (доступен ли метод в виде R-пакета или как самостоятельный программный продукт). Успешно ведутся также работы по ранжированию совокупности построенных моделей по степени их компетентности и построению их ансамблей (коллективов), в которых предсказания нескольких моделей взвешиваются и усредняются (Breiner et al., 2018).
Из множества применяемых алгоритмов можно отметить чаще всего используемый метод максимальной энтропии, реализованный в программе MaxEnt (Phillips et al., 2006; Лисовский, Дудов, 2020). Алгоритм предсказывает вероятность присутствия вида в произвольной точке географического пространства, основываясь только на точках, где он уже был зарегистрирован (РО – presence-only). Итогом работы MaxEnt является расчет экспоненциальной функции, аргументами которой являются частные функции отдельных предикторов (линейные, квадратичные, множественные и др.) с коэффициентами λ, оценивающими вклад соответствующего экологического фактора. Пошаговый выбор оптимальной модели и настройка коэффициентов λ осуществляется с учетом минимизации ошибки предсказания как на исходной выборке РО, так и на множестве случайно отобранных точек, где, как предполагается, вид отсутствует (РА – pseudo-absence, или “background” points). Успешность работы алгоритма во многом зависит от выбора формы частных функций, объема выборки РА, предварительной фильтрации исходных данных, использования слоя коррекции и др. (Лисовский, Дудов, 2020).
Использование случайных фоновых точек – это классическая процедура, которая известна как функция выбора ресурсов (Resource Selection Functions; Johnson, 1980), предполагающая сравнение текущих условий среды обитания с оценками доступности необходимых ресурсов для сообщества. Поскольку часто очень трудно подтвердить на практике отсутствие вида, эта процедура оценивает не столько искомую вероятность присутствия вида, сколько неоднородность используемых эмпирических данных. В частности, показатели успеха предсказания отсутствия часто определяются “капризными ноликами”, т.е. теми точками, где вид просто не может встречаться (Hastie, Fithian, 2013; Guisande et al., 2017). Поэтому если доступны данные “присутствия–отсутствия” или тем более количественные оценки численности популяций, целесообразно применять адекватные статистические методы.
Модели SDM были в основном разработаны для моделирования ареала только одного вида, в то время как часто возникает задача оценить совместное распределение многих видов, образующих сообщества (Clark et al., 2014; Warton et al., 2015). Один из возможных подходов – сложение моделей распределения (stacked SDM, SSDM), где на первом этапе строится совокупность моделей для отдельных видов, а затем их результаты комбинируются (Calabrese et al., 2014). В отличие от него, другой обобщенный способ анализа (joint SDM, JSDM) объединяет видовой уровень данных модели в одну модель, которая одновременно подстраивается под структуру всего сообщества. Это позволяет не только выявлять межвидовые ассоциации, но и соотнести полученные закономерности с характеристиками видов (Abrego et al., 2017), их филогенетическими особенностями или паттернами совместного сосуществования (Pollock et al., 2014). Наконец, класс моделей SDFA (Spatial Dynamic Factor Analysis; Thorson et al., 2016) рассматривает распределение структуры сообществ под влиянием факторов среды не только в пространстве, но и во времени.
Изменения в характере межвидовых взаимодействий, связанные с различиями условий окружающей среды, были обнаружены для широкого спектра таксономических групп (например, Brooker, 2006). Выводы о наличии и силе межвидовых взаимодействий традиционно делают на основе данных наблюдений за встречаемостью видов с использованием разных статистических методов: многомерной ординации, парной корреляции, моделей агрегации и сегрегации видов и т.д. (Legendre P., Legendre L., 2012). Здесь важная проблема состоит в том, что выводы о совместном сосуществовании, определяемом межвидовыми взаимодействиями, смешиваются с эффектами, порожденными совместной вариацией отклика видов на абиотические изменения. Поскольку JSDM включают в явном виде измеренные экологические ковариаты, найденные с их помощью оценки ассоциативности видов более адекватны для выявления истинных взаимодействий, чем “сырые” индексы совместной встречаемости (Warton et al., 2015).
Далее рассматривается методика построения JSDM с использованием одной из версий моделей GLMM (Generalized Linear Mixed Models), которая по статистической терминологии трактуется как многомерная иерархическая обобщенная линейная модель со смешанными параметрами, основанная на байесовской процедуре их оценки. В качестве рабочего примера нами использовались результаты многолетней гидробиологической съемки донных сообществ малых и средних рек на территории Среднего и Нижнего Поволжья (Зинченко, 2009, 2011; Golovatyuk et al., 2018). Представленные результаты вычислений получены с использованием статистической среды R ver. 3.6 и пакета HMSC (Hierarchical Modelling of Species Communities – иерархическое моделирование сообществ видов), разработанного Оваскайненом с соавторами. В связи с этим, последующее изложение методического материала осуществляется на основе книги (Ovaskainen, Abrego, 2020) и предыдущих статей этого авторского коллектива (Ovaskainen et al., 2016а, b, 2017; Tikhonov et al., 2017, 2020).
МАТЕРИАЛ И МЕТОДЫ
Описание статистической модели HMSC
Типичный набор данных, полученный в ходе экологических исследований сообществ, включает совокупность видов j = 1… ns, выявленных на множестве ny биотопов (строже говоря – в точках отбора проб, sampling units), i = 1… ny. Используемая обобщенная линейная смешанная модель GLMM может быть применена к различным показателям обилия видов yij (наличие/отсутствие, количество, биомасса, покрытие и т.д.) путем включения различных функций связи и постулирования законов распределения ошибок. В контексте HMSC выборочные данные подгоняются многомерной моделью, т.е. число переменных отклика совпадает с числом видов ns. Для каждого вида задается статистическое распределение yij ∼ D{Lij, $\sigma _{j}^{2}$), где Lij – математическое ожидание плотности вида j в точке i, а $\sigma _{j}^{2}$ – параметр дисперсии (не используется в случае распределения Пуассона или Бернулли). В случае нормального распределения значение Lij моделируется как линейная функция от двух групп предикторов, представляющие фиксированные и случайные факторы:
(1)
${{L}_{{ij}}} = \sum\limits_{k = 1}^{{{n}_{c}}} {{{x}_{{ij}}}{{\beta }_{{jk}}} + {{\varepsilon }_{{ij}}}} ,\,\,\,\,{\text{где}}\,\,\,\,{{\varepsilon }_{{ij}}} = \sum\limits_{h = 1}^{{{n}_{f}}} {{{\eta }_{{ih}}}{{\lambda }_{{jh}}}({{z}_{i}})} .$Первый член выражения (1), моделирующий влияние фиксированных факторов, является обычной линейной регрессией, где xik – значение k-й переменной окружающей среды, наблюдаемое в точке i, k = 1… nc, а βjk – коэффициент регрессии, представляющий долю линейного отклика вида j на эту ковариату. Чтобы обеспечить параметризацию модели с разреженными данными или редкими видами, принимается распределение коэффициентов регрессии как βj. ~ N(µ, V), где вектор µ является оценкой средней реакции вида на измеренные ковариаты, а дисперсионно-ковариационная матрица V соответствует вариации отдельных видов относительно математического ожидания. Здесь и далее точка в выражении βj. означает, что индекс k пробегает все значения от 1 до nc для каждого фиксированного j.
Вклад совокупности случайных факторов, включая пространственную автокорреляцию и межвидовые взаимодействия, моделируется вторым членом εij, который представляет собой сумму произведений nf латентных факторов и их нагрузок. Здесь ηih, h = 1… nf – это значение фактора для выборочной точки i, а λjh(zi⋅) – факторная нагрузка на вид j со стороны латентного фактора h, обобщающего произвольный набор предикторов zi.. Если, в частности, принять, что ${{\lambda }_{{jh}}}({{z}_{i}}) = \sum\nolimits_{k = 1}^{{{n}_{c}}} {{{x}_{{ik}}}{{\lambda }_{{jhk}}}} $, то структура ковариаций между видами εij становится функцией состояния окружающей среды, определяемого исходным набором переменных-ковариат x. Некоторые случайные факторы могут быть связаны с вложенной структурой плана исследований (например, бассейн водохранилища → река → точка отбора проб), поэтому рассматриваемая модель трактуется как иерархическая.
Коэффициенты модели (1) рассчитываются по данным наблюдений x с использованием байесовской методологии, которая основана на итеративном процессе подстройки исходных (априорных) оценок модельных параметров θ и получении их результирующего (апостериорного) распределения. Этот процесс реализуется методом построения длинных итеративных последовательностей нескольких марковских цепей Монте-Карло (MCMC), для которых распределение переходов определяется функцией P(θ|Y, X). Процесс моделирования часто довольно длительный и продолжается до тех пор, пока флуктуации текущих значений параметров не приблизятся к некоторому стационарному распределению. Для проверки сходимости цепей используются приемы визуальной и формальной диагностики. Для проверки адекватности модели и сравнения ее различных вариантов используется алгоритм перекрестной проверки.
Связь модели с основными теоретическими конструкциями экологии сообществ
После построения и диагностики параметризованная модель HMSC (как и любая JSDM) может использоваться для объяснения экологических процессов в сообществах и/или для прогнозирования. Связи информационной структуры платформы HMSC с основными задачами экологии сообществ представлены на рис. 1. Прямоугольники включают обозначения матриц исходных данных, а эллипсы – вычисляемые параметры модели (1), которые могут быть использованы для анализа структуры экологических ниш и межвидовых взаимодействий в сообществе.
Рис. 1.
Связи между теоретическими конструкциями экологии сообществ и статистической структурой платформы HMSC (Ovaskainen, Abrego, 2020). Матрицы исходных данных: Y – обилие видов, X – факторы среды, T – свойства видов, C – филогенетические корреляции, Π – план исследования, S – географические координаты. Переменные и параметры модели: L – линейные предикторы, LF – фиксированные эффекты, LR – случайные эффекты, β – ниши видов, Γ – влияние характеристик видов в нише, ρ – филогенетический сигнал в нише, V – остаточная ковариация видов в нише, Н – факторные нагрузки биотопов, α – пространственная шкала биотопов, Λ – факторные нагрузки видов, Ω – матрица объединения видов, Φ – локальные потери нагрузок видов, δ – глобальные потери нагрузок видов, Σ – матрица остаточной дисперсии.
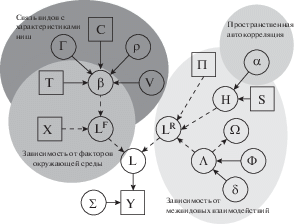
Часть коэффициентов βj. ~ N(µ, V) модели HMSC, описывающих фиксированные эффекты, определяют, в какой мере изменчивость факторов X окружающей среды влияет на встречаемость и/или обилие видов. Каждый вид имеет свой вектор β-параметров, ограничивающий некоторый объем гиперпространства, а значит, и свою экологическую нишу. Однако границы ниши определяются не только параметрами внешнего воздействия, но и изменчивостью внутрипопуляционных характеристик Γ (species-specific traits), таких как размер тела, морфологические особенности или тип питания у животных, размер семян или жизненная форма у растений и т.д.
Другим важным фиксированным эффектом, определяющим разбиение сообщества на экологические ниши, является филогенетическое родство между видами. Для того чтобы структурировать ниши по этому признаку, филогенетическое дерево преобразуется в матрицу C ns × ns, элементы которой (cij = 0÷1) оценивают филогенетические корреляции, определяемые как доля общего эволюционного времени для каждой пары видов i и j. HMSC реализует филогенетическую корреляционную модель как βf ~ N(μf, W), где W = ρ C + + (1 – ρ)I, а вычисляемый параметр ρ = 0÷1 измеряет силу филогенетического сигнала. Если предположить, что ниши полностью филогенетически структурированы, то ρ = 1 и коэффициенты модели имеют многомерное нормальное распределение βf ~ N(μf, C). Эта модель имеет одинаковое ожидание μf для всех видов, но предсказывает, что филогенетически близкие виды в среднем будут иметь меньший статистический разброс, чем филогенетически отдаленные виды.
Совокупность случайных эффектов HMSC (показана справа на рис. 1) моделирует влияние различных биотических или абиотических факторов на изменчивость отклика Y (не изменяя его математического ожидания). В большинстве случаев при реализации плана исследований выборочные точки связаны с пространственными координатами, и тогда зависимость между остатками εij обусловлена явлением, называемым пространственной автокорреляцией (наблюдения в точках, расположенных близко друг к другу, вероятнее всего, будут более сходными, чем для выборочных единиц, расположенных далеко друг от друга). HMSC моделирует любую автоковариационную структуру, заданную пользователем и зависящую от расстояния dij между выборочными точками i–j. Чаще всего используется экспоненциальная функция f(dij) = $\sigma _{S}^{2}$exp(–dij/α), где пространственная дисперсия ($\sigma _{S}^{2}$) и вектор масштаба (α) являются положительными параметрами пространственного случайного эффекта, который оценивается при построении модели.
Если отклоняется гипотеза, что все виды в сообществе функционируют независимо, то совокупность статистически значимых положительных или отрицательных взаимодействий между видами может в конечном итоге влиять на индивидуальную численность Y каждого из них. По этой причине целесообразно включить в многомерный анализ случайный эффект, учитывающий дополнительную информацию о том, какие виды встречаются совместно “чаще, чем случайно”. Последняя фраза означает одновременное присутствие пары видов на i-м участке с вероятностью, превышающей ту, которая ожидается из сходства параметров βi. их ниш. В матричной форме эффект ассоциативности видов записывается как $L_{i}^{R}$ ~ N(0, Ω), где Ω =ΛTΛ – экологически ограниченная корреляционная матрица видов. Таким образом, эта группа случайных эффектов генерирует остаточные ковариации сверх тех, что учтены фиксированными эффектами, т.е. выделяет только те ассоциации, которые не могут быть объяснены экологическими ковариатами xik, уже включенными в модель.
Состав исходных данных
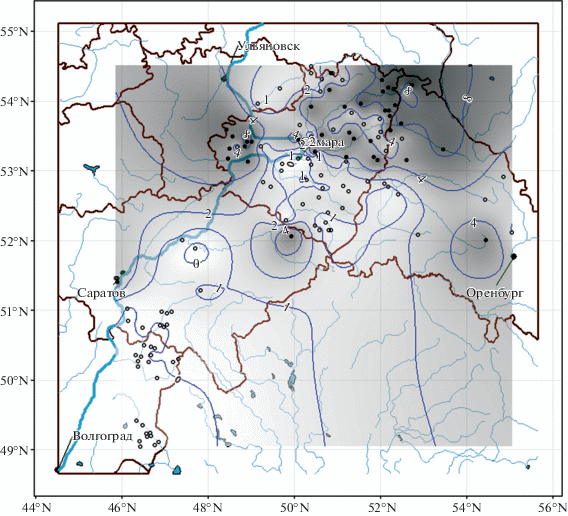
Построение моделей HMSC рассматривается на примере анализа данных гидробиологической съемки донных сообществ бассейна Средней и Нижней Волги (Зинченко, 2011) в разные месяцы вегетационного периода 1990–2019 гг. Гидробиологическую съемку макрозообентоса проводили на 90 малых и 12 средних равнинных реках, притоках Куйбышевского, Саратовского и Волгоградского водохранилищ, в том числе на 6 реках аридного региона бассейна оз. Эльтон (рис. 2). Средние реки были разделены на приблизительно однородные участки: верхнее, среднее, нижнее течение и устье, а каждая малая река принималась как целостный объект. Таким образом, было исследовано 132 локальных сообщества, в каждом из которых по стандартным методикам выделено до 40 видов макрозообентоса. Всего было выполнено 1400 проб и обнаружено 740 видов и таксонов рангом выше вида, удельная численность (экз./м2) которых использовалась для формирования матрицы отклика Y.
Рис. 2.
Карта проведения исследований, районы гидробиологической съемки (• – точки, где обнаружены личинки Prodiamesinae, ο – отсутствие их в пробе) и пространственное распределение численности подсемейства, ln(экз.)/м2, предсказанное моделью HMSC.
В тех же точках отбора проб параллельно проводился мониторинг 30 факторов среды, включающих гидрологические параметры водотоков, показатели качества воды и содержание основных химических ингредиентов (состав донных грунтов, насыщение воды кислородом, минерализация и др.). Растровые таблицы с разрешением 2.5', содержащие основные метеорологические и геоморфологические показатели для региона исследований, были загружены с серверов свободно распространяемой информации WorldClim и ENVIREM (ENVIronmental Rasters for Ecological Modeling). Эти данные использовались для моделирования фиксированных LF и случайных LR эффектов.
РЕЗУЛЬТАТЫ
Модели пространственного распределения одного вида
С использованием данных исследования донных сообществ была построена совокупность одномерных моделей HMSC распределения численности важнейших видов и таксономических групп, что позволило сделать определенные выводы об их связи с факторами окружающей среды и экологических предпочтениях в пределах исследованного региона. Методику анализа рассмотрим на примере подсемейства Prodiamesinae (Diptera, Chironomidae), все виды которого условно принимались экологически идентичными, а их численности суммировались и логарифмировались. Всего виды этого таксона были обнаружены на 41 участке рек из 132 обследованных.
Полная модель (m1) была построена на основе четырех геофизических и климатических показателей, трех показателей качества воды (фиксированные факторы), географических координат участков и категорий типов грунтов дна рек (случайные факторы Rivers и Ground соответственно). Апостериорное распределение коэффициентов модели было получено с использованием марковского процесса из 30 000 итераций для 4 цепей Монте-Карло. Степень уверенности в истинности коэффициентов оценивали с использованием 2.5–97.5% квантильных значений (табл. 1) и дополнительных статистик, таких как длина эффективной цепочки и коэффициент уменьшения масштаба. Относительную важность каждого показателя, использованного для прогнозирования величины отклика, оценивали по их доле в разложении общей объясненной дисперсии по всем фиксированным и случайным факторам.
Таблица 1.
Апостериорное распределение коэффициентов модели HMSC для прогнозирования пространственного распределения численности таксонов Prodiamesinae
| Наименование и обозначение факторов | Среднее | Стандартное отклонение | Квантили распределения | Доля объясненной вариации отклика | |||
|---|---|---|---|---|---|---|---|
| 2.5% | 97.5% | фактор | группа | ||||
| Среднегодовая температура | β[MTemp] | –0.0532 | 0.0685 | –0.182 | 0.086 | 12.16% | 43.84% |
| Осадки самого засушливого квартала | β[PrecDQ] | –0.0148 | 0.0514 | –0.117 | 0.082 | 4.36% | |
| Высота | β[Alt] | 0.0246 | 0.0098 | 0.00553 | 0.044 | 22.14% | |
| Индекс шероховатости рельефа | β[TRI] | 0.0116 | 0.0096 | –0.0071 | 0.031 | 5.18% | |
| Минерализация воды | β[Miner] | 1.5E-05 | 6.1E-05 | –0.0001 | 0.00014 | 2.24% | 6.64% |
| Аммонийный азот | β[NH4] | 0.0287 | 0.116 | –0.202 | 0.251 | 1.99% | |
| Насыщение кислородом | β[O2] | –0.0089 | 0.0143 | –0.0382 | 0.0193 | 2.41% | |
| Пространственная шкала | α[Rivers] | 2.861 | 2.98 | 0 | 9.83 | 48.68% | 49.52% |
| Категория грунтов | λ[Ground] | -0.618 | 0.69 | –2.44 | –0.01 | 0.84% | |
Качество полученной модели оценивали по стандартному отклонению для остатков (RMSE = = 3.11), коэффициенту детерминации, соответствующему доле общей дисперсии переменной отклика Y, которая объясняется структурой модели (R2 = 0.613), а также информационному критерию Уидли (WAIC = 9634).
Как следует из анализа коэффициентов модели (табл. 1), только 7.5% объясненной вариации приходится на характеристики биотопа – гидрохимические показатели качества воды и состав грунтов. Эти факторы можно рассматривать как статистически незначимые, поскольку 95% интервал надежности их коэффициентов включает ноль. В связи с этим рассматривались еще три модели-претендента с меньшим числом переменных:
− (m2) с использованием только 7 фиксированных факторов: RMSE = 3.99, R2 = 0.287;
− (m3) на основе факторов, характеризующих условия среды в биотопе (Miner, NH4, O2 и Ground): RMSE = 4.48, R2 = 0.116;
− (m4) с использованием климатических (MTemp, PrecDQ) и геофизических (Alt, TRI) фиксированных факторов, а также случайного фактора Rivers, определяющую пространственную автокорреляционную зависимость: RMSE = 2.184, R2 = 0.854, WAIC = 9262.
На рис. 2 представлена картосхема проведения исследований, на которых кружками отмечены районы взятия проб (черным цветом залиты точки, где были обнаружены Prodiamesinae). Серым цветом разной интенсивности показано прогнозируемое по модели m4 HMSC распределение популяционной плотности этого подсемейства. Контурами отмечены изолинии прологарифмированной численности (экз./м2).
Совместное распределение ансамбля видов
Многомерная модель HMSC строилась для оценки пространственного распределения сообщества из 31 вида личинок хирономид, наименования которых, частота встречаемости и филогенетическое дерево приведены в табл. 2. Для численностей видов предварительно выполняли преобразование, приводящее к χ2-дистанции, которое является, по всей вероятности, наиболее разумным компромиссом при учете как роли ведущих компонент, так и вклада редких или малочисленных таксонов (Legendre, Gallagher, 2001).
Таблица 2.
Результаты построения модели HMSC для сообщества хирономид
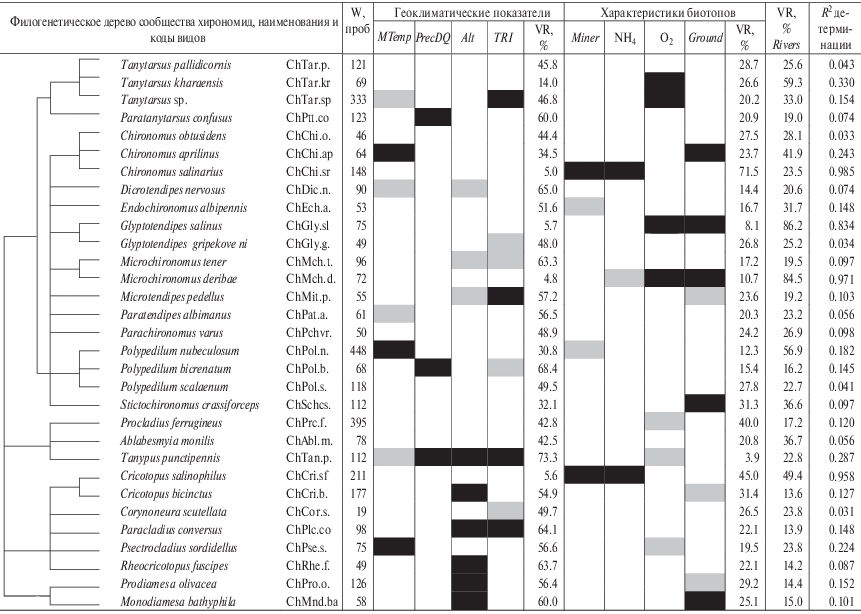
Примечание. W – встречаемость вида в пробах (n = 1400), VR – доля вариации отклика, объясненная группой факторов. Черным цветом отмечены статистически значимые факторы (обозначения см. в табл. 1), положительно влияющие на численность вида, серым цветом – факторы с отрицательным воздействием.
В качестве предикторов модели использовались те же переменные, что и для модели m1 (см. табл. 1) с добавлением матрицы филогенетических корреляций С. Характеристики биотопов, выраженные категориальной переменной Ground (от 1 – чистый песок или галька, до 6 – черный ил и растительные остатки), в этот раз интерпретировали как фиксированный фактор. В табл. 2 черным цветом отмечены ячейки для видов, апостериорное распределение коэффициентов которых статистически значимо смещено в положительную область, т.е. в сторону увеличения соответствующего предиктора. Серым цветом отмечена обратная ситуация, когда снижение значения независимой переменной приводит к увеличению численности видов. Для каждой группы факторов (геоклиматических и гидрохимических показателей, а также пространственной автоковариации Rivers) приведены доли дисперсии VR, объясненной построенной моделью, в общей вариации отклика Y.
Филогенетический сигнал ρ имеет апостериорное распределение со средним 0.991 ± 0.00024, что доставляет убедительные доказательства весьма высокого влияния таксономической иерархии при выделении экологических ниш.
Вектор пространственного масштабирующего фактора α имеет характерную пульсирующую последовательность значений со средними α1 = 2.97, α2 = 0.006, α3 = 3.34, α4 = 2.35, α5 = 0.22, и мы пока не нашли разумного объяснения этому феномену.
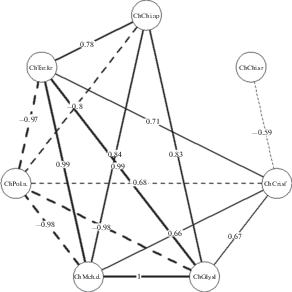
Из матрицы Ω, которая определяет остаточные ковариационные связи между рассматриваемыми видами, были выбраны только те отрицательные или положительные ассоциации, для которых апостериорная вероятность составляет не менее 0.95. Таких видов оказалось 7 из 31 и их корреляционный граф представлен на рис. 3.
ОБСУЖДЕНИЕ И ВЫВОДЫ
Анализ коэффициентов моделей HMSC позволяет установить приоритеты внешних факторов по степени их влияния на пространственное распределение популяционной плотности видов. В частности, представленные результаты свидетельствуют о сильной зависимости численности Prodiamesinae от картографических координат и высоты местности над уровнем моря. Это обычно характерно для ареалов, ограничивающих четко выраженный географический кластер. Действительно, личинки этого подсемейства являются выраженными рео- и оксибионтами и обитают на каменно-песчаных биотопах проточных рек арктоальпийского типа (Макарченко Е., Макарченко М., 1999). В нашем исследовании они встречались в основном в реках Бугульминско-Белебеевской возвышенности лесостепной провинции Высокого Заволжья Самарской области (рис. 2).
Для некоторых других видов характерна выраженная зависимость от гидрохимических условий водной среды и типов биотопа. Например, Chironomus salinarius и Cricotopus salinophilus являются типичными галофилами и обитают в водотоках Приэльтонья с высокой минерализацией и содержанием ионов аммония. Доля дисперсии численности этих видов, объясненная гидрохимическими показателями, высока и составляет до 71.5%, а коэффициент детерминации R2 достигает 0.985. Аналогично, такие эвриоксибионтные виды, как Prodiamesa olivacea, встречаются в основном в реках на песчано-илистых грунтах и поэтому их встречаемость также мало обусловлена влиянием геоклиматических показателей. Доля дисперсии, объясненной пространственной автокорреляцией, в большинстве случаев весьма высока, что вытекает, вероятно, из мозаичной природы окружающей среды (Hutchinson, 1959), где существуют определенные внутренние закономерности.
Низкий уровень коэффициента детерминации у многих видов из табл. 2 объясняется отнюдь не слабыми возможностями построения моделей HMSC, а рядом объективных причин. Во-первых, многие виды-эврибионты относительно равномерно встречаются по всей территории, границы их ареалов размыты и четко выраженные географические кластеры отсутствуют. Во-вторых, нами использовался весьма ограниченный список ковариат окружающей среды x.k и вполне можно предположить, что ведущий фактор, определяющий популяционную плотность вида, просто не вошел в этот список (это может быть и неучтенный гидрохимический показатель, и скорость течения, и некая трудно формализуемая ландшафтная особенность).
В этой связи чрезвычайно ответственным является выбор состава экологических предикторов, которые используются при построении модели. Общей теории в данном случае не существует, и отбор потенциально важных факторов опирается обычно на опыт и интуицию исследователя. Однако не всегда разумно стремление на всякий случай использовать как можно больше исходных переменных. Это вызывает неоправданный рост сложности модели и, следовательно, риск переобучения, что часто приводит к снижению предсказывающей силы модели, а не к ее увеличению. Подтверждение этому – значительное возрастание коэффициента детерминации R2 модели (m4) по сравнению с полной моделью (m1).
За последнее время большое развитие получили компьютерно-интенсивные методы селекции информативных переменных и построения моделей оптимальной сложности (генетический алгоритм, ресэмплинг, кросс-проверка – см. Шитиков, Розенберг, 2014; Шитиков, Мастицкий, 2017). Использование этих алгоритмов в HMSC часто проблематично из-за большой ресурсоемкости построения цепей МСМС достаточной эффективности. Рекомендация применять R2 или информационные критерии для сравнения моделей не является в полном смысле проверкой научных гипотез, поскольку разности этих величин статистически не интерпретируемы. Например, можно ли считать существенным для выбора модели-претендента уменьшение критерия Уидли с 9634 до 9262 или это обусловлено случайными обстоятельствами?
Авторы HMSC, моделируя связи между условиями окружающей среды и встречаемостью видов, тесно связывают свой подход с концепцией экологической ниши. Однако использование термина “ниша” в контексте коррелятивных SDM подвергается критике, поскольку, “чтобы смоделировать нишу, необходимо понять, каким образом морфология, физиология и особенно поведение организмов определяются факторами окружающей среды, а также оценить, как условия среды обитания влияют на приспособляемость вида (рост, выживание и размножение)” (Kearney, 2006, р. 186). В рассматриваемом примере с численностью Prodiamesinae ведущие факторы – высота над уровнем моря и среднегодовая температура – не являются, разумеется, непосредственно параметрами фундаментальной ниши и далеко ими не исчерпываются, хотя косвенным образом определяют механизмы экологических процессов и характеристики биотопов.
Оваскайнен с соавторами расширяют рамки фундаментальной ниши, включая в анализ независимую от экологических факторов матрицу межвидовых ассоциаций Ω и реанимируя тем самым важную для теории ниш концепцию конкурентного исключения и симбиотических отношений. Тем не менее само понятие “экологическая ниша” продолжает оставаться достаточно расплывчатым. Можно задать размерность гиперпространства и оценить для каждого из видов центры статистических распределений всех независимых переменных, но это пока не позволяет оперировать математически многими теоретическими конструкциями ниши. Такие важнейшие понятия, как “объем гиперпространства” и “плотность упаковки”, требуют предварительно определить не только средние, но и граничные (т.е. нормативные) значения параметров ниши, а количественная оценка различий между нишами видов и степени их перекрытия связана с обоснованием многомерной метрики дистанций.
В завершение отметим, что разработанная методическая платформа и пакет функций моделирования сообществ HMSC, на наш взгляд, представляет собой универсальную и комплексную вычислительную среду, позволяющую интегрировать многие разделы данных и давать ответы на широкий спектр вопросов. Специалистов в области экологии сообществ могут заинтересовать следующие привлекательные возможности апробированного метода:
− модели распределения и последующего прогнозирования популяционной плотности видов строятся не только на основе комплекса показателей x.k, определяющих условия окружающей среды, но и с учетом изменчивости характерных признаков видов γ, филогенетических отношений ρ и функции пространственной автоковариации α;
− HMSC обеспечивает построение моделей SDM как на уровне отдельных видов, так и обобщенно для произвольных сообществ; в последнем случае прогноз отклика делается с учетом вклада межвидовых отношений, рассчитанного по матрице Ω остаточных ассоциаций;
− метод может быть применен к различным вариантам схем проведения исследований (включая иерархические, временные или пространственные планы) и ко многим типам распределений эмпирических данных (наличие/отсутствие, счетные показатели и непрерывные величины).
Авторы выражают признательность О. Оваскайнену (University of Helsinki, Finland) и Г. Тихонову (Aalto University, Finland) за методическую поддержку и важные замечания по ходу подготовки рукописи. Исходные данные и тексты скриптов в кодах языка R, выполняющих построение представленных моделей HMSC, доступны на https://stok1946.blogspot.com/2020/11/sdm.html.
Список литературы
Зинченко Т.Д., 2009. Биоиндикационная роль хирономид (Díptera, Chironomidae) в водных экосистемах: проблемы и перспективы // Успехи соврем. биологии. Т. 129. № 3. С. 257–270.
Зинченко Т.Д., 2011. Эколого-фаунистическая характеристика хирономид (Diptera, Chhironomidae) малых рек бассейна Cредней и Нижней Волги (Атлас). Тольятти: Кассандра. 258 с.
Лисовский А.А., Дудов С.В., 2020. Преимущества и ограничения использования методов экологического моделирования ареалов. 2. MaxEnt // Журн. общ. биологии. Т. 81. № 2. С. 135–146.
Лисовский А.А., Дудов С.В., Оболенская Е.В., 2020. Преимущества и ограничения использования методов экологического моделирования ареалов. 1. Общие подходы // Журн. общ. биологии. Т. 81. № 2. С. 123–134.
Макарченко Е.А., Макарченко М.А., 1999. Chironomidae. Комары-звонцы // Определитель пресноводных беспозвоночных России и сопредельных территорий. Т. 4. Высшие насекомые. Двукрылые. СПб.: ЗИН РАН. С. 210–296.
Розенберг Г.С., Шитиков В.К., Зинченко Т.Д., 2020. Mark Vellend. The theory of ecological communities. Princeton; Oxford: Princeton University Press. 2016. 229 р. М. Велленд. Теория экологических сообществ. Принстон; Оксфорд: Изд-во Принстонского университета, 2016. 229 с. // Журн. общ. биологии. Т. 81. № 5. С. 394–400.
Шитиков В.К., Мастицкий С.Э., 2017. Классификация, регрессия и другие алгоритмы Data Mining с использованием R. Электронная книга. 351 с. https://stok1946.blogspot.com (дата обращения 10.10.2020).
Шитиков В.К., Розенберг Г.С., 2014. Рандомизация и бутстреп: статистический анализ в биологии и экологии с использованием R. Тольятти: Кассандра. 314 с.
Abrego N., Norberg A., Ovaskainen O., 2017. Measuring and predicting the influence of traits on the assembly processes of wood-inhabiting fungi // J. Ecol. V. 105. № 4. P. 1070–1081.
Araújo M.B., Anderson R.P., Barbosa A.M., Beale C.M., Dormann C.F. et al., 2019. Standards for distribution models in biodiversity assessments // Sci. Adv. V. 5. № 1. P. eaat4858.
Breiner F.T., Nobis M.P., Bergamini A., Guisan A., 2018. Optimizing ensembles of small models for predicting the distribution of species with few occurrences // Methods Ecol. Evol. V. 9. P. 802–808.
Brooker R.W., 2006. Plant-plant interactions and environmental change // New Phytol. V. 171. P. 271–284.
Busby J.R., 1991. BIOCLIM – a bioclimate analysis and prediction system // Plant Prot. Q. V. 6. P. 8–9.
Calabrese J.M., Certain G., Kraan C., Dormann C.F., 2014. Stacking species distribution models and adjusting bias by linking them to macroecological models // Glob. Ecol. Biogeogr. V. 23. P. 99–112.
Clark J.S., Gelfand A.E., Woodall C.W., Zhu K., 2014. More than the sum of the parts: Forest climate response from joint species distribution models // Ecol. Appl. V. 24. P. 990–999.
D’Amen M., Rahbek C., Zimmermann N. E., Guisan A., 2017. Spatial predictions at the community level: From current approaches to future frameworks // Biol. Rev. V. 92. P. 169–187.
Franklin J., 2009. Mapping Species Distributions: Spatial Inference and Prediction. Cambridge: Cambridge Univ. Press. 320 p.
Golovatyuk L.V., Shitikov V.K., Zinchenko T.D., 2018. Estimation of the zonal distribution of species of bottom communities in lowland rivers of the Middle and Lower Volga Basin // Biol. Bull. V. 45. № 10. P. 1262–1268.
Guisan A., Thuiller W., Zimmermann N.E., 2017. Habitat Suitability and Distribution Models: With Applications in R. Cambridge: Cambridge Univ. Press. 478 p.
Guisande C., Garcia-Rosello E., Heine J., Gonzalez-Dacosta J., Gonzalez-Vilas L. et al., 2017. SPEDInstabR: An algorithm based on a fuctuation index for selecting predictors in species distribution modeling // Ecol. Inform. V. 37. P. 18–23.
Hastie T., Fithian W., 2013. Inference from controversy // Ecography. V. 36. P. 864–867.
Hutchinson G.E., 1959. Homage to Santa Rosalia or Why are there so many kinds of animals? // Am. Nat. V. 43. № 870. P. 145–159.
Johnson D.H., 1980. The comparison of usage and availability measurements for evaluating resource preference // Ecology. V. 61. № 1. P. 65–71.
Kearney M.R., 2006. Habitat, environment and niche: What are we modelling? // Oikos. V. 115. № 1. P. 186–191.
Legendre P., Gallagher E., 2001. Ecologically meaningful transformations for ordination of species data // Oecologia. V. 129. P. 271–280.
Legendre P., Legendre L., 2012. Numerical Ecology. Amsterdam: Elsevier Sci. BV. 1006 p.
Norberg A., Abrego N., Blanchet F.G., Adler F.R., Anderson B.J. et al., 2019. A comprehensive evaluation of predictive performance of 33 species distribution models at species and community levels // Ecol. Monogr. V. 89. № 3. P. e01370.
Ovaskainen O., Abrego N., 2020. Species Distribution Modelling: With Applications in R. Cambridge: Cambridge Univ. Press. 370 p.
Ovaskainen O., Abrego N., Halme P., Dunson D., 2016a. Using latent variable models to identify large networks of species-to-species associations at different spatial scales // Methods Ecol. Evol. V. 7. P. 549–555.
Ovaskainen O., Roy D.B., Fox R., Anderson B.J., 2016b. Uncovering hidden spatial structure in species communities with spatially explicit joint species distribution models // Methods Ecol. Evol. V. 7. P. 428–436.
Ovaskainen O., Tikhonov G., Norberg A., Blanchet F.G., Duan L. et al., 2017. How to make more out of community data? A conceptual framework and its implementation as models and software // Ecol. Lett. V. 20. P. 561–576.
Peterson A.T., Soberón J., Pearson R.G., Anderson R.P., Martínez-Meyer E. et al., 2011. Ecological Niches and Geographic Distributions (MPB-49). Princeton: Princeton Univ. Press. 328 p.
Phillips S.J., Anderson R.P., Schapire R.E., 2006. Maximum entropy modeling of species geographic distributions // Ecol. Model. V. 190. № 3–4. P. 231–259.
Pollock L.J., Tingley R., Morris W.K., Golding N., O’Hara R.B. et al., 2014. Understanding co-occurrence by modelling species simultaneously with a Joint Species Distribution Model (JSDM) // Methods Ecol. Evol. V. 5. P. 397–406.
Thorson J.T., Ianelli J.N., Larsen E.A., Ries L., Scheuerell M.D. et al., 2016. Joint dynamic species distribution models: A tool for community ordination and spatio-temporal monitoring // Glob. Ecol. Biogeogr. V. 25. P. 1144–1158.
Tikhonov G., Abrego N., Dunson D., Ovaskainen O., 2017. Using joint species distribution models for evaluating how species-to-species associations depend on the environmental context // Methods Ecol. Evol. V. 8. P. 443–452.
Tikhonov G., Opedal Ø.H., Abrego N., Lehikoinen A., Jonge M.M.J., de, et al., 2020. Joint species distribution modelling with the R-package Hmsc // Methods Ecol. Evol. V. 11. P. 442–447.
Vellend M., 2016. The Theory of Ecological Communities. Princeton: Princeton Univ. Press. 224 p.
Warton D.I., Blanchet F.G., O’Hara R.B., Ovaskainen O., Taskinen S. et al., 2015. So many variables: Joint modeling in community ecology // Trends Ecol. Evol. V. 30. P. 766–779.
Zurell D., Thuiller W., Pagel J., Cabral J.S., Münkemüller T. et al., 2016. Benchmarking novel approaches for modelling species range dynamics // Glob. Change Biol. V. 22. № 8. P. 2651–2664.
Дополнительные материалы отсутствуют.
Инструменты
Журнал общей биологии