Молекулярная биология, 2023, T. 57, № 4, стр. 726-735
Пространственная реконструкция TRPC-механорецепторов гребневика Mnemiopsis leidyi A. Agassiz, 1865
А. В. Кузнецов a, b, Д. Н. Втюрина c, *
a Федеральный исследовательский центр Институт биологии южных морей им. А.О. Ковалевского
Российской академии наук
299011 Севастополь, Россия
b Севастопольский государственный университет
299053 Севастополь, Россия
c Федеральный исследовательский центр химической физики им. Н.Н. Семенова Российской академии наук
119991 Москва, Россия
* E-mail: vtyurinad@gmail.com
Поступила в редакцию 03.09.2022
После доработки 06.11.2022
Принята к публикации 22.11.2022
- EDN: QLQZZG
- DOI: 10.31857/S0026898423040122
Аннотация
Гребневик (Ctenophora) Mnemiopsis leidyi A. Agassiz, 1865 отвечает на слабое механическое раздражение интенсивной люминесценцией, однако механизм этого явления не известен. Нами проведен поиск возможных тактильных рецепторов, которые инициируют сигнальную трансдукцию, заканчивающуюся люминесценцией фотобелков. В геноме M. leidyi найдены три гена ортолога TRPC-белков мыши (5z96) и дрозофилы (5vkq) – ML234550a-PA (860 аа), ML03701a-PA (828 аа) и ML038011a-PA (1395 аа). Последний белок содержит длинную анкириновую спираль, состоящую из 16 ANK-доменов. Изучение аннотированных доменов и сети взаимодействий между белками интерактома позволяет предположить, что белки ML234550a-PA и ML03701a-PA осуществляют цитоплазматическую, а ML038011a-PA – внутриядерную трансдукцию механических сигналов. Пространственная реконструкция выявила различия в структуре этих белков, что может быть связано с их разными функциями в клетке. Обсуждается, какой из этих белков участвует в инициации люминесценции после механического раздражения.
ВВЕДЕНИЕ
Представители суперсемейства ионных каналов с транзиторным рецепторным потенциалом (TRP) – эволюционно древние сенсорные белки, которые регулируют проникновение катионов кальция в клетки [1]. Эти белки отвечают за восприятие тепла, физических контактов, болевых и вкусовых ощущений, а также иногда участвуют в зрительных процессах [2]. Большинство TRP-каналов объединены в две группы: первая группа включает катионные каналы TRPA (A – анкирин), TRPC (C – канонический), TRPN (N – нет механорецепторного потенциала C), TRPM (M – меластатин), TRPS (S – соромеластатин), TRPV (V – ванилоидный) и TRPVL (VL – ванилоидоподобный); вторая группа содержит TRPML (L – муколипин) и TRPP (P – поликистоз) [3]. Некоторые TRP-каналы активируются соединениями, содержащимися в чесноке (аллицин), перце (капсаицин), васаби (аллилизотиоцианат), другие чувствительны к камфоре, ментолу, мяте перечной или соединениям конопли [4]. Известны TRP-каналы, которые действуют как датчики вибрации, растяжения и осмотического давления [5]. Большинство ионных каналов регулируется окружающими их липидами клеточной мембраны [6].
Мы предположили, что биологические функции TRP-рецепторов могут модулироваться “соседними” белками и низкомолекулярными лигандами, а спектральная чувствительность этих сенсоров зависит от числа анкириновых повторов (AR), что позволяет им выполнять различные функции – от терморецепторов с короткими анкириновыми спиралями и высокой резонансной частотой до тактильных рецепторов с длинными анкириновыми спиралями и низкой резонансной частотой макромолекулы [7]. В представленном исследовании проведен анализ трех белков гребневика Mnemiopsis leidyi, возможных кандидатов на роль механорецепторов. M. leidyi, геном которого полностью секвенирован [8], входит в число базальных Metazoa [9–11], которые могут использоваться при изучении эволюции сенсорных систем [12, 13].
Цель нашей работы состояла в 3D-реконструкции TRP-рецепторов гребневика M. leidyi. С использованием данных [7] в геноме M. leidyi найдены гомологичные гены и определены аминокислотные последовательности белков, кодируемых этими генами, что позволило описать доменную структуру белков, выявить их ближайших соседей в сети белок-белковых взаимодействий и восстановить трехмерную структуру исследуемых полипептидов. Такой большой класс механорецепторов, как пьезобелки [14], мы не рассматривали, так как гены этих белков в геноме M. leidyi предположительно отсутствуют (данные не представлены).
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Аминокислотные последовательности механорецепторов гребневика M. leidyi найдены в базе данных NCBI [8].
Множественное выравнивание полипептидных цепей и построение филогенетического дерева выполнено с помощью программ Clustal Omega (1.2.4) [15] и TreeDyn 198.3 [16] соответственно.
Профили гидрофобности по Kyte-Doolittle определяли с помощью программы ProtScale [17].
Анализ доменной структуры белков осуществляли на сервере Pfam 35.0 [18], взаимодействие доменов исследовали и визуализировали с помощью сервера STRING (Protein–Protein Interaction Networks Functional Enrichment Analysis), согласно [19].
Построение пространственных моделей механорецепторов проводили на сервере Phyre2, используя метод сравнения гомологичных аминокислотных последовательностей с известными пространственными структурами белков. Оценивали достоверность и степень покрытия исследуемых последовательностей [20].
Визуализацию молекулярных структур выполняли с использованием программ RasMol [21] и UCSF Chimera [22].
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
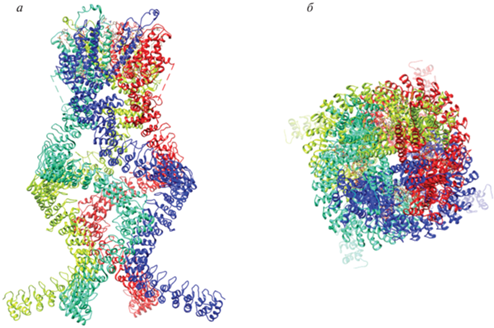
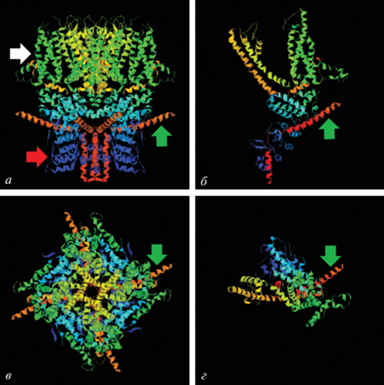
В основу нашего исследования положены знания о классическом механорецепторе NompC (5vkq) Drosophila melanogaster [23]. NompC входит в семейство TRPN-белков, включающих трансмембранный домен, область шейки и четыре анкириновых спирали. Проницаемость канала для ионов Ca2+ изменяется под воздействием механической нагрузки (рис. 1).
Рис. 1.
Гомотетрамерная структура механотрансдуцирующего ионного канала NompC (5vkq) плодовой мушки Drosophila melanogaster. а – Вид сбоку, б – вид сверху. Отдельные полипептидные цепи показаны различными цветами, видна полость канала; визуализация с помощью UCSF Chimera [22].
Поиск последовательностей, гомологичных NompC, первоначально выявил лишь один аннотированный TRPC-белок, кодируемый геномом M. leidyi, а именно, белок AFK83798.1 (ML234550a-PA) с полипептидной цепью из 860 аа (рис. S1 , см. Дополнительные материалы на сайте http://www.molecbio.ru/downloads/2023/4/supp_ Kuznetsov_rus.pdf) и трансмембранными участками, расположенными, начиная примерно с позиции 400 аа, в направлении к C-концу полипептидной цепи (рис. 2). Детальный анализ показал, что белок ML234550a-PA включает три анкириновых повтора в положениях 125–160, 210–239 и 245–274, каждый из которых состоит в среднем из 31 аа и участвует в белок-белковых взаимодействиях; далее следует область трансмембранных спиралей, которые начинаются в позиции 413 и заканчивается в позиции 435; ближе к концу полипептидной цепи в положении 485–723 расположен ионный канал, имеющий шесть трансмембранных α-спиралей, две последние из которых окружают петлю, определяющую ионную селективность (рис. 3а).
Рис. 2.
График гидропатии белка AFK83798.1 (ML234550a-PA) гребневика M. leidyi по Kyte-Doolittle [17], построенный с использованием модели линейного изменения веса, где размер окна равен 9, а области с положительным значением являются гидрофобными.
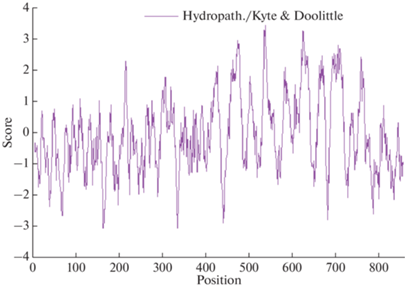
Рис. 3.
Доменные структуры белков ML234550a-PA (а), ML03701a-PA (б) и ML038011a-PA (в) гребневика M. leidyi. Шкалы внизу с шагом 100 соответствуют числу аминокислотных остатков, трансмембранные участки обозначены синим цветом, неструктурированные области – малиновым, анкириновые домены маркированы как ANK, катионный канал PKD_channel (Polycystic Kidney Disease Channel) и участок Ion_trans (Ionic Transmembrane Domain) представлены большими прозрачными прямоугольниками [19].
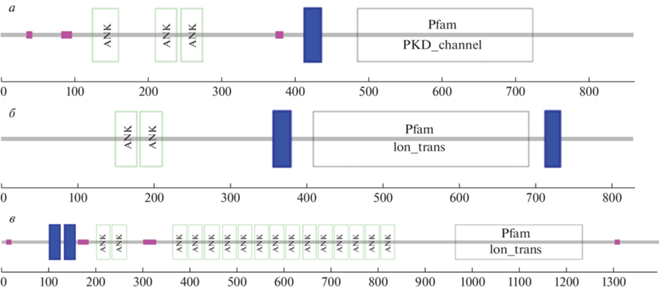
Дальнейший углубленный поиск с использованием аминокислотной последовательности AFK83798.1 в качестве запроса показал, что белок ML234550a-PA имеет неаннотированные гомологи ML03701a-PA (828 аа) и ML038011a-PA (1395 аа), кодируемые геномом M. leidyi (рис. S2 , см. Дополнительные материалы на сайте http://www.molecbio.ru/downloads/2023/4/supp__Kuznetsov_rus.pdf). Оказалось, что белок ML03701a-PA длиной 828 aа, содержит два ANK-домена в позициях 150–178, 182–211 и ионный канал в положении 409–691, фланкированный трансмембранными α-спиралями с координатами 357–379 и 713–732 (рис. 3б). Другой белок, а именно ML038011a-PA, длиной 1395 aa включает два трансмембранных участка в положениях 102–124 и 134–156, два близкорасположенных ANK-домена в позициях 201–230, 234–265 и ниже еще 14 ANK-доменов в положении с 363 по 834, а также ионный канал в положении 963–1232 (рис. 3в).
Множественное выравнивание TRP-белков выявило значительные различия в их N-концевой области, где находятся анкириновые повторы, не столь заметные различия в C-концевой части и достаточную консервативность средней части аминокислотной последовательности, где располагается ионпроводящий канал (данные не представлены). Проведенный далее филогенетический анализ выявил сходство между белком 5z96 мыши и белками ML234550a-PA и ML03701a-PA мнемиопсиса с малым числом анкириновых последовательностей, а также между белком 5vkq дрозофилы и ML038011a-PA мнемиопсиса с длинными анкириновыми спиралями (рис. S3 , см. Дополнительные материалы на сайте http://www.molecbio.ru/downloads/2023/4/ supp_Kuznetsov_rus.pdf).
Изучение взаимодействий первого порядка белка ML234550a-PA (рис. 4а) установило его разнообразные отношения (совместное появление в разных геномах, коэкспрессия и др., общий балл 0.771) с гомологом ML03701a-PA (238.8 бит для 552 аминокислот), а также с еще девятью белками, в том числе с фосфолипазой С (ML04921a-PA). Два белка – ML047931a-PA, содержащий домены SAM (Sterile Alpha Motif) и SOAR (STIM1 Orai1-Activating Region), а также ML07264a-PA с PKD-полицистиновым каналом, отличаются слабой связностью. Остальные белки имеют высокую связность, что означает высокую вероятность специфического взаимодействия по данным сервера STRING. Так, белок ML017411a-PA содержит домены FKBP_C (FKBP-type peptidyl-prolyl сis-trans isomerase, FK506-Binding Protein, FK506 – drug tacrolimus) и TPR_2 (Tetratrico Peptide Repeat); ML04921a-PA включает домены EF-hand (helix-loop-helix Ca2+ binding motif; the index finger and thumb represent the E- and F-helices, respectively), PLCXc, PLCYc (phospholipase C) и C2 (Ca2+-dependent membrane-targeting module); ML13777a-PA – пять доменов PDZ (binding to C-terminal peptide of other proteins); ML13931a-PA – два C1-домена и по одному домену C2 (domains of protein kinase C), S_Tkc и S_TK_X (Ser/Thr-type protein kinases); белок ML14122a-PA содержит три домена PDZ, а ML46823a-PA – один домен 7TM_GPCR_Srsx (7 transmembrane G-protein-coupled receptor class of proteins which are of the serpentine type).
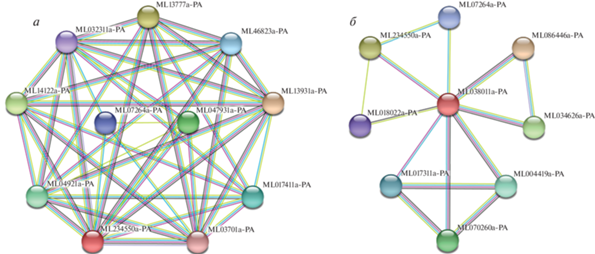
Рис. 4.
Графы белок-белковых взаимодействий первого порядка для TRPC-белка AFK83798.1 (ML234550a-PA) (а) и гипотетического белка ML038011a-PA (б) из M. leidyi, построенные с помощью сервера STRING согласно [19].
Рассматривая коэкспрессию ортологов ML234550a-PA в других организмах, можно отметить, что TRP-белок переходного рецепторного потенциала D. melanogaster – это светочувствительный кальциевый канал для проникновения Ca2+ в сетчатку во время опосредованной фосфолипазой C (PLC) фототрансдукции. Этот белок необходим для способности видеть в темноте и при тусклом свете, он связывает кальмодулин, активируется инозитом, жирными кислотами и GTP-связывающими белками (показатель коэкспрессии 0.410).
Белок ML038011a-PA вступает во множественные взаимодействия с белками ML086446a-PA, ML034626a-PA, ML004419a-PA и ML070260a-PA, а также в менее тесные отношения с ML234550a-PA особенно с полипептидами ML017311a-PA, ML018022a-PA и ML07264a-PA (рис. 4б). Так, белок ML086446a-PA содержит рецепторный и дополнительный тирозинкиназный домены; белок ML034626a-PA включает три Hr1-домена (rho-binding domain) и каталитические домены серин/треониновой протеинкиназы; в белке ML004419a-PA имеются четыре PH-домена (Pleckstrin homology domain) и домен гистондеацетилазы [EC 3.5.1.98]; белок ML070260a-PA содержит ДНК-связывающий домен WAC_Acf1 (ATP-utilising chromatin assembly and remodelling factor), WSD-мотив (Williams-Beuren syndrome), PH-домен с мотивом цинкового пальца и бромодомен, взаимодействующий с гистонами, ацетилированными по остаткам лизина. Интересно также, что белок ML038011a-PA непосредственно взаимодействует со своим гомологом ML234550a-PA, известным как AFK83798.1. Белок ML017311a-PA, возможно, ассоциирован с хроматином, так как имеет три бромодомена и BAH-гомолог (bromo-adjacent homology). Белок ML018022a-PA является пьезобелком с семью трансмембранными доменами и характерным пьезодоменом. Белок ML07264a-PA входит в число полицистиновых катионных PKD-каналов.
С целью понимания сенсорной роли TRPC-белка AFK83798.1 (ML234550a-PA) из M. leidyi в проведении сигналов о механической нагрузке была предпринята попытка определить его трехмерную структуру. Гомологическое сравнение аминокислотной последовательности AFK83798.1 с известными трехмерными структурами белков позволило выявить пространственное положение 631 аа за исключением N- и C-концов, что составляет 73% аминокислотной последовательности белка, на основе единственного шаблона С-цепи мембранного ионного канала TRPC4 (5z96) мыши [24]. Помимо этого, использовали дополнительные шаблоны, которые позволили охватить другие части исследуемого белка. В частности, в качестве одного из шаблонов использовали исходный механорецептор NompC (5vkq) D. melanogaster [23]. С помощью нескольких шаблонов смоделированы пространственные координаты 801 аа с достоверностью более 90%, что соответствует 93% всего белка AFK83798.1. На рис. 5 можно видеть, что пространственная модель единственной полипептидной цепи белка AFK83798.1 гребневика M. leidyi напоминает отдельную субъединицу TRPC-канала мыши.
Рис. 5.
Пространственная структура тетрамера мембранного TRPC-канала 5z96 домовой мыши (а, в) и 3D-модели одной субъединицы TRPC-белка AFK83798.1 M. leidyi (б, г). Вид сбоку (а, б), вид сверху (в, г), использована спектральная окраска от N- до C-концевой части белка; белая стрелка указывает на трансмембранный домен, красная – на анкириновые повторы, которые располагаются в цитоплазме и формируют “пружину”, зеленая – на α-спираль с изломом, визуализация в RasMol [21].
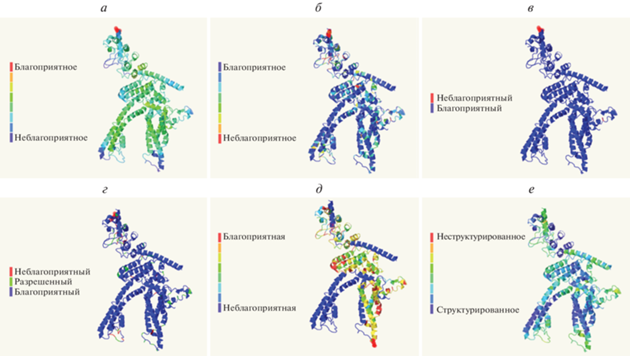
Моделирование с применением специальных алгоритмов позволило детально оценить достоверность модели. Так, с использованием ProQ2-алгоритма [25] достигнут средний уровень качества локального и глобального прогнозирования белковых моделей (рис. 6а). В силу того, что отдельные атомы в некоторых аминокислотных остатках могут располагаться в модели слишком близко друг к другу, большое количество пересечений может означать неудовлетворительное расположение смежных цепей или, возможно, неправильную укладку в этой области. Поэтому критерий пересечений является особенно строгим. Характер окрашивания аминокислотных остатков по количеству наблюдаемых пересечений говорит о хорошем качестве модели (рис. 6б). Ротамерный анализ также свидетельствует о том, что базовое выравнивание боковых цепей в модели осуществлено правильно (рис. 6в). Подавляющее число аминокислотных остатков в модели находится в благоприятных областях (синий), лишь несколько остатков локализуются в разрешенных (зеленый) областях (рис. 6г). Ничего не отмечено в запрещенных областях (красный) диаграммы Рамачандрана, что указывает на корректность расчетов phi-psi торсионных углов полипептидного остова. Достоверность парного выравнивания запрос-шаблон по данным Hhsearch, полученная из апостериорных вероятностей, которые рассчитывали в алгоритме Forward-Backward [26], в большинстве случаев была хорошей или удовлетворительной (рис. 6д). Так как неупорядоченные и динамически гибкие области играют важную роль в функционировании многих белков, предсказание неструктурированных областей было дополнено с помощью метода Disopred [27, 28] с приемлемым результатом (рис. 6е).
Рис. 6.
Оценка качества пространственной модели TRPC-белка AFK83798.1 из M. Leidyi. а – Локальное и глобальное прогнозирование; б – пересечения; в – ротамерный анализ; г – анализ Рамачандрана; д – достоверность выравнивания; е – предсказание неструктурированных областей. Локальное качество моделирования отмечено цветом на каждой части – от синего до красного, а изображения субъединицы белка перевернуты по сравнению с рис. 5б.
Найденные аминокислотные последовательности и созданные пространственные структуры гипотетических механосенсоров ML03701a-PA и ML038011a-PA гребневика M. leidyi приведены на рис. S3 (см. Дополнительные материалы на сайте http://www.molecbio.ru/downloads/2023/4/supp_ Kuznetsov_rus.pdf). К сожалению, длина белка ML038011a-PA (1395 аа) (рис. S2 ) не позволила загрузить задачу на сервер Phyre2 и построить полную трехмерную модель. Поэтому было решено удалить часть анкириновой спирали из исходного файла с координатами атомов этого белка для построения урезанной 3D-модели (рис. S4 б). Следует отметить, что помимо длинной анкириновой спирали, гипотетический белок ML038011a-PA отличается от ML03701a-PA и ML234550a-PA отсутствием цитоплазматической α-спирали с изломом, одна часть которой следует в направлении канала, а другая обращена под углом наружу тетрамерного комплекса (рис. 5, рис. S4 а – отмечены зеленым цветом).
Кроме того, гипотетические белки ML03701a-PA и ML038011a-PA не взаимодействуют непосредственно друг с другом, но связываются с исходным белком ML234550a-PA (AFK83798.1) (рис. 4). Оба полипептида ML234550a-PA и ML03701a-PA имеют цитоплазматические α-спирали с изломом и короткие анкириновые спирали, как и мембранный канал TRPC4 (5z96) мыши, являющийся температурным сенсором [24], тогда как ML038011a-PA обладает структурным сходством с тактильным сенсором NompC (5vkq) дрозофилы [23], поскольку имеет длинные анкириновые спирали и не содержит надломленных α-спиралей (рис. 1, 5, рис. S4 Дополнительных материалов).
Помимо этого, белок ML038011a-PA взаимодействует с пьезобелком ML018022a-PA (рис. 4б), что может приводить к образованию надмолекулярного тактильного комплекса. Последний факт подтверждает нашу гипотезу, согласно которой биологическая функция TRP-рецепторов модулируется соседними белками. Влияние низкомолекулярных лигандов будет в дальнейшем проверено экспериментально. То, что спектральная чувствительность этих сенсоров может зависеть от длины анкиринового повтора, следует из механических моделей [7]. Кроме того, анализ доменов показал, что ML038011a-PA способен опосредованно связываться с белками хроматина, в то время как ML234550a-PA взаимодействует преимущественно с цитоплазматическими белками, что, возможно, позволяет первому белку осуществлять передачу сигнала в ядро, а второму в цитоплазму клетки.
Следует отметить, что скорость передачи сигнала посредством белок-белковых взаимодействий намного ниже наблюдаемой реакции мнемиопсисов в виде люминесценции на механическое возмущение. Кроме того, фотобелки не имеют взаимодействующих доменов (данные не представлены). Следовательно, роль TRPC-каналов в передаче фотобелкам сигнала, вызванного механическим раздражением, связана со способностью каналов пропускать такие катионы, как Ca2+ [29], и инициировать деполяризацию потенциала покоя клеточной мембраны, которая приводит к быстрому распространению волны активации вдоль поверхности клетки и к передаче сигнала от клетки к клетке.
ЗАКЛЮЧЕНИЕ
Появление крупных инноваций и диверсификация в биологии происходят благодаря модульной эволюции [30], при этом сенсорные системы, лежащие в основе функционирования нервной системы, зародились еще на ранних этапах развития жизни [31]. TRP-каналы с транзиторным рецепторным потенциалом образуют большое семейство мультимодальных молекулярных сенсоров, которые реагируют на широкий спектр стимулов, включая тепловые, механические и химические [32]. TRP-каналы обнаружены у большинства эукариот, подробно описаны у млекопитающих, но лучше всего изучены у нематоды и дрозофилы, служащих генетическими моделями [7]. При этом построение пространственных моделей мембранных белков, в том числе TRP-каналов, остается сложной задачей, несмотря на успехи в рентгеновской кристаллографии, ядерном магнитном резонансе и криогенной электронной микроскопии [33]. Создание полноразмерных гомологических структурных моделей затрудняется субъединичной природой и большим размером TRP-каналов, имеющих длинные анкириновые спирали. Кроме того, требуются дополнительные методы проверки надежности моделей [25–28].
Суперсемейство белков катионных TRP-каналов примечательно бόльшим разнообразием механизмов активации и селективности, чем у любой другой группы белков ионных каналов. TRP-белки играют решающую роль в сенсорной физиологии зрения, обоняния, вкуса, осязания, слуха, термо- и осмоощущений, а также позволяют отдельным клеткам ощущать изменения в локальной среде. Многие TRP-каналы активируются множеством различных стимулов и функционируют как интеграторы сигналов [34] (биофизические и структурно-функциональные исследования термочувствительных TRP-каналов обобщены в обзоре [35]).
Нами впервые построены 3D-модели TRP-субъединиц механорецепторов гребневика M. leidyi (Ctenofora). На основании пространственной реконструкции трудно предложить однозначную функцию белков ML234550a-PA (AFK83798.1) и ML03701a-PA с короткой анкириновой последовательностью. Однако гребневик M. leidyi отвечает на механические раздражения интенсивной люминесценцией, поэтому можно предположить, что роль тактильного сенсора, при отсутствии активности других кандидатов [36], выполняет гомологичный гипотетический белок ML038011a-PA с длинными анкириновыми спиралями. Имеющиеся данные не раскрывают полностью связь между механическим раздражением и люминесценцией у M. leidyi, но наглядно показывают сложность сигнальных комплексов и запутанность путей трансдукции, возникших в ходе эволюционного развития, что принципиально отличает их от технических систем, созданных человеком.
Можно отметить, что сенсорная активность механочувствительных рецепторов особенно важна для гидробионтов, так как вода практически не сжимаема по сравнению с воздухом и в ней лучше передаются волны давления, что мы видим на примере люминесценции гребневиков при турбуленции. Возможно, механорецепторы, как TRP-каналы, которые появились на ранних этапах эволюции [37], играют бόльшую роль у морских животных по сравнению с сухопутными [38, 39]. При выходе жизни на сушу часть функций была утрачена, а часть модифицирована к новой среде обитания в результате диверсификации TRP-каналов [40].
Авторы благодарят М.В. Фадееву и Е.В. Сергееву за помощь в работе, О.В. Машукову, Б.Е. Аннинского, С.А. Царина, О.С. Завьялову за поддержку, К.А. Рыбакову, В.Д. Ратникова, Е.В. Моцарь и М.И. Силакова за обсуждение рукописи и рекомендации, а также анонимных рецензентов за полезные замечания, позволившие значительно улучшить манускрипт.
Работа выполнена в рамках Государственного задания ФИЦ ИнБЮМ тема № 0828-2018-0002.
Настоящая статья не содержит описания каких-либо исследований с участием людей или животных в качестве объектов.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Himmel N.J., Cox D.N. (2020) Transient receptor potential channels: current perspectives on evolution, structure, function and nomenclature. Proc. Biol. Sci. 287(1933), 20201309. https://doi.org/10.1098/rspb.2020.1309
Cao E. (2020) Structural mechanisms of transient receptor potential ion channels. J. Gen. Physiol. 152(3), e201811998. https://doi.org/10.1085/jgp.201811998
Samanta A., Hughes T.E., Moiseenkova-Bell V.Y. (2018) Transient receptor potential (TRP) channels. Subcell. Biochem. 87, 141‒165. https://doi.org/10.1007/978-981-10-7757-9_6
Nilius B., Owsianik G. (2011) The transient receptor potential family of ion channels. Genome Biol. 12(3), 218. https://doi.org/10.1186/gb-2011-12-3-218
Lehnert B.P., Santiago C., Huey E.L., Emanuel A.J., Renauld S., Africawala N., Alkislar I., Zheng Y., Bai L., Koutsioumpa C., Hong J.T., Magee A.R., Harvey C.D., Ginty D.D. (2021) Mechanoreceptor synapses in the brainstem shape the central representation of touch. Cell. 184(22), 5608‒5621. https://doi.org/10.1016/j.cell.2021.09.023
Robinson C.V., Rohacs T., Hansen S.B. (2019) Tools for understanding nanoscale lipid regulation of ion channels. Trends Biochem. Sci. 44(9), 795‒806. https://doi.org/10.1016/j.tibs.2019.04.00
Liang X., Sun L., Liu Z. (2017) Mechanosensory transduction in Drosophila melanogaster. Singapore: Springer, pp. 82. https://doi.org/10.1007/978-981-10-6526-2
Ryan J.F., Pang K., Schnitzler C.E., Nguyen A.D., Moreland R.T., Simmons D.K., Koch B.J., Francis W.R., Havlak P., NISC Comparative Sequencing Program; Smith S.A., Putnam N.H., Haddock S.H., Dunn C.W., Wolfsberg T.G., Mullikin J.C., Martindale M.Q., Baxevanis A.D. (2013) The genome of the ctenophore Mnemiopsis leidyi and its implications for cell type evolution. Science. 342(6164), 1242592. https://doi.org/10.1126/science.1242592
Moroz L.L. (2015) Convergent evolution of neural systems in ctenophores. J. Exp. Biol. 218(4), 598‒611. https://doi.org/10.1242/jeb.110692
Moroz L.L., Kohn A.B. (2016) Independent origins of neurons and synapses: insights from ctenophores. Philos. Trans. R. Soc. B. 371(1685), 20150041. https://doi.org/10.1098/rstb.2015.0041.
Moroz L.L. (2021) Multiple origins of neurons from secretory cells. Front. Cell Dev. Biol. 9, 669087. https://doi.org/10.3389/fcell.2021.669087
Aronova M.Z. (2009) Structural models of “simple” sense organs by the example of the first Metazoa. J. Evol. Biochem. Phys. 45(2), 179‒196. https://doi.org/10.1134/S0022093009020017
Jékely G., Godfrey-Smith P., Keijzer F. (2021) Reafference and the origin of the self in early nervous system evolution. Philos. Trans. R. Soc. B. 376(1821), 20190764. https://doi.org/10.1098/rstb.2019.0764
Bagriantsev S.N., Gracheva E.O., Gallagher P.G. (2014) Piezo proteins: regulators of mechanosensation and other cellular processes. J. Biol. Chem. 289(46), 31673‒31681. https://doi.org/10.1074/jbc.R114.612697
Madeira F., Park Y.M., Lee J., Buso N., Gur T., Madhusoodanan N., Basutkar P., Tivey A.R.N., Potter S.C., Finn R.D., Lopez R. (2019) The EMBL-EBI search and sequence analysis tools APIs in 2019. Nucl. Acids Res. 2(47), W636‒W641. https://doi.org/10.1093/nar/gkz268
Chevenet F., Brun C., Bañuls A.L., Jacq B., Christen R. (2006) TreeDyn: towards dynamic graphics and annotations for analyses of trees. BMC Bioinformatics. 10(7), 439. https://doi.org/10.1186/1471-2105-7-439
Kyte J., Doolittle R.F. (1982) A simple method for displaying the hydropathic character of a protein. J. Mol. Biol. 157(1), 105‒132. https://doi.org/10.1016/0022-2836(82)90515-0
Mistry J., Chuguransky S., Williams L., Qureshi M., Salazar G.A., Sonnhammer E.L., Tosatto S.C.E., Paladin L., Raj S., Richardson L.J., Finn R.D., Bateman A. (2021) Pfam: The protein families database in 2021. Nucl. Acids Res. 49(D1), D412‒D419. https://doi.org/10.1093/nar/gkaa913
Szklarczyk D., Gable A.L., Nastou K.C., Lyon D., Kirsch R., Pyysalo S., Doncheva N.T., Legeay M., Fang T., Bork P., Jensen L.J., von Mering C. (2021) The STRING database in 2021: customizable protein–protein networks, and functional characterization of user-uploaded gene/measurement sets. Nucl. Acids Res. 49(D1), D605‒D612. https://doi.org/10.1093/nar/gkaa1074
Kelley L.A., Mezulis S., Yates C.M., Wass M.N., Sternberg M.J. (2015) The Phyre2 web portal for protein modeling, prediction and analysis. Nat. Protoc. 10(6), 845‒858. https://doi.org/10.1038/nprot.2015.053
Sayle R.A., Milner-White E.J. (1995) RASMOL: biomolecular graphics for all. Trends Biochem. Sci. 20(9), 374‒376. https://doi.org/10.1016/S0968-0004(00)89080-5
Pettersen E.F., Goddard T.D., Huang C.C., Couch G.S., Greenblatt D.M., Meng E.C., Ferrin T.E. (2004) UCSF Chimera – a visualization system for exploratory research and analysis. J. Comput. Chem. 25(13), 1605‒1612. https://doi.org/10.1002/jcc.20084
Jin P., Bulkley D., Guo Y., Zhang W., Guo Z., Huynh W., Wu S., Meltzer S., Cheng T., Jan L.Y., Jan Y.N., Cheng Y. (2017) Electron cryo-microscopy structure of the mechanotransduction channel NOMPC. Nature. 547(7661), 118‒122. https://doi.org/10.1038/nature22981
Duan J., Li J., Zeng B., Chen G.L., Peng X., Zhang Y., Wang J., Clapham D.E., Li Z., Zhang J. (2018) Structure of the mouse TRPC4 ion channel. Nat. Commun. 9(1), 1‒10. https://doi.org/10.1038/s41467-018-05247-9
Ray A., Lindahl E., Wallner B. (2012) Improved model quality assessment using ProQ2. BMC Bioinform. 13(1), 1‒12. https://doi.org/10.1186/1471-2105-13-224
Russell S., Norvigb P. (2010) Intelligence Artificielle: Avec Plus de 500 Exercices. Pearson Education, France.
Ward J.J., McGuffin L.J., Bryson K., Buxton B.F., Jones D.T. (2004) The DISOPRED server for the prediction of protein disorder. Bioinformatics. 20(13), 2138‒2139. https://doi.org/10.1093/bioinformatics/bth195
Jones D.T., Cozzetto D. (2015) DISOPRED3: precise disordered region predictions with annotated protein-binding activity. Bioinformatics. 31(6), 857‒863. https://doi.org/10.1093/bioinformatics/btu744
Perissinotti P.P., Martínez-Hernández E., Piedras-Rentería E.S. (2021) TRPC1/5-Cav3 complex mediates leptin-induced excitability in hypothalamic neurons. Front. Neurosci. 15, 679078. https://doi.org/10.3389/fnins.2021.679078
Watson R.A. (2006) Compositional Evolution: The Impact of Sex, Symbiosis, and Modularity on the Gradualist Frame-work of Evolution. Vienna Series in Theoretical Biology: A Bradford Book. 344 p. ISBN-10: 9780262232432
Oteiza P., Baldwin M.W. (2021) Evolution of sensory systems. Curr. Opin. Neurobiol. 71, 52‒59. https://doi.org/10.1016/j.conb.2021.08.005
Li H. (2017) TRP channel classification. Adv. Exp. Med. Biol. 976, 1‒8. https://doi.org/10.1007/978-94-024-1088-4_1
Hellmich U.A., Gaudet R. (2014) Structural biology of TRP channels. Handb. Exp. Pharmacol. 223, 963‒990. https://doi.org/10.1007/978-3-319-05161-1_10
Venkatachalam K., Montell C. (2007) TRP channels. Annu. Rev. Biochem. 76, 387‒417. https://doi.org/10.1146/annurev.biochem.75.103004.142819
Voets T. (2012) Quantifying and modeling the temperature-dependent gating of TRP channels. Rev. Physiol. Biochem. Pharmacol. 162, 91‒119. https://doi.org/10.1007/112_2011_5
Coste B., Mathur J., Schmidt M., Earley T.J., Ranade S., Petrus M.J., Dubin A.E., Patapoutian A. (2010) Piezo1 and Piezo2 are essential components of distinct mechanically activated cation channels. Science. 330(6000), 55‒60. https://doi.org/10.1126/science.1193270
Peng G., Shi X., Kadowaki T. (2015) Evolution of TRP channels inferred by their classification in diverse animal species. Mol. Phylogenet. Evol. 84, 145‒157. https://doi.org/10.1016/j.ympev.2014.06.016
Voets T., Nilius B. (2003) TRPs make sense. J. Membr. Biol. 192(1), 1‒8. https://doi.org/10.1007/s00232-002-1059-8
Voets T., Talavera K., Owsianik G., Nilius B. (2005) Sensing with TRP channels, Nat. Chem. Biol. 1(2), 85‒92. https://doi.org/10.1038/nchembio0705-85
Kadowaki T. (2015) Evolutionary dynamics of metazoan TRP channels. Pflugers Arch. 467(10), 2043‒2053. https://doi.org/10.1007/s00424-015-1705-5
Дополнительные материалы
- скачать ESM.zip
- Приложение 1. Рис. S1. - Рис. S4.
Инструменты
Молекулярная биология


