Доклады Российской академии наук. Науки о жизни, 2023, T. 509, № 1, стр. 155-160
Филогения видового комплекса Dipus sagitta по результатам секвенирования ядерных генов
А. А. Лисенкова 1, *, В. С. Лебедев 2, Э. Ундрахбаяр 3, В. Ю. Богатырева 2, М. Н. Мельникова 1, Р. А. Назаров 2, К. А. Роговин 4, член-корреспондент РАН А. В. Суров 4, Г. И. Шенброт 5, А. А. Банникова 1
1 Биологический факультет Московского государственного университета
имени М.В. Ломоносова
Москва, Россия
2 Зоологический музей Московского государственного университета имени М.В. Ломоносова
Москва, Россия
3 Институт общей и экспериментальной биологии Монгольской Академии Наук
Улан-Батор, Монголия
4 Институт проблем экологии и эволюции
им. А.Н. Северцова Российской академии наук
Москва, Россия
5 Mitrani Department of Desert Ecology, Jacob Blaustein Institutes for Desert Research, Ben-Gurion University
of the Negev
Midreshet, Ben-Gurion, Israel
* E-mail: biolisenkova@yandex.ru
Поступила в редакцию 10.12.2022
После доработки 16.12.2022
Принята к публикации 19.12.2022
- EDN: NBKKVY
- DOI: 10.31857/S2686738922700159
Аннотация
Мохноногий тушканчик Dipus sagitta длительное время считался единым политипическим видом. Ранее на основании митохондриальных и нескольких ядерных генов было выявлено высокое генетическое разнообразие D. sagitta и выдвинута гипотеза о существовании в его пределах нескольких видов. Однако небольшое число ядерных генов не позволило установить взаимоотношения между выявленными филогенетическими линиями. Мы значительно расширили набор используемых локусов ядерной ДНК, что привело к достаточно высокому разрешению филогенетического дерева для 10 форм D. sagitta. Выявленная структура вида в основном подтвердила характер и взаимоотношения линий мтДНК. Вместе с тем нельзя говорить о полном соответствии митохондриальной и ядерной филогений, часть генетических линий D. sagitta является, вероятно, продуктом ретикулярных процессов. Таким образом, этот таксон представляет собой сложный видовой комплекс D. sagitta sensu lato, в котором давно разошедшиеся линии не всегда репродуктивно изолированы.
ВВЕДЕНИЕ
Род Dipus Zimmermann, 1780 (Dipodidae, Mammalia) долгое время считался монотипическим и включал в себя единственный широкоареальный вид мохноногого тушканчика Dipus sagitta (Pallas, 1773) с 12–15 подвидами, распространенными в полупустынных и пустынных областях умеренной зоны Евразии от Волгоградской области России до Китая. В результате морфологической ревизии, проведенной Г.И. Шенбротом [1, 2], в составе Dipus sagitta были выделены две хорошо дифференцированные видовые группы: “lagopus” и “sagitta”. Группа “lagopus” населяет западную часть видового ареала мохноногого тушканчика, которая охватывает территории Европейской части России, Казахстана и Турана, и включает семь подвидов: D. s. nogai Satunin, 1907; D. s. innae Ognev, 1930; D. s. austrouralensis Shenbrot, 1991; D. s. lagopus Lichtenstein, 1823; D. s. turanicus Shenbrot, 1991; D. s. megacranius Shenbrot, 1991; D. s. usuni Shenbrot, 1991 [2]. Группа “sagitta” обитает в восточной части, на территории Центральной Азии и включает пять подвидов: D. s. sagitta (Pallas, 1773); D. s. zaissanensis Selevin, 1934; D. s. bulganensis Shenbrot, 1991; D. s. sowerbyi Thomas, 1908; D. s. halli Sowerbi, 1920 [1]. Кроме того, с последней группой сближали три подвида из Таримской и Цайдамской котловин (Китай): D. s. deasyi Barret-Hamilton, 1900, D. s. aksuensis Wang, 1964 и D. s. fuscocanus Wang, 1964.
Исследования генетической изменчивости D. sagitta s.l. на основе митохондриальных генов показали наличие нескольких линий потенциально видового уровня [3–5], наиболее дивергентной из которых – китайскому подвиду D. s. deasyi – было предложено присвоить видовой статус. Для разных выделяемых таксонов и групп были отмечены как совпадения, так и отличия от результатов морфологической ревизии. Однако имеющиеся данные по совокупно шести ядерным генам не позволили получить статистически значимую филогенетическую реконструкцию и, вместе с тем, указали на события интрогрессии мтДНК [3–5].
Учитывая, что митохондриальные филогении часто не соответствуют таковым, построенным на основании ядерных генов, для подтверждения существования генетических линий и установления уровня их дивергенции мы решили увеличить число ядерных локусов в нашем филогенетическом анализе D. sagitta s.l. Целью нашего исследования таким образом стали получение разрешенного филогенетического дерева для форм видового комплекса мохноногого тушканчика при помощи набора из фрагментов 26 ядерных генов и сравнение полученной топологии с морфологической ревизией и филогенией на основе митохондриальных данных.
МАТЕРИАЛЫ И МЕТОДЫ
Для исследования были выбраны 18 представителей видового комплекса Dipus sagitta s.l., принадлежащие различным подвидам (рис. 1). Выделение ДНК, амплификацию и секвенирование по Сэнгеру проводили согласно опубликованым ранее методам [6, 7]. Были амплифицированы фрагменты интронов и экзонов 26 ядерных генов с использованным как опубликованных ранее, так и разработанных впервые праймеров (в скобках указана длина ПЦР-продукта): A2AB (541 п.о.), ATP7A (724 п.о.), CREM (406 п.о.), vWF (541 п.о.), ACP5, (472 п.о.), APOB (992 п.о.), C-MYC (670 п.о.), LCAT (485 п.о.), GHR (903 п.о.), CNR1 (998 п.о.), MGF (515 п.о.), THY (603 п.о.), SPTBN (801 п.о.), BRCA1 (889 п.о.), BRCA2 (962 п.о.), IRBP (1153 п.о.), ABHD11 (554 п.о.), AAT (515 п.о.), CHY (597 п.о.), DMP1 (587 п.о.), G6PD (601 п.о.), NHSL1 (596 п.о.), ostA (537 п.о.), PRF (587 п.о.), ROGDI (416 п.о.), TNF (541 п.о.) [7].
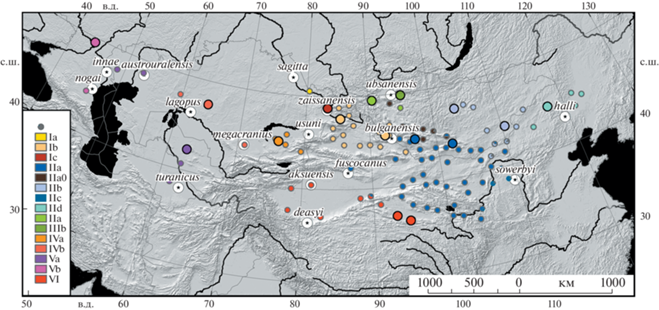
Рис. 1.
Точки сбора материала для исследования представителей видового комплекса Dipus sagitta. Цветовая легенда соответствует митохондриальным гаплогруппам [3]. Крупные цветные точки обозначают места происхождения образцов для исследования ядерных генов в данной работе. Белые точки со звездами обозначают типовые локалитеты подвидов D. sagitta sensu lato.
Всего было получено 459 последовательностей для указанных образцов и маркеров (номера в базе данных GenBank: OP800526-OP800657; OP819961-OP820046; OP823299-OP823388; OP866973-OP866990; OP970466-OP970500; MF535892; MF535896; MF535877; MF535876; MF535880; MF535849; MF535850; MF535853; MF535863; MF535871; MF535835; MF535842; OP970450; MF535824; MF535819; OP970453; OP970454; MF535798; MF535804; MF535837; MF535800; MF535816; OP970460-OP970465), из них 19 были получены в ходе предыдущей работы [3]. На их основе было создано объединенное множественное выравнивание для дальнейшего анализа в программе MEGA v6.06 [8]. Филогенетический анализ выполнен методом максимального правдоподобия (maximum likelihood, ML) в программе IQ-TREE v. 1.6.12 [9, 10] с делением на партиции, соответствующие отдельным маркерам и применением fast bootstrap (10 000 реплик). Jaculus jaculus использован в качестве внешней группы. Филогенетическая сеть построена в программе SplitsTree v. 4 методом Split decomposition [11]. Хронограмма получена в программе BEAST v. 1.10.4 [12] с использованием в качестве вторичной калибровочной точки разделение Jaculus/Dipus (логнормальное распределение: mean = 1.517; stdev = 0.6124; offset = 8.2 [7]). Для разных генов использовались как строгие, так и свободные часы в зависимости от результатов тестов отношения правдоподобия (likelihood-ratio tests), проведенных с помощью PAUP v. 4b10 [13].
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
Наши результаты секвенирования ядерных генов (рис. 2а) подтверждают базальное положение D. deasyi относительно всех остальных генетических линий комплекса Dipus sagitta s.l., что полностью соответствует известным генетическим данным и подтверждает видовой статус этого таксона на новой расширенной выборке ядерных локусов [3, 4]. Образцы подвидов группы “lagopus” (D. s. lagopus, D. s. usuni, D. s. turanicus, D. s. nogai), образуют монофилетическую группу, в то время как группа “sagitta” (D. s. ubsanensis, D. s. halli, D. s. sowerbyi, D. s. bulganensis, D. s. zaissanensis) парафилетична относительно “lagopus”. На митохондриальном дереве (рис. 2б) выявленные филогруппы довольно хорошо соответствуют морфологическим подвидам, однако четкого подразделения их на группы “lagopus” и “sagitta” не наблюдается [3].
Рис. 2.
Филогения видового комплекса Dipus sagitta sensu lato, реконструированная на основе ядерных (а) и митохондриальных (б) данных
методами максимального правдоподобия. Числа вблизи узлов дерева на рисунке a показывают
поддержки, полученные методом fast bootstrap. Символы на рисунке B обозначают высокие
поддержки (звезда (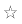 ) – 0.95–1.0 BPP/91-100 бутстрэп), средние поддержки (квадрат ($\square $) – 0.9–0.96 BPP/75-90 бутстрэп), либо отсутствие значимых поддержек (круг ($\bigcirc $) – <0.89 BPP/<74 бутстрэп) (где BPP – апостериорные вероятности из Байесова анализа
в MrBayes); реконструкция и поддержки приведены на основе [3, 4]. Цветовая легенда соответствует рис. 1. Внешние группы удалены.
) – 0.95–1.0 BPP/91-100 бутстрэп), средние поддержки (квадрат ($\square $) – 0.9–0.96 BPP/75-90 бутстрэп), либо отсутствие значимых поддержек (круг ($\bigcirc $) – <0.89 BPP/<74 бутстрэп) (где BPP – апостериорные вероятности из Байесова анализа
в MrBayes); реконструкция и поддержки приведены на основе [3, 4]. Цветовая легенда соответствует рис. 1. Внешние группы удалены.
D. s. ubsanensis, как по ядерной, так и по мтДНК, занимает обособленное положение, что не согласуется с выводами морфологической ревизии, согласно которой D. s. ubsanensis включен в состав подвида D. s. sowerbyi [1]. D. s. bulganensis монофилетичен только на митохондриальном дереве. Экземпляры, принадлежащие Dipus s. ssp. из восточной Гоби и центральной Монголии (M03-4, M14-99), которые на митохондриальном дереве объединяются с D. s. sowerbyi, на ядерном дереве тяготеют к D. s. ubsanensis.
Поддержка многих узлов на ядерном дереве низкая (рис. 2а), что, учитывая приведенные выше факты несоответствия топологии митохондриального и ядерного деревьев (mitonuclear discordance), говорит о вероятных ретикулярных процессах в эволюции группы. Митохондриальные и ядерные данные, полученные ранее, также указывали на наличие потенциальной гибридизации между D. s. ubsanensis и D. s. sowerbyi [3].
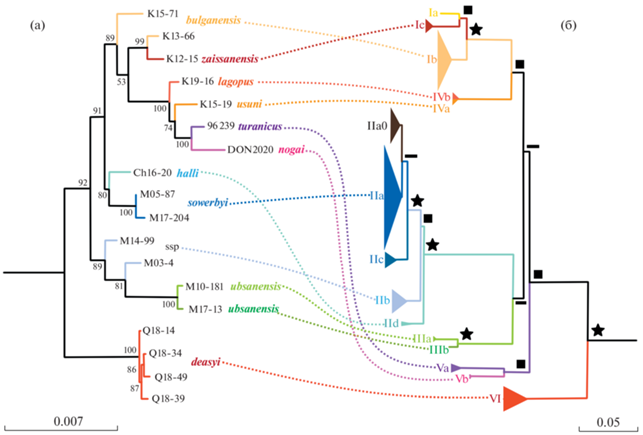
С предположением о гибридном происхождении некоторых линий согласуется и структура филогенетической сети (рис. 3), которая содержит несколько циклов. В частности, это относится к D. s. bulganensis (из Монгольской Джунгарии) и Dipus s. ssp из восточной Гоби и центральной Монголии. При исключении из анализа принадлежащих этим формам образцов (M03-4, M14-99 и M15-71), поддержки на дереве заметно возрастают (рис. 4).
Рис. 3.
Филогенетическая сеть представителей Dipus sagitta sensu lato, построенная методом Split decomposition на основе ядерных маркеров. Цветовая легенда соответствует рис. 1.
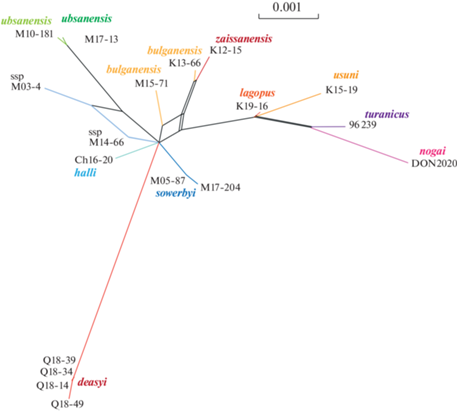
Рис. 4.
Калиброванное филогенетическое дерево видового комплекса Dipus sagitta sensu lato, реконструированное на основе ядерных данных в программе BEAST v1.10.4. Таксоны, потенциально имеющие гибридное происхождение, были исключены из анализа. Числа вблизи узлов дерева показывают апостериорные вероятности (BPP) программы BEAST и бутстрэп поддержки, полученные в программе IQ-TREE для сокращенной выборки. Цветовая легенда соответствует рис. 1. Таксоны внешней группы удалены.
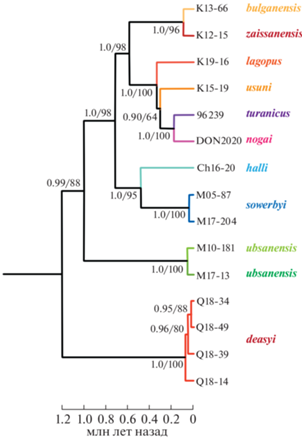
Времена дивергенции, полученные в нашем филогенетическом анализе объединенной последовательности из 26 ядерных генов, не сильно отличаются от наших прежних датировок на основе видового дерева по cytb и двум ядерным генам [3]. Отделение D. deasyi от остальных форм видового комплекса произошло 1.2 млн лет назад; обособление D. s. ubsanensis произошло около 1 млн лет назад; группы “lagopus” и “sowerbyi” разделились 720 тыс. лет назад.
ЗАКЛЮЧЕНИЕ
Наши результаты демонстрируют сложность интерпретации филогенетического сигнала, получаемого от ядерных, митохондриальных и морфологических данных. Расширение выборки маркеров до 26 локусов позволило получить филогенетическое дерево D. sagitta s.l. с удовлетворительными поддержками значительной части узлов. Невзирая на расхождения между разными генетическими маркерами, подтверждается высокий уровень разнообразия этого видового комплекса и аутентичность большинства генетических линий, выявленных ранее по мтДНК [3]. Поддерживаются базальное положение и несомненный видовой статус D. deasyi, монофилия группы “lagopus” и раннее обособление D. s. ubsanensis. Недостаток разрешения филогенетических реконструкций объясняется, вероятно, ретикулярными процессами в эволюции группы. Несмотря на то что дивергенция между линиями достигает видового уровня, при существенном их возрасте (около 1 млн лет) обмен генами между ними сохраняется, а часть из линий имеет, возможно, гибридогенное происхождение.
Список литературы
Шенброт Г.И. Географическая изменчивость мохноногого тушканчика Dipus Sagitta (Rodenita, Didodidae). Подвидовая дифференциация на территории Восточного Казахстана, Тувы и Монголии // Зоологический журнал. 1991. V. 70. № 7. P. 91–97.
Шенброт Г.И. Географическая изменчивость мохноногого тушканчика Dipus Sagitta (Rodenita, Didodidae). Общий характер внутривидовой изменчивости и подвидовая дифференциация в западной части видового ареала // Зоологический журнал. 1991. V. 70. № 5. P. 101–110.
Lebedev V.S., Bannikova A.A., Lu L., et al. Phylogeographical study reveals high genetic diversity in a widespread desert rodent, Dipus sagitta (Dipodidae: Rodentia) // Biological Journal of Linnean Society. 2018. V. 123. № 2. P. 445–462.
Cheng J., Ge D., Xia L., et al. Phylogeny and taxonomic reassessment of jerboa, Dipus (Rodentia, Dipodinae), in inland Asia // Zoologica Scripta. 2018. V. 47. № 6. P. 630–644.
Cheng J., Lv, X., Xia L., et al. Impact of orogeny and environmental change on genetic divergence and demographic history of Dipus sagitta (Dipodoidea, Dipodinae) since the Pliocene in Inland East Asia // Journal of Mammalian Evolution. 2019. V. 26. № 2. P. 253–266.
Sambrook J., Fritsch E.F., Maniatis T. Molecular cloning – a laboratory manual. Sec. ed. N.Y.k: Cold Spring Harbor Lab. Press, 1989. 385 p.
Lebedev V.S., Shenbrot G.I., Krystufek B., et al. Phylogenetic relations and range history of jerboas of the Allactaginae subfamily (Dipodidae, Rodentia) // Scientific Reports. 2022. V. 12. № 1. P. 842.
Tamura K., Stecher G., Peterson D., et al. MEGA6: Molecular Evolutionary Genetics Analysis Version 6.0 // Molecular Biology and Evolution. 2013. V. 30. № 12. P. 2725–2729.
Nguyen L.-T., Schmidt H.A., Haeseler von A., et al. IQ-TREE: a fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies // Molecular Biology and Evolution. 2015. V. 32. № 1. P. 268–274.
Chernomor O., von Haeseler A., Minh B.Q. Terrace aware data structure for phylogenomic inference from supermatrices // Systematic Biology. 2016. V. 65. № 6. P. 997–1008.
Huson D.H., Bryant D. Application of phylogenetic networks in evolutionary studies // Molecular Biology and Evolution. 2006. V. 23. № 2. P. 254–267.
Suchard M.A., Lemey P., Baele G., et al. Bayesian phylogenetic and phylodynamic data integration using BEAST 1.10 // Virus Evolution. 2018. V. 4. № 1. vey016.
Swofford D.L. PAUP*: Phylogenetic analysis using parsimony (* and other methods), version 40b10: 40b10. Sunderland: Sinauer, 2003.
Дополнительные материалы отсутствуют.
Инструменты
Доклады Российской академии наук. Науки о жизни


