Почвоведение, 2023, № 7, стр. 853-863
Изменение структуры прокариотного сообщества нефтезагрязненного чернозема при внесении нитрата и хлорида калия
А. П. Власова a, *, К. В. Павлов a, Е. В. Морачевская a, Д. Н. Липатов a, Л. А. Поздняков a, Н. А. Манучарова a, **
a МГУ им. М.В. Ломоносова
119991 Москва, Ленинские горы, 1, Россия
* E-mail: anastasya.nast-vlasova@yandex.ru
** E-mail: manucharova@mail.ru
Поступила в редакцию 29.09.2022
После доработки 12.03.2023
Принята к публикации 12.03.2023
- EDN: VOFTDS
- DOI: 10.31857/S0032180X22601165
Аннотация
В лабораторном эксперименте изучено влияние солей нитрата и хлорида калия на структуру метаболически активного прокариотного сообщества чернозема, загрязненного нефтью. Объектами исследования служили образцы чернозема, отобранные в Воронежской области. Рассматривали филогенетическое и функциональное разнообразие прокариотного комплекса чернозема, загрязненного нефтью, при внесении нитрата и хлорида калия в условиях слабощелочной реакции среды. Загрязнение чернозема нефтью в количестве 5% от массы почвы приводило к подщелачиванию среды от 7.1 до 7.9. Внесение нитрата и хлорида калия, как раздельно, так и совместно в суммарной дозе 2 ммоль/100 г почвы снимало этот негативный эффект. Совместное внесение нитрата и хлорида калия приводило к двукратному увеличению биомассы метаболически активных клеток прокариот и числа копий функциональных генов, отвечающих за синтез ферментов алканмонооксигеназ, участвующих в разложении нефти. В присутствии нефти выявлено формирование специфического комплекса бактерий, в котором преобладали представители Actinobacteria (Rhodococcus erythropolis) и Alphap-roteobacteria (Bradyrhizobium japonicum). Rhodococcus erythropolis и Bradyrhizobium japonicum, являясь автохтонными организмами в незагрязненной почве, начали занимать доминирующие позиции в нефтезагрязненных образцах, а внесение нитратов усилило этот эффект.
ВВЕДЕНИЕ
Стимулирование биологических процессов деградации нефти в почве – это наиболее экологичный и простой прием ремедиации нефтезагрязненных почв, в рамках которого применяются минеральные удобрения [12].
Внесение удобрений с целью очистки почв от нефти и нефтепродуктов позволяет стимулировать работу местного микробного комплекса разных типов почв, не прибегая к использованию недешевых препаратов микробных деструкторов нефти. Повышенная концентрация легкорастворимых солей, так же как щелочная реакция среды, являются наиболее распространенными сопутствующими факторами при нефтезагрязнении. Предполагается, что эффект от воздействия хлоридов и нитратов на микробное сообщество изменяется в зависимости от реакции среды. Изучение химических и агрохимических условий деятельности микроорганизмов в нарушенных агроценозах и природных системах в дальнейшем позволит усилить биотехнологические свойства микробного сообщества почвы.
В рамках работы оценивали разнообразие, численность и биомассу метаболически активного прокариотного сообщества исследуемого чернозема; наличие функциональных генов аlkB и alkM, синтез которых индуцируется алканами, а также численность метаболически активных клеток прокариот, обладающих этими генами.
Цель работы – изучение структуры прокариотного комплекса чернозема, загрязненного нефтью, при внесении нитрата и хлорида калия в условиях слабощелочной реакции среды.
ОБЪЕКТЫ И МЕТОДЫ
Объектами исследования служили образцы горизонта (А) чернозема типичного (табл. 1) среднемощного среднесуглинистого на лёссовидных суглинках (51°1′41″ N, 40°43′31″ E) [2]. Модельный лабораторный эксперимент по внесению нефти и минеральных солей проводили в сосудах, вмещающих 100 г почвы (объем 500 мл3, 82, 108, 70 мм). Почвенные образцы увлажняли водой, доводя давление почвенной влаги до –3.4 кПа (оптимальные значения влажности для развития большинства микроорганизмов), и поддерживали ее в течение всего эксперимента. Нефть добавляли в количестве, превышающем показатели сильнозагрязненных почв (5% от массы почвы). KNO3 и KCl вносили в почву равномерно в форме растворов, концентрации анионов выравнивали в мольном отношении 2 ммоля анионов ${\text{NO}}_{3}^{ - }$ и Cl– на 100 г почвы (табл. 2).
Таблица 1.
Агрохимическая характеристика образца гумусового горизонта чернозема типичного (Haplic Chernozem (Loamic, Pachic)), использованного в модельном эксперименте
| Показатель | Значение |
|---|---|
| Место отбора образцов | Каменная степь |
| Глубина отбора проб, см | 2–10 |
| Гумус, % | 8.1 |
| рH H2O | 7.1 |
| Фосфор подвижный по Чирикову, мг P2O5/100 г | 9.7 |
| Азот минеральный, мг N/100 г | 0.5 |
| Калий подвижный по Чирикову мг K2O/100 г | 26.8 |
Таблица 2.
Концентрация внесенных солей по вариантам опыта
| Вариант опыта | KNO3 | КСl | ||
|---|---|---|---|---|
| мг/100 г | ммоль/100 г | мг/100 г | ммоль/100 г | |
| Контроль | 0 | 0 | 0 | 0 |
| Н (нефть) | 0 | 0 | 0 | 0 |
| H + NO3 | 202 | 2 | 0 | 0 |
| H + Cl | 0 | 0 | 149 | 2 |
| H + NO3 + Cl | 202 | 2 | 149 | 2 |
Эксперимент проводили в течение 9 мес. В почву вносили нефть Азово-Кубанского нефтегазоносного бассейна (месторождение “Кубанская площадь”, скважина С-1, глубина отбора 2800–2832 м, возраст ${\text{N}}_{1}^{2},$ плотность нефти 0.835 г/см3 при 15°С, цетановое число 51, иодное число 0.4, вязкость условная 1.5 и кинематическая 4.9 при 20°С, мм2/с).
Измерение массовой доли нефтепродуктов проводили методом ИК-спектрометрии. Из каждого варианта опыта отбирали почвенные пробы массой 5 г, высушивали при комнатной температуре до воздушно-сухого состояния. Содержание нефтепродуктов в высушенных пробах исследовали методом ИК-спектрометрии в соответствии с ПНД Ф 16.1:2.2.22-98.
Количественный учет клеток прокариот проводили с помощью люминесцентной микроскопии. Люминесцентно-микроскопическим методом с использованием различных флуорохромов: акридина оранжевого, Су3, определяли общую численность и численность метаболически активных клеток в исследуемых образцах [13]. Общую численность прокариот определяли с помощью красителя акридина оранжевого, вступающего в реакцию с ДНК клеток. Численность метаболически активных клеток устанавливали при окрашивании рРНК клеток специфическими флуоресцентно-мечеными олигонуклеотидными зондами, что позволяет сделать выводы не только о жизнеспособности микроорганизмов, но и исследовать микробное разнообразие in situ. В основе FISH-метода лежит реакция гибридизации между искусственно созданным флуоресцентным РНК-зондом и комплементарной ему нуклеотидной последовательностью участка 16S рибосомальной PНК, что позволяет учесть количество только метаболически активных представителей различных филумов. Гибридизацию исследуемых образцов проводили с помощью зондов, специфичных для некоторых филумов представителей доменов Archaea и Bacteria. Гибридизацию препаратов с флуоресцентно-мечеными зондами проводили в соответствии с методикой [4] при температуре 46°С. Условия гибридизации, использованные для различных зондов, концентрация формамида в гибридизационном буфере и концентрация NaCl в буфере для промывки приведены в табл. 3.
Таблица 3.
Примененные рРНК-специфичные олигонуклеотидные зонды
| Зонд | Целевая группа организмов | Целевой участок 16S pPHK | Нуклеотидная последовательность зонда (5'–3') | Формамид, % | NaCl, мМ | Ссылка |
|---|---|---|---|---|---|---|
| ЕUВ338 | Bacteria (+ Planctomycetales и Verrucomicrobiales) | 338–355 | GCT GCC ТСС CGT AGG AGT GC(W) GCC (W)CC CGT AGG (W)GTа |
20 | 225 | [4, 5] |
| ARCH915 | Archaea | 915–934, | GTG СТС ССС CGC САА ТТС СТ | 30 | 112 | [21] |
| ARC344 | 344–363 | TCGCGCCTGCTGCIC ССС GT | ||||
| ALF1b ALF968 | Alphaproteobacteria | 19–35 | CGTTCGYTCTGAGCCAGа | 20 | 225 | [6, 15] |
| ВЕТ42а | Betaproteobacteria | 1027–1043г | GCC ТТС CCA CTT CGT TT | 35 | 80 | [15] |
| GAM42a | Gammaproteobacteria | 1027–1043г | GCC TTC CCA CAT CGT TT | 35 | 80 | [15] |
| SRB385Db | Deltaproteobacteria | 385–402 | CGG CGT TGC TGC GTC AGG | 20 | 225 | [19] |
| CF319a | Cytophaga-Flavobacterium | 319–336 | TGG TCC GTG TCT CAG TAC | 35 | 80 | [14] |
| CFB560 | Bacteroidetes | 560–575 | WCC CTT TAA ACC CAR Tа | 30 | 112 | [22] |
| HGC69a | Actinobacteria | 1901–1918б | TAT AGT TAC CAC CGC CGTв | 25 | 159 | [20] |
| LGC354A, LGC354B, LGC354C | Firmicutes | 354–371 | TGG GAA GAT ТСССТА CTG С, CGG GAA GAT ТСССТА CTG С, CCG GAA GAT ТСССТА CTG С |
35 | 80 | [16] |
| НоАс1402 | Acidobacteria | 1402–1420 | CTT TCG TGA TGT GAC GGGг | 10 | 450 | [9] |
| PLA886 | Planctomycetes | 886–904 | GCC TTG CGA CCA TAC TCC C | 30 | 112 | [18 ] |
| VERR139 | Verrucomicrobia | 138–155 | CGA GCT ATT CCC CTC TTG | 10 | 450 | [7] |
Аналогичный метод гибридизации in situ с рРНК-специфичными флуоресцентно-мечеными олигонуклеотидными зондами (FISH – fluorescent in situ hybridization) использовали для определения экспрессии функциональных генов алкан-монооксигеназ (alkB и alkM), отвечающих за деструкцию н-алканов в исследуемых образцах [10, 23, 25]. Праймер разрабатывали в соответствии с последовательностью генов alkB Pesudomonas AJ233397. Целевая область усиления составляет 870 базовых пар нуклеотидов. Гибридизацию препаратов с флуоресцентно-мечеными зондами проводили при температуре 45°С и концентрацией формамида 50% [26]. Условия гибридизации, использованные для зондов alkB и alkM, концентрация формамида в гибридизационном буфере и концентрация NaCl в буфере для промывки приведены в табл. 4–6.
Таблица 4.
Примененные в настоящем исследовании рРНК-специфичные олигонуклеотидные зонды для определения экспрессии функционального гена алкан-монооксигеназы (alkB и alkM) (метод FISH)
| Зонд | Нуклеотидная последовательность зонда (5'–3') | Формамид, %* | NaCl, мМ** |
|---|---|---|---|
| alkB(F) alkB(R) |
Cy3-tgg-ccg-gct-act-ccg-atg-atc-gga-atc-tgg Cy3-cgc-gtg-gtg-atc-cga-gtg-ccg-ctg-aag-gtg |
50 | 500 |
| alkM(F) alkM(R) |
Cy3-cc-tgt-ctc-att-tgg-cgc-tcg-ttc-cta-cag-g Cy3-gt-gat-gat-ctg-aat-gtc-gtt-gta-act-gg |
50 | 500 |
Таблица 5.
Праймеры, использованные в работе qPCR
| Целевая группа организмов | Праймеры | Нуклеотидная последовательность зонда (5'–3') | Ссылка |
|---|---|---|---|
| Bacteria (BAC) | 11F 11-21 1492R 1492-1513 |
AGAGTTTGATCMTGGCTCAG TACGGYTACCTTGTTACGACTT |
[17] |
| AlkB | 480F 480-502 954R 954-971 |
GGTACGGSCAYTTCTACRTCGA CGGRTTCGCGTGRTGRT |
[3] |
| AlkM | CCTGTCTCATTTGGCGCTCGTTCCTACAGG GTGATGATCTGAATGTCGTTGTAACTGG |
[24] |
Таблица 6.
Протоколы программ амплификации, используемые при работе real-time PCR
| Bac 16S | AlkB | AlkM |
|---|---|---|
| 1. 95°C – 03:00 | 1. 95°C – 05:00 | 1. 95°C – 05:00 |
| 2. 95°C – 00:10 | 2. 94°C – 01:00 | 2. 94°C – 01:00 |
| 50°C – 00:10 | 60°C – 01:00 | 60°C – 01:00 |
| 72°C – 00:20 | 72°C – 01:00 | 72°C – 01:00 |
| 100 циклов | 30 циклов | 30 циклов |
| 3. 90°C – 25:00 | 3. 72°C – 03:00 | 3. 72°C – 03:00 |
Для прямого учета микроорганизмов использовали микроскоп Axioskop 2 plus, ZEIZZ, Германия. Численность микробных клеток, содержащихся в 1 г образца, вычисляли по формуле:
Метод ПЦР в реальном времени (real-time PCR, qPCR) применяли для анализа числа копий ДНК представителей бактерий и обнаружения наличия генов alkB, кодирующих синтез алканмонооксигеназы. Метод основан на обнаружении в реальном времени меченых молекул ДНК, флуоресценция которых увеличивается по мере накопления продукта ПЦР в течение каждого цикла амплификации. Количественный анализ числа копий ДНК прокариот в исследуемых вариантах проводили путем ПЦР в реальном времени на определенные сутки сукцессии (6, 15, 21 сут) на детектирующем амплификаторе DТLite4 ДНК-Технология. Для проведения полимеразной цепной реакции в реальном времени предварительно выделяли тотальное ДНК из исследуемых образцов на разных этапах сукцессии. Для этого использовали набор Power Soil DNA Kit. Последовательности праймеров, используемых в работе для выявления представителей домена Bacteria и обнаружения наличия генов, кодирующих алканмонооксигеназы, а также протоколы программ амплификации, представлены в табл. 5. Результаты измерений обрабатывали с использованием пакета программы Realtime PCR. Данный детектирующий амплификатор DТLite4 совмещает в себе функции программируемого термоциклера и оптической системы, позволяющей регистрировать флуоресценцию реакционной смеси в пробирках в ходе полимеразной цепной реакции. Калибровку прибора проводили по зависимости интенсивности флуоресценции от логарифма концентрации ДНК стандартных растворов.
Для анализа разнообразия прокариотного комплекса исследуемых образцов почв применяли метод высокопроизводительного секвенирования консервативного участка гена 16S рРНК. Для экстракции тотальной ДНК применяли стандартные методы Power Soil DNA Isolation Kit (MO BIO, США), руководствуясь инструкциями производителя. Амплификацию фрагментов гена 16S рРНК осуществляли с помощью вырожденных праймеров, комплементарных последовательностям как бактерий, так и архей: PRK341F (CCTACGGGRBGCASCAG) и PRK806R (GGACTACYVGGGTATCTAAT). Полученные ПЦР-фрагменты очищали на колонках QIAquick согласно протоколу производителя. Каждый ПЦР-фрагмент растворяли в 50 мкл ТЕ-буфера, полученного материала было достаточно для дальнейшего анализа. Нуклеотидные последовательности вариабельных фрагментов генов 16S рибосомальных РНК из образцов метагеномной ДНК определяли с помощью высокопроизводительного секвенирования. Секвенирование проводили на полигеномном секвенаторе Illumina Miseq, время прочтения 39 ч, количество pair-endreads (парных прочтений) 8 млн. После проведенных прочтений с обоих концов ДНК формировали файл с прямыми и обратными прочтениями, которые представляли собой текстовое описание первичной структуры линейных макромолекул в виде последовательности мономеров. Обработку данных секвенирования проводили с использованием автоматизированного алгоритма QIIME 1.9.1, включающего объединение прямых и обратных прочтений. Удаление технических последовательностей, фильтрацию последовательностей с низкими показателями достоверности прочтения отдельных нуклеотидов (качество <Q20), фильтрацию химерных последовательностей, выравнивание прочтений на референсную последовательность 16S рРНК, распределение последовательностей по таксономическим единицам проводили с использованием базы данных Silva версии 132. Применяли алгоритм классификации операционных таксoномических единиц (ОТЕ) с открытым референсом (Open-reference OTU), порог классификации 97% [13].
Статистическую обработку данных проводили с помощью программы Statistica 6.0. Все пробы почв анализировали в 5-кратной повторности (каждый вариант включал пять повторений).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Загрязнение почвенных образцов нефтью в концентрации, соответствующей 5% от массы почвы и относящейся к сильному загрязнению, способствует снижению как общей микробной биомассы, так и биомассы метаболически активных прокариот, в среднем в 2 раза по сравнению с незагрязненной почвой. Филогенетическая структура метаболически активных представителей домена Bacteria в черноземе значительно изменилась в присутствии нефти. Повысилась доля актинобактерий, минорные филумы сокращали численность или выпадали из метаболически активного комплекса. При этом протеобактерии сохраняли свое место среди доминантов. Отмеченные закономерности наблюдались ранее [12, 13].
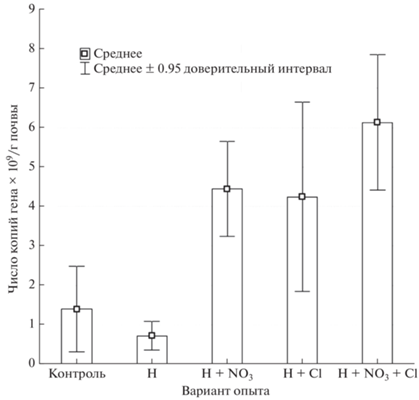
Наибольшая численность бактерий (по количеству копий гена 16S рРНК) в нефтезагрязненных образцах наблюдалась в вариантах с совместным внесением нитратов и хлоридов калия (рис. 1). Это может быть обусловлено тем, что нитраты восполняют недостаток азота, а хлориды являются физиологическим подкислителем, не давая проявляться подщелачиванию, которое возникает при попадании нефти со щелочными примесями. Оценка величины кислотности показала, что в почвенных образцах с внесенной нефтью значение pH повышалось по сравнению с контролем до 7.9, тогда как в образцах, подвергшихся ремедиации, приближалось к 7.1 и соответствовало значениям контрольных незагрязненных вариантов.
Рис. 1.
Число копий гена 16S РНК в образцах опыта: контроль, Н – чернозем с внесением нефти, Н + NO3 – чернозем с внесением нефти и KNO3, H + Cl – чернозем с внесением нефти и KCl, H + NO3 + Cl – чернозем с внесением нефти, KNO3 и КСl, n = 5.
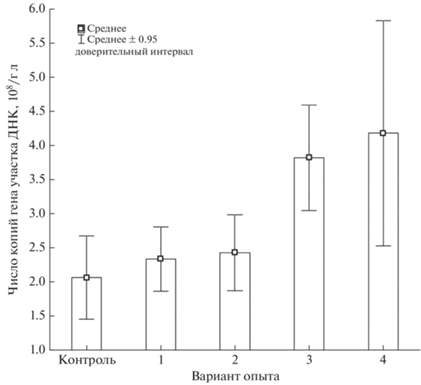
Отмеченная закономерность проявляется и при анализе численности копий функционального гена аlkB – кодирующего синтез фермента алканмонооксигеназы. Наибольшее количество копий этого гена обнаружено в вариантах при совместном внесении хлоридов и нитратов калия (рис. 2), что указывает на повышенную скорость разложения углеводородов нефти среди исследуемых вариантов опыта.
Рис. 2.
Число копий участка функционального гена аlkB в образцах опыта: контроль, 1 – чернозем с внесением нефти, 2 – чернозем с внесением нефти и KNO3, 3 – чернозем с внесением нефти и KCl, 4 – чернозем с внесением нефти, KNO3 и КСl.
Присутствие генов аlkB и аlkM свидетельствует об участии микробного сообщества в разложении алифатических углеводородов нефти (рис. 3). Что подтверждается определением остаточной концентрации нефти в почве. Так, содержание нефтепродуктов в первый месяц опыта в образцах чернозема было максимальным и достигало 11.7 г/кг. При добавлении хлорида калия и нитрата калия отмечалось снижение содержания нефтепродуктов в почве. Наименьшая концентрация нефти составляла 10.4 г/кг.
Рис. 3.
Биомасса метаболически активных прокариот, имеющих ген аlkB в исследуемых образцах чернозема: контроль, 1 – чернозем с внесением нефти, 2 – чернозем с внесением нефти и KNO3, 3 – чернозем с внесением нефти и KCl, 4 – чернозем с внесением нефти, KNO3 и КСl, n = 5.
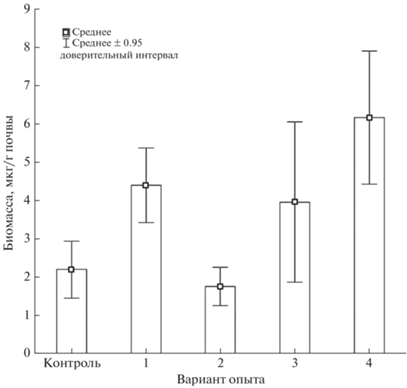
Таким образом, совместное внесение нитрата и хлорида калия в загрязненный нефтью чернозем в концентрации 2 ммоля/100 г приводит к увеличению в 2 раза биомассы метаболически активных клеток прокариот и числа копий функциональных генов, обеспечивающих разложение нефти.
Метагеномный анализ структуры микробных сообществ нефтезагрязненной почвы показал (рис. 4), что в качестве доминирующих групп прокариотного бактериального сообщества во всех рассматриваемых вариантах (контрольных, загрязненных и подверженных агроремедиации) выделялись Actinobacteria, Proteobacteria и Acidobacteria. Различия в структуре прокариотных сообществ между вариантами выявлены только на уровне родов. В нефтезагрязненном варианте в присутствии нитрата калия среди филума Actinobacteria доминировали представители Rhodococcus erythropolis groop, составляя 37% от актинобактерий и 18% от всех выявляемых бактерий (рис. 5). Согласно данным некоторых авторов [11], Rhodococcus erythropolis PR4 способен разлагать дизельное топливо, нормальные, изо- и циклопарафины, а также ароматические соединения.
Рис. 4.
Филогенетическая структура бактериального комплекса исследуемых образцов чернозема: контроль, Н – чернозем с внесением нефти, Н + NO3 – чернозем с внесением нефти и KNO3, H + Cl – чернозем с внесением нефти и KCl, H + NO3 + Cl – чернозем с внесением нефти, KNO3 и КСl.
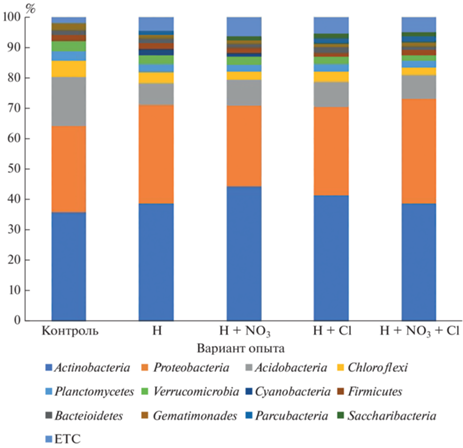
Кроме того, в вариантах без внесения KNO3 доля представителей Gammаproteobacteria составляла 5–10% от всех выявляемых протеобактерий, в то время как в вариантах с азотом их доля удваивается и достигает 19–22%. Среди протеобактерий доминирующими оказались представители Bradyrhizobium japonicum Group (H + N 22%). По литературным данным, Bradyrhizobium japonicum – вид клубеньковых бактерий, способных разлагать катехин, который содержит фенольные кольца [1, 8 ].
Таким образом, результаты метагеномных исследований образцов чернозема показали, что Rhodococcus erythropolis и Bradyrhizobium japonicum, являясь минорным компонентом в контрольной почве, начинают занимать доминирующие позиции в структуре микробного комплекса в условиях загрязнения нефтью, а внесение нитрата калия усиливает этот эффект.
ЗАКЛЮЧЕНИЕ
В настоящее время существуют различные подходы к рекультивации нефтезагрязненных почв в зависимости от типа и степени загрязнения, учитывая биоклиматические, геоморфологические факторы, а также почвенно-растительные условия. Например, для биологической рекультивации земель могут применяться посевы однолетних трав – мелиорантов, обладающих развитой корневой системой и повышенной устойчивостью к загрязнению почвы нефтепродуктами, с последующим внесением удобрений. Данная технология направлена на активацию имеющейся в почве (или привнесенной) биомассы нефтеокисляющих микроорганизмов.
Результаты исследований на примере чернозема показали, что сильное загрязнение (5% от массы почвы) приводило к подщелачиванию среды с 7.1 до 7.9. Внесение нитрата и хлорида калия в дозе 2 ммоль/100 г почвы снимало этот негативный эффект и поддерживало реакцию среды нефтезагрязненной почвы на постоянном уровне.
Совместное внесение в загрязненный нефтью чернозем нитрата и хлорида калия приводило к увеличению в 2 раза биомассы метаболически активных клеток прокариот и копий функциональных генов, отвечающих за синтез ферментов (алканмонооксигеназ), участвующих в разложении нефти.
В почвах, загрязненных нефтью, наблюдалось формирование специфического комплекса бактерий, в котором преобладали представители Actinobacteria (Rhodococcus erythropolis) и Alphaproteobacteria (Bradyrhizobium japonicum). Являясь автохтонными организмами в незагрязненном черноземе, Rhodococcus erythropolis и Bradyrhizobium japonicum начинают доминировать в структуре микробного комплекса в условиях нефтяного загрязнения, а внесение агроремедианта (нитрата калия) лишь усиливает этот эффект.
Список литературы
Добровольская Т.Г. Структура бактериальных сообществ почв. М.: Академкнига, 2002. 281 с.
Егоров В.В., Иванова Е.Н., Фридланд В.М. Классификация и диагностика почв СССР. М.: Колос, 1977. 221 с.
Коршунова А.В. Рибосомные и кодирующие белки гены (gyrB, alkB и parE) бактерий рода Geobacillus и использование их в таксономии и экологии. Дис. … канд. биол. наук. М., 2014. 124 с.
Amann R.I., Binder B.J., Olson R.J., Chisholm S.W., Devereux R., Stahl D.A. Combination of 16S rRNA-Targeted Oligonucleotide Probes with Flow Cytometry for Analyzing Mixed Microbial Populations // Appl. Environ. Microbiol. 1990. V. 56. P. 1919–1925. https://doi.org/10.1128/aem.56.6.1919-1925
Daims H., Bruhl A., Amann R., Schleifer K., Wagner M. The domain-specific probe EUB338 is insufficient for the detection of all Bacteria: development and evaluation of a more comprehensive probe set // Systematic Appl. Microbiol. 1999. V. 22. № 3. P. 434–444. https://doi.org/10.1016/S0723-2020(99)80053-8
Dedysh S.N., Panikov N.S., Tiedje J.M. Acidophilic Methanotrophic Communities from Sphagnum Peat Bogs // Appl. Environ. Microbiol. 1998. V. 64. № 3. P. 922–929. https://doi.org/10.1128/AEM.64.3.922-929.1998
Dedysh S.N., Pankratov T.A., Belova S.E., Kulichevskaya I.S., Liesack W. Phylogenetic Analysis and In Situ Identification of Bacteria Community Compositioninan Acidic Sphagnum Peat Bog // Appl. Environ. Microbiol. 2006. V. 72. № 3. P. 2110–2117. https://doi.org/10.1128/AEM.72.3.2110-2117.2006
Hopper W., Mahadevan A. Degradation of catechin by Bradyrhizobium japonicum // Biodegradation. 1997. № 8. P. 159–165. https://doi.org/10.1023/A:1008254812074
Juretschko S., Loy A., Lehner A., Wagner M. The microbial community composition of a nitrifying-denitrifying activated sludge from an industrial sewage treatment plant analyzed by the full-cycle rRNA approach // Systematic Appl. Microbiol. 2002. V. 25. № 1. P. 84–99. https://doi.org/10.1078/0723-2020-00093
Kok M., Oldenhuis R., vander Linded M.P.G., Meulenberg C.H.C., Kingma J., Witholt B. The Pseudomonas oleovorans alkBAC operon encodes two structurally related rubredoxins and an aldehyde dehydrogenase // J. Biol. Chem. 1989. V. 264. № 10. P. 5442–5451. https://doi.org/10.1016/S0021-9258(18)83565-7
Laczi K., Kis Á., Horváth B. et al. Metabolic responses of Rhodococcuserythropolis PR4 grown on diesel oil and various hydrocarbons // Appl. Microbiol Biotechnol. 2015. V. 99. № 22. P. 9745–9759. https://doi.org/10.1007/s00253-015-6936-z
Manucharova N.A., Ksenofontova N.A., Belov A.A., Kamenskiy N.N., Arzamazova A.V., Zenova G.M., Kinzhaev R.R., Trofimov S.Y., Stepanov A.L. Prokaryotic component of oil-contaminated oligotrophic peat soil under different levels of mineral nutrition: biomass, diversity, and activity // Eurasian Soil Science. 2021. V. 54. № 1. P. 89–97.https://doi.org/10.31857/s0032180x2101010x
Manucharova N.A., Ksenofontova N.A., Karimov T.D., Vlasova A.P., Zenova G.M., Stepanov A.L. Changes in the phylogenetic structure of the metabolically active prokaryotic soil complex induced by oil pollution // Microbiology. 2020. V. 89. № 2. P. 219–230. https://doi.org/10.31857/S0026365620020093
Manz W., Amann R., Ludwig W., Vancanneyt M., Schleifer K.H. Application of a suite of 16S rRNA-specific oligonucleotide probes designed to investigate bacteria of the phylum cytophaga-flavobacter-bacteroides in the natural environment // Microbiology. 1996. V. 142. P. 1097–1106. https://doi.org/10.1099/13500872-142-5-1097
Manz W., Amann R., Ludwig W., Wagner M., Schleifer K.H. Phylogenetic Oligodeoxynucleotide Probes for the Major Subclasses of Proteobacteria: Problems and Solutions // Systematic Appl. Microbiol. 1992. V. 15. I. 4. P. 593–600. https://doi.org/10.1016/S0723-2020(11)80121-9
Meier H., Amann R., Ludwig W., Schleifer K.H. Specific Oligonucleotide Probes for in situ Detection of a Major Group of Gram-positive Bacteria with low DNA G + C Content // Systematic Appl. Microbiol. 1999. V. 22. I. 2. P. 186–196. https://doi.org/10.1016/S0723-2020(99)80065-4
Monciardini P., Sosio M., Cavaletti L., Chiocchini C., Donadio S. New PCR primers for the selective amplification of 16S rDNA from different groups of actinomycetes // FEMS Microbiol. Ecol. 2002. V. 42. № 3. P. 419–429. https://doi.org/10.1111/j.1574-6941.2002.tb01031.x
Neef A., Amann R., Schlesner H., Schleifer K.H. Monitoring a widespread bacterial group: In situ detection of planctomycetes with 16S rRNA-targeted probes // Microbiology. 1998. V. 144. I. 12. P. 3257–3266. https://doi.org/10.1099/00221287-144-12-3257
Rabus R., Wilkes H., Schramm A. et al. Anaerobic utilization of alkylbenzenes and n-alkanes from crude oil in an enrichment culture of denitrifying bacteria affiliating with the beta-subclass of Proteobacteria // Environ. Microbiol. 1999. V. 1. № 2. P. 145–157. https://doi.org/10.1046/j.1462-2920.1999.00014.x
Roller C., Wagner M., Amann R., Ludwig W., Schleifer K.H. In situ probing of Gram-positive bacteria with high DNA G + C content using 23S rRNA-targeted oligonucleotides // Microbiology. 1994. V. 140. I. 10. P. 2849–2858. https://doi.org/10.1099/00221287-140-10-2849
Stahl D.A. Amann R. Development and application of nucleic acid probes. In Nucleic acid techniques in bacterial systematics / Eds. Stackebrandt E., Goodfellow M. Wiley, 1991. P. 205–248.
Weller R., Glöckner F.O., Amann R. 16S rRNA-Targeted Oligonucleotide Probes for the in situ Detection of Members of the Phylum Cytophaga-Flavobacterium-Bacteroides // Systematic Appl. Microbiol. 2000. V. 23. I. 1. P. 107–114. https://doi.org/10.1016/S0723-2020(00)80051-X
Whyte L.G., Schultz A., Beilen J.B. et al. Assessment of the bio degradation potential of psychrotrophic microorganisms // Can. J. Microbiol. 1996. V. 42. № 2. P. 99–106. https://doi.org/10.1139/m96-016
Whyte L.G., Schultz A., Beilen J.B. et al. Prevalence of alkane monooxygenase genes in Arctic and Antarctic hydrocarbon-contaminated and pristine soils // FEMS Microbiol. Ecol. 2002. V. 41. № 2. P. 41–50. https://doi.org/10.1111/j.1574-6941.2002.tb00975.x
Whyte L.G., Smits T.H., Labbé D., Witholt B., Greer C.W., van Beilen J.B. Gene cloning and characterization of multiple alkane hydroxylase systems in Rhodococcus strains Q15 and NRRL B-16531 // Appl. Environ. Microbiol. 2002. V. 68. № 12. P. 5933–5942. https://doi.org/10.1128/AEM.68.12.5933-5942.2002
Zhili He, Liyou Wu, Matthew W. Fields, Jizhong Zhou. Use of Microarrays with Different Probe Sizes for Monitoring Gene Expression // Appl. Environ. Microbiol. 2005. V. 71. № 9. P. 5154–5162. https://doi.org/10.1128/AEM.71.9.5154-5162.2005
Дополнительные материалы отсутствуют.



